
Un abstract grafico dell'asse intestino-cervello (a sinistra) e della piattaforma in vitro stampata in 3D (a destra). Credito:Università del Maryland
Chiunque abbia mai sperimentato "farfalle nello stomaco" prima di fare una grande presentazione non sarà sorpreso di apprendere che esiste una vera connessione fisica tra il loro intestino e il loro cervello. Neuroscienziati e professionisti medici chiamano questo "asse intestino-cervello" (GBA). Una migliore comprensione del GBA potrebbe portare a trattamenti e cure per disturbi neurologici dell'umore come depressione e ansia, così come per una serie di malattie infiammatorie autoimmuni croniche come la sindrome dell'intestino irritabile (IBS) e l'artrite reumatoide (RA).
Il problema è che fino ad ora le "farfalle" sono state tutto ciò con cui i medici hanno dovuto lavorare per rilevare questi disturbi legati al GBA. Ancora oggi queste malattie vengono principalmente diagnosticate dai resoconti dei sintomi da parte dei pazienti. Trovare "biomarcatori" - misurazioni oggettive di una sostanza nel corpo che indica la presenza di un disturbo - potrebbe migliorare notevolmente la diagnosi e togliere ai pazienti un enorme onere per identificare correttamente i propri sintomi.
Gli scienziati sospettano che il neurotrasmettitore chimico serotonina sia il biomarcatore per una serie di disturbi GBA. La serotonina stimola il sistema nervoso in azione attraverso il nervo vago, il connettore fisico tra il cervello e il colon. Generato in profondità all'interno del rivestimento dell'intestino, la serotonina alla fine influenza tutto, dall'umore e le emozioni al sonno, digestione e la secrezione di ormoni. La sua produzione è in qualche modo influenzata dal "microbioma" batterico presente in questo ambiente. I ricercatori sperano che la creazione di strumenti per analizzare la produzione e la disfunzione della serotonina nel microbioma intestinale aiuterà a svelare i misteri dei disturbi legati al GBA.
Con 1 milione di dollari in finanziamenti della National Science Foundation, Ingegneri dell'Università del Maryland (UMD), neuroscienziati, microbiologi e fisici hanno compiuto progressi significativi nello sviluppo di una piattaforma in grado di monitorare e modellare l'elaborazione in tempo reale dell'attività della serotonina del microbioma intestinale. Il loro obiettivo è un giorno impacchettare la piattaforma in una capsula ingeribile in grado di rilevare, trattamento e monitoraggio delle malattie da GBA.
La convergenza delle discipline è fondamentale, dice il professor Reza Ghodssi, ricercatore principale del progetto. "Stiamo convergendo neuroscienze, segnalazione molecolare, e dispositivi e sistemi micro-nano. Questo ci consente di misurare e investigare i dati all'interfaccia di ogni giunzione di una piattaforma GBA simulata, da cellula a cellula, cellula a molecola, molecola a nervo e sviluppare metodologie ingegneristiche per analizzarlo e interpretarlo."
Il lavoro si basa sull'esperienza dei dispositivi medici ingeribili che è stata sviluppata nel laboratorio di sensori e attuatori MEMS UMD, il Dipartimento di Bioingegneria Fischell, e l'iniziativa Brain and Behavior.
Tre nuovi articoli pubblicati descrivono in dettaglio i progressi nella rilevazione della serotonina, valutare i suoi effetti neurologici, e percependo piccoli cambiamenti nell'epitelio intestinale.
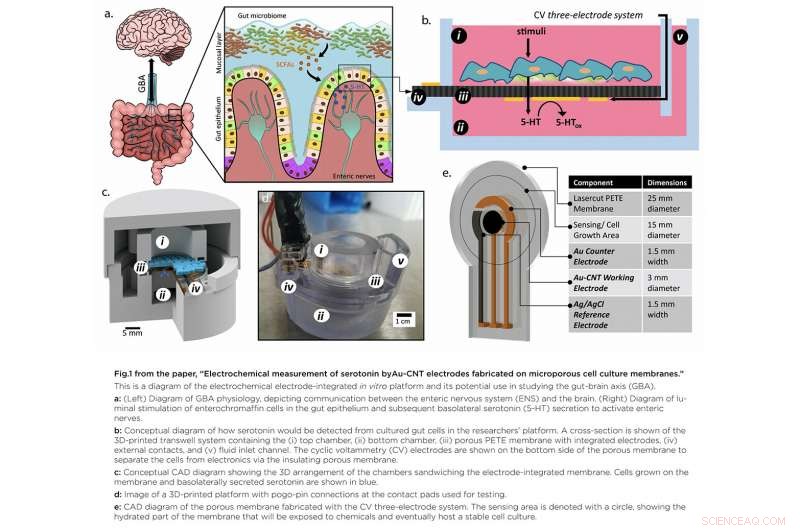
In "Misurazione elettrochimica della serotonina mediante elettrodi Au-CNT fabbricati su membrane di coltura cellulare porosa, " il team ha sviluppato una piattaforma che fornisce l'accesso al sito specifico di produzione di serotonina, importante perché la serotonina è secreta dal fondo delle cellule. Un'innovativa membrana porosa con un sensore di serotonina integrato su cui può essere coltivato un modello del rivestimento intestinale ha consentito ai ricercatori di accedere ai lati superiore e inferiore della coltura cellulare.
Il documento è stato pubblicato online il 7 settembre 2020 nel Natura rivista, Microsistemi e Nanoingegneria. Gli autori sono Bioingegneria Ph.D. la studentessa Ashley Chapin, ex ricercatore postdottorato ISR Pradeep Ramiah Rajasekaran, alunno David N. Quan (BioE Ph.D. 2015), Professore Liangbing Hu (MSE/MEII), Professore Associato Jens Herberholz (Psicologia/NACS), Professor William Bentley (BioE/Fischell Institute/IBBR), e il professor Reza Ghodssi (ECE/ISR).
Credito:Università del Maryland
Utilizzando la deposizione di metalli, hanno modellato un sistema a tre elettrodi direttamente su una membrana di coltura cellulare porosa sospesa all'interno di un alloggiamento personalizzato stampato in 3D. Le cellule possono essere coltivate sulla parte superiore della membrana con il sensore della serotonina orientato sul fondo per il rilevamento diretto. Il team ha quindi migliorato la sensibilità del rilevamento della serotonina aumentando la superficie effettiva dell'elettrodo, colata a goccia un piccolo volume di nanotubi di carbonio sulla superficie dell'elettrodo. Le soluzioni preparate di serotonina erano rilevabili ben all'interno dell'intervallo di concentrazione fisiologico previsto.
Il lavoro è il primo a dimostrare un metodo fattibile per rilevare molecole redox, come la serotonina, direttamente su un substrato di coltura cellulare poroso e flessibile. Garantisce un accesso superiore alle molecole rilasciate dalle cellule e crea un ambiente intestinale modello controllabile senza ricorrere a procedure invasive su esseri umani o animali.
Il secondo documento della squadra, "Un sistema di biomonitoraggio ibrido per la comunicazione tra intestino e neurone, " si basa sui risultati del primo:i ricercatori hanno sviluppato ulteriormente la piattaforma di misurazione della serotonina in modo che potesse valutare gli effetti neurologici della serotonina. Aggiungendo e integrando un modello di nervo di gambero sezionato con il modello di rivestimento intestinale, il team ha creato un'interfaccia intestino-neurone in grado di valutare elettrofisiologicamente la risposta nervosa alla serotonina rilevata elettrochimicamente. Questo progresso consente lo studio della segnalazione molecolare tra cellule intestinali e nervose, rendendo possibile per la prima volta il monitoraggio in tempo reale di entrambi i tessuti GBA.
Il documento è stato pubblicato online nel giugno 2020 IEEE Journal of Microelectromechanical Systems . È stato scritto da Chapin, Dottorato in Ingegneria Elettrica e Informatica studente Jinjing Han, Neuroscienze e Scienze Cognitive Ph.D. studente Ta-Wen Ho, Herberholz e Ghodssi.
Finalmente, il concetto, design and use for the entire biomonitoring platform is described in a third paper, "3-D Printed Electrochemical Sensor Integrated Transwell Systems, " published online Oct. 5, 2020 in the Natura rivista Microsystems and Nanoengineering . The paper was written by Rajasekaran, Chapin, Quan, Herberholz, Bentley and Ghodssi.
This paper delves into the development of the 3-D-printed housing, the maintenance of a healthy lab-on-a-chip gut cell culture, and the evaluation of the two types of sensors integrated on the cell culture membrane. The dual sensors are particularly important because they provide feedback about multiple components of the system—namely, the portions that model the gut lining's permeability (a strong indicator of disease) and its serotonin release (a measure of communication with the nervous system). Alongside the electrochemical sensor—evaluated using a standard redox molecule ferrocene dimethanol—an impedance sensor was used to monitor cell growth and coverage over the membrane. Using both these sensors would allow monitoring of a gut cell culture under various environmental and dietary conditions. It also would enable researchers to evaluate changes to barrier permeability (a strong indicator of disease), and serotonin release (a measure of communication with the nervous system).
"These works represent a big step forward in our understanding of the gut/brain axis, " says Cornell University's John March, Chair of the Department of Biological and Environmental Engineering. "One of the limitations of this field is the inability to perform highly controlled experiments in a 'close to in vivo' system. These papers provide ways around this problem with simple, elegant experiments that are highly accessible. I expect these will be used frequently."
Because the engineering aspects of the platform are well underway, the researchers are working towards culturing multi-tissue interfaces with the help of Jay Pasricha and Subhash Kulkarni at Johns Hopkins University. Eventually multiple platforms will be created, each colonized with a different combination of gut bacteria, to measure the neurophysiological effects of serotonin production in varying microbiome environments.
Con queste informazioni, Professor Wolfgang Losert (Physics/IPST/IREAP) will lead a machine learning effort to process the sensor data through a computer model that can simulate the outcomes of the different microbiomes. This will provide the clearest picture yet of how a system as complex and individually unique as the gut microbiome affects both gut and brain health. It also may help researchers better understand the connection between nutrition and mental health.
"Understanding biology at the level of whole organisms is a frontier in biology, and essential to forming a basis for precision medicine, " says the University of California, Berkeley's Amy Herr, the John D. &Catherine T. MacArthur Professor of Bioengineering. "By harnessing hallmarks of engineering—integrated, systems-level design—the new research from the Ghodssi-Bentley-Herberholz team presents an integrated approach to elegantly perturb and then probe the electrons and molecules that are key conduits of information flow in whole organisms."