Fig. 1. Da tempo gli scienziati tentano di decodificare le complesse sottostrutture delle proteine. Ora, i ricercatori della Tokyo Tech hanno finalmente fatto luce su questo fronte con lo studio dell'autoassemblaggio di proteine ingegnerizzate utilizzando aghi proteici. Credito:Takafumi Ueno del Tokyo Institute of Technology
L'assemblaggio delle proteine è essenziale per la formazione di strutture biologiche ordinate, ma immaginatene di ingegnerizzarne una. Questo è esattamente ciò che i ricercatori della Tokyo Tech hanno ottenuto con gli aghi proteici. Regolando le interazioni punta-punta di questi aghi, hanno consentito il loro autoassemblaggio in strutture reticolari, stati monomerici ordinati e assemblaggi di fibre, aprendo la strada alla costruzione controllata di più di tali architetture proteiche.
Le proteine sono i mattoni di base del nostro corpo. Tuttavia, le loro strutture molecolari e macroscopiche sono complesse e varie, con molteplici modelli e sottostrutture di piegatura. Gli scienziati stanno cercando da tempo di decodificare queste strutture e sono stati fatti molti progressi grazie alla microscopia a fluorescenza (FM), alla microscopia a forza atomica (AFM) e all'AFM ad alta velocità (HS-AFM). Tuttavia, non sono stati in grado di osservare direttamente i movimenti dinamici delle proteine durante l'assemblaggio. Ciò è dovuto principalmente alla struttura intricata delle proteine, che sono troppo piccole per essere misurate con le tecniche esistenti.
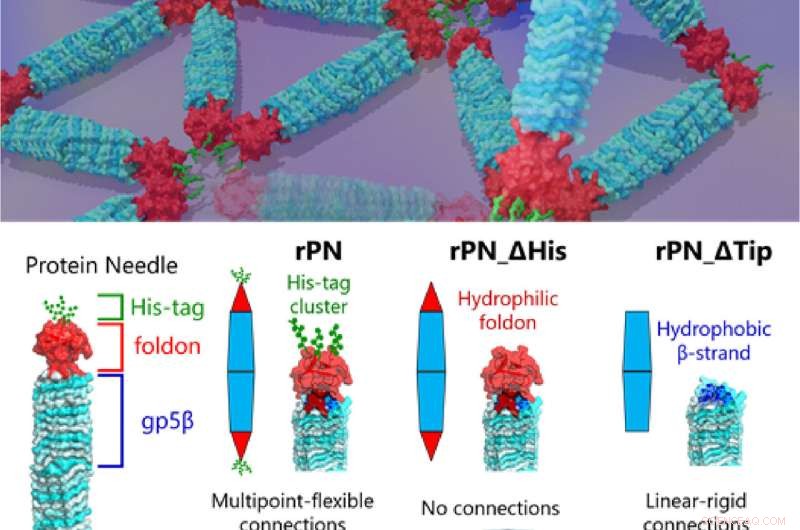
Un team di ricercatori in collaborazione del Tokyo Institute of Technology (Tokyo Tech), dell'Università di Kyushu, dell'Università di Nagoya e del National Institutes of Natural Sciences ha ora sviluppato un ago proteico anisotropo (PN) specializzato per aiutare a determinare l'assemblaggio di proteine similmente anisotrope, dandoci indizi sulla loro microstruttura e assemblaggio.
Il Prof. Takafumi Ueno della Tokyo Tech, che ha guidato lo studio, spiega la premessa del loro lavoro:"La nostra PN è una proteina a forma di ago composta dal corpo rigido (β-elica), dal cappuccio terminale (foldon) e da un legame motivo (tag esa-istidina, His-tag). Modificando questi PN eliminando il motivo His-tag e il foldon cap, possiamo produrre tre diversi tipi di PN. Questo ci ha permesso di regolare e osservare diversi schemi di assemblaggio e come cambiano , fornendoci indizi sulla meccanica delle diverse interazioni proteina-proteina che troviamo in natura". I risultati di questo studio sono stati pubblicati sulla rivista Small.
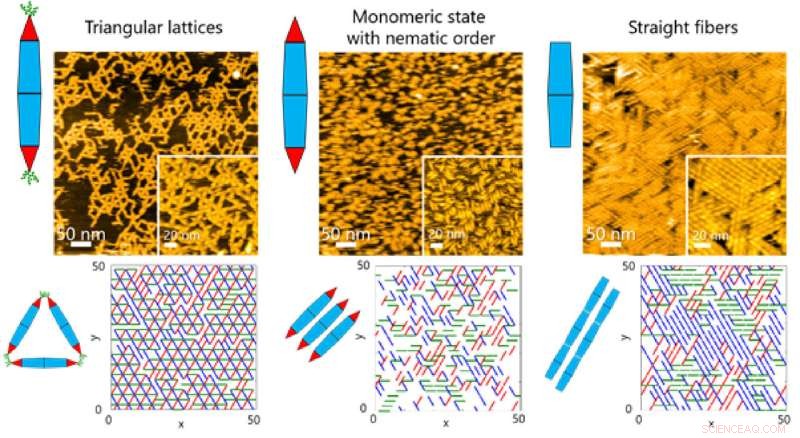
Fig. 2. Le catene proteiche di base spesso subiscono un autoassemblaggio dinamico per formare strutture supramolecolari complesse. Gli scienziati della Tokyo Tech sono ora riusciti a esplorare le dinamiche di assemblaggio utilizzando aghi proteici ingegnerizzati. Credito:Takafumi Ueno del Tokyo Institute of Technology
In soluzione, le PN formano spontaneamente una struttura altamente stabile con una lunghezza di circa 20 nm e una larghezza di circa 3,5 nm, abbastanza piccola da seguire il movimento rotatorio delle singole molecole ma meccanicamente forte.
Sulle superfici, il team ha osservato diversi tipi di strutture ordinate mentre le PN si autoassemblavano. Queste strutture variavano da reticoli triangolari e stati monomerici con ordine nematico (orientamento unidimensionale) ad assiemi di fibre (Figura 1).
Ciò, a sua volta, ha consentito al team di studiare i processi dinamici coinvolti nell'assemblaggio delle proteine attraverso una combinazione di HS-AFM e simulazioni (Figura 2). I risultati hanno rivelato che la formazione della struttura reticolare triangolare è stata guidata dai moti dinamici di PN, che contribuiscono a formare reticoli ordinati (Figura 3).
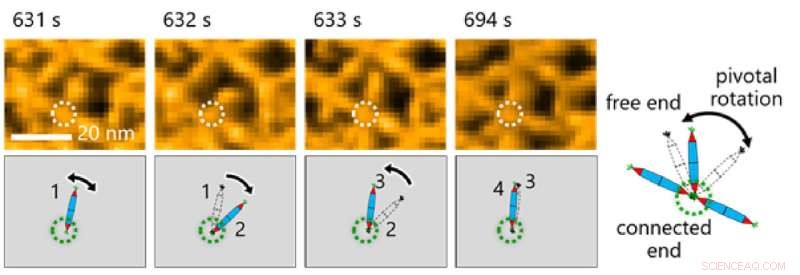
Fig. 3. L'osservazione del movimento molecolare delle PN è stata fondamentale per fare osservazioni in questo studio. Qui, i ricercatori hanno notato la rotazione fondamentale attorno all'interazione His-tag tra gli aghi proteici. Credito:Takafumi Ueno del Tokyo Institute of Technology
Questi risultati hanno entusiasmato i ricercatori, che stanno contemplando le sue potenziali ramificazioni. "Queste molecole svolgono un ruolo così cruciale nei sistemi biologici che la comprensione della loro struttura aumenterebbe notevolmente il campo. Ad esempio, potremmo usarle per gettare le basi per la costruzione di strutture supramolecolari progettando i movimenti collettivi dinamici delle proteine. Questo concetto può portare a l'ingegneria di materiali in fogli biocompatibili, trasporti mirati di farmaci e persino nano-robot a base di proteine", afferma il prof. Ueno. + Esplora ulteriormente