Credito:Università di Harvard
Man mano che gli scienziati hanno sondato i misteri della vita su scale sempre più piccole, hanno inventato strumenti per aiutarli a capire ciò che osservano. Determinare l'identità delle molecole di DNA e RNA è ormai diventato un luogo comune grazie allo sviluppo commerciale delle tecnologie di sequenziamento di prossima generazione, ma lo stesso non è ancora vero per le proteine, che sono attori di importanza critica in quasi tutti i processi biologici. Le proteine sono molto più complesse del DNA e dell'RNA e sono spesso modificate chimicamente, rendendo difficile da raggiungere l'obiettivo di identificare facilmente singole proteine all'interno di un campione (proteomica a singola molecola).
Ora, gli scienziati che lavorano alla Molecular Robotics Initiative all'interno del Wyss Institute dell'Università di Harvard, del Blavatnik Institute della Harvard Medical School (HMS) e del Boston Children's Hospital (BCH) hanno usato il DNA, la sostanza fondamentale della vita stessa, per creare ciò che può essere il righello più piccolo del mondo per misurare le proteine.
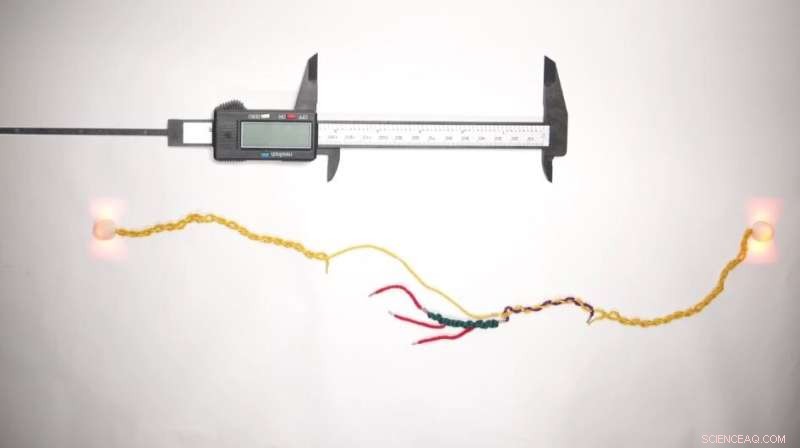
Soprannominata "DNA Nanoswitch Calipers" (DNC), questa tecnologia consente ai ricercatori di eseguire misurazioni della distanza su singoli peptidi (i mattoni delle proteine) con elevata precisione applicando piccole quantità di forza. Effettuando rapidamente molte misurazioni della distanza sulla stessa molecola, il DNC crea un'unica "impronta digitale" che può essere utilizzata per identificarla negli esperimenti successivi. Il risultato è riportato in Nature Nanotechnology .
"Quando stai cercando di capire qualcosa in biologia, ci sono due metodi principali di indagine:puoi osservare il tuo soggetto nel suo stato naturale, oppure puoi perturbarlo e vedere come reagisce. Le osservazioni possono fornire molte grandi informazioni biologiche, ma a volte il modo migliore per conoscere qualcosa è interagire fisicamente con esso", ha affermato il co-autore Wesley Wong, Ph.D., membro della facoltà associata al Wyss Institute e professore associato presso HMS che è anche ricercatore presso BCH . "La determinazione del modello degli amminoacidi all'interno di una molecola peptidica mediante l'applicazione della forza è un nuovo paradigma nella continua ricerca scientifica di tecniche che ci consentiranno di sequenziare le proteine con la stessa facilità con cui sequenziamo il DNA".
Usa la forza
Il DNC si basa sulla tecnologia alla base del DNA nanoswitch:un singolo filamento di DNA con "maniglie" molecolari attaccate ad esso in più punti lungo la sua lunghezza. Quando due di queste maniglie si legano l'una all'altra, creano un anello nel filamento di DNA e la lunghezza complessiva del filamento si accorcia. Quando viene applicata la forza per separare le maniglie, il filo si estende alla sua lunghezza originale. La differenza tra la lunghezza del filo negli stati ad anello e non ad anello riflette la dimensione dell'anello, e quindi la distanza tra le maniglie.
Il team di ricerca si è reso conto che potrebbero portare i nanoswitch del DNA un ulteriore passo avanti:se invece progettassero le maniglie per legarsi a una biomolecola, le maniglie potrebbero effettivamente "pizzicare" la molecola tra loro come le due punte di un calibro, piuttosto che legarsi a ciascuna Altro. Misurando il modo in cui l'aggiunta della molecola bersaglio tra le maniglie ha modificato la lunghezza complessiva del nanoswitch del DNA nei suoi stati in loop e non in loop, il team ha ipotizzato di poter misurare efficacemente le dimensioni della molecola.
"In un certo senso, i nanoswitch del DNA sfruttano uno dei metodi meccanici più classici per misurare gli oggetti:basta applicare la forza a qualcosa e vedere come cambia in risposta", ha affermato il co-primo autore Darren Yang, ricercatore post-dottorato presso il Wyss Institute e BCH. "È un approccio che non abbiamo mai visto utilizzato nel campo della proteomica a singola molecola, perché applicare la forza a oggetti così piccoli è incredibilmente impegnativo. Ma siamo stati all'altezza della sfida."
Per trasformare in realtà la loro idea di una nuova tecnica di misurazione basata sulla forza, Yang e i suoi colleghi hanno prima collegato due diversi tipi di impugnature a una molecola bersaglio:una maniglia "forte" per ancorare saldamente la molecola a un'estremità del DNC e diverse maniglie "deboli" che potrebbero essere collegate all'altra estremità del DNC. Hanno quindi legato entrambe le estremità del DNC a due perline "intrappolate otticamente" sospese in raggi laser. Avvicinando le perline, hanno indotto una delle maniglie deboli della molecola bersaglio a legarsi al DNC, creando uno stato ad anello. Quando hanno quindi aumentato la forza spostando ulteriormente le perline, l'impugnatura debole alla fine ha rilasciato il suo legame, riportando il DNC al suo stato più lungo e senza anello.
Il team ha prima testato questa tecnica su semplici molecole di DNA a filamento singolo (ssDNA) e ha confermato che il cambiamento nelle misurazioni della distanza tra lo stato in loop e non in loop del DNC era direttamente correlato alla lunghezza della molecola bersaglio. Queste variazioni di lunghezza possono essere misurate con una precisione a livello di angstrom (che è dieci volte inferiore alla larghezza di una doppia elica del DNA), consentendo l'identificazione di variazioni di lunghezza piccole quanto quella di un singolo nucleotide.
Poiché la molecola bersaglio contiene più maniglie deboli che possono legarsi al DNC, cicli ripetuti di legame e rottura di quelle maniglie creano una serie di misurazioni della distanza tra l'impugnatura forte e le maniglie deboli che sono uniche per ciascuna molecola misurata. Questa "impronta digitale" può essere utilizzata per identificare una molecola nota all'interno di un campione o per dedurre informazioni strutturali su una molecola sconosciuta.
Proteine di sondaggio
Avendo confermato che il DNC potrebbe misurare in modo affidabile le dimensioni delle molecole di DNA, i ricercatori hanno spostato l'attenzione sul loro vero obiettivo:le proteine. Hanno progettato un peptide sintetico (una catena corta di aminoacidi) con una lunghezza e una sequenza note e hanno ripetuto l'esperimento, attaccandolo a un'estremità del DNC via l'impugnatura forte e attaccando e rompendo ripetutamente i legami tra le sue impugnature deboli e il DNC applicando diverse quantità di forza. Hanno scoperto che tutte le distanze misurate dal loro strumento tra le impugnature forti e deboli corrispondevano alle distanze previste in base alla lunghezza del DNC e alle lunghezze degli amminoacidi nel peptide. Hanno anche ottenuto risultati simili quando hanno utilizzato il DNC per misurare un peptide linearizzato presente in natura chiamato NOXA BH3.
Questo processo ha anche generato impronte digitali di misurazione univoche per ciascun peptide. Il team ha creato un modello computerizzato per prevedere quante proteine umane potrebbero essere identificate in modo univoco utilizzando questo metodo e ha scoperto che oltre il 75% delle proteine in un database di proteine comunemente utilizzato potrebbe essere identificato tramite impronte con una probabilità di almeno il 90%.
"In realtà siamo rimasti alquanto sorpresi dal modo in cui questa tecnica ha funzionato", ha affermato il co-primo autore Prakash Shrestha, Ph.D., Postdoctoral Fellow presso il Wyss Institute e BCH. "Le pinzette ottiche esistono da decenni e il ciclo del DNA tra uno stato in loop e uno non in loop esiste da circa 10 anni, e non eravamo sicuri di poter ottenere misurazioni sufficientemente ad alta risoluzione combinando queste idee. Ma si è scoperto che queste impronte digitali sono molto efficaci per identificare le proteine."
Identificare singole molecole proteiche è di per sé un'impresa impressionante, ma essere in grado di farlo per più proteine contemporaneamente è il vero Santo Graal per la proteomica a singola molecola. Il team ha inoltre dimostrato che sostituendo le sfere ottiche con un sistema di pinzette magnetiche, sono stati in grado di eseguire misurazioni su più peptidi diversi in parallelo, nonché di determinare le concentrazioni relative di diverse molecole.
"La proteomica di una singola molecola è ancora in gran parte un sogno irrealizzabile a causa delle sfide nel ridimensionamento e nella risoluzione. Il nostro presente lavoro mostra che il fingerprinting di sequenza basato sulla forza ha il potenziale per realizzare questo sogno", ha affermato il co-autore William Shih, Ph.D. , un Core Faculty Member presso il Wyss Institute e Professore presso HMS e il Dana-Farber Cancer Institute. "La nostra ambizione finale è quella di leggere in modo efficiente non solo le sequenze proteiche, ma anche le strutture proteiche in modo ad alto rendimento."
Il prossimo passo degli scienziati verso questo obiettivo è convalidare i loro calibri per misurazioni strutturali a bassa forza sulle proteine ripiegate e sui loro complessi, studiando il loro potenziale utilizzo per la biologia strutturale e la proteomica. Stanno anche lavorando per aumentare la produttività della tecnologia per accelerare ulteriormente l'analisi di campioni misti.
"Questa ricerca integra la biofisica molecolare con la nanotecnologia del DNA all'avanguardia sperimentata qui al Wyss Institute per consentirci di interagire e analizzare le molecole biologiche in un modo davvero nuovo. Quando William e Wesley hanno proposto per la prima volta questa idea come una sfida fondamentale per la neonata Molecular Robotics Initiative, sembrava davvero fantascienza, ma questo è esattamente il tipo di progetto che vogliamo intraprendere al Wyss. Sono molto orgoglioso del team per aver reso questa tecnologia una realtà:ha il potenziale per cambiare totalmente come facciamo scienza e sviluppiamo terapie", ha affermato il direttore fondatore di Wyss Don Ingber, M.D., Ph.D., che è anche Judah Folkman Professor of Vascular Biology presso la Harvard Medical School e BCH, e Professor of Bioengineering presso la Harvard John A. Paulson Scuola di Ingegneria e Scienze Applicate. + Esplora ulteriormente