Nelle piante e negli animali, le unità fondamentali di confezionamento del DNA, che trasportano le informazioni genetiche, sono i cosiddetti nucleosomi. Un nucleosoma è costituito da un segmento di DNA avvolto attorno a otto proteine chiamate istoni.
Durante l'espressione genica (il processo alla base della produzione delle proteine), i nucleosomi sono coinvolti in vari cambiamenti strutturali dinamici, come lo scorrimento dei nucleosomi, lo svolgimento del DNA e altre interazioni DNA-istone. Di particolare importanza in questi processi sono le strutture terminali, o code, degli istoni.
Le code degli istoni subiscono modifiche chimiche, cambiando la funzionalità dell'istone secondo necessità. Studi dettagliati, e soprattutto visualizzazioni, delle dinamiche dei nucleosomi sono cruciali per comprendere meglio il ruolo delle code degli istoni.
Mikihiro Shibata dell'Università di Kanazawa e colleghi sono ora riusciti a effettuare registrazioni video di nucleosomi senza coda, dimostrando che l'assenza di code istoniche aumenta significativamente l'attività dinamica di un nucleosoma. Lo studio è pubblicato su Nano Letters .
Gli scienziati hanno utilizzato la microscopia a forza atomica ad alta velocità (HS-AFM), un potente strumento di nanoimaging per visualizzare le strutture molecolari e la loro dinamica ad alta risoluzione spaziale e temporale.
Per questo, i nucleosomi dovevano essere messi su un substrato. Shibata e colleghi hanno utilizzato come substrato un film dei cosiddetti areni pilastro[5] (molecole con struttura tubolare pentagonale), formando una superficie ideale in cui i nucleosomi vengono facilmente adsorbiti senza che i processi dinamici vengano soppressi.
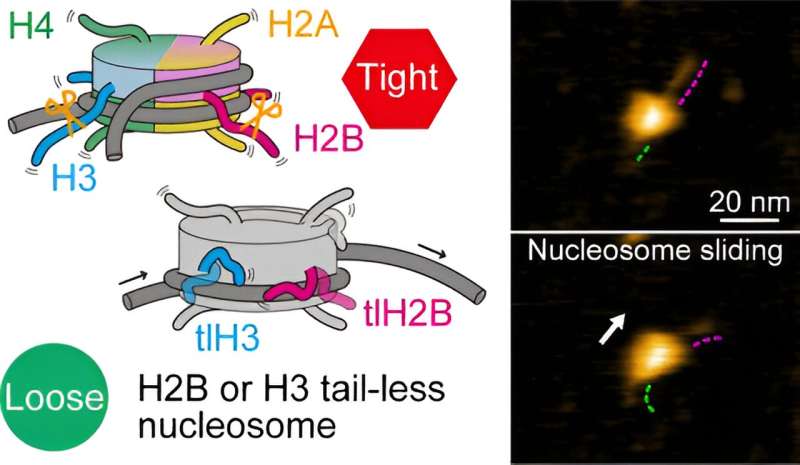
I ricercatori hanno prima esaminato i nucleosomi per i quali tutti gli otto istoni erano privi di coda. Sulla base delle loro osservazioni HS-AFM, hanno concluso che lo scorrimento dei nucleosomi e lo svolgimento/riavvolgimento del DNA si sono verificati più spesso rispetto ai nucleosomi normali (canonici). Ciò suggerisce che senza code, l'interazione istone-DNA è indebolita, portando a una situazione in cui il DNA può staccarsi più facilmente dagli istoni.
Per comprendere meglio i ruoli delle specifiche code degli istoni, Shibata e colleghi hanno preparato nucleosomi in cui un tipo di istone era senza coda. Esistono quattro diversi tipi di istoni, chiamati H2A, H2B, H3 e H4. Esperimenti HS-AFM sui nucleosomi hanno rivelato che i nucleosomi senza coda H2B e H3 mostravano una maggiore frequenza di dinamica. Al contrario, ciò significa che gli istoni canonici H2B e H3 sono essenziali per la stabilità del nucleosoma.
Gli scienziati sottolineano che non sono riusciti a osservare alcun movimento effettivo delle code degli istoni:molto probabilmente la risoluzione temporale dello studio, 0,3 secondi, era molto più lenta della velocità della dinamica di avvolgimento/svolgimento delle code. Nonostante questa limitazione, il lavoro di Shibata e colleghi dimostra chiaramente che le code degli istoni H2B e H3 contribuiscono principalmente alla dinamica dei nucleosomi.
Per quanto riguarda il lavoro futuro, citando i ricercatori, "una tecnica per contrassegnare le punte delle code degli istoni potrebbe consentire all'HS-AFM di catturare i movimenti delle code degli istoni stesse."
Microscopia a forza atomica ad alta velocità
Il principio generale della microscopia a forza atomica (AFM) è quello di effettuare una scansione con una punta molto piccola della superficie di un campione. Durante questa scansione orizzontale (xy), la punta, che è fissata a un piccolo cantilever, segue il profilo verticale (z) del campione, inducendo una forza sul cantilever che può essere misurata.
L'entità della forza nella posizione xy può essere correlata al valore z; i dati xyz generati durante una scansione risultano quindi in una mappa di altezza che fornisce informazioni strutturali sul campione analizzato. Nell'AFM ad alta velocità (HS-AFM), il principio di funzionamento è leggermente più complicato:il cantilever viene fatto oscillare vicino alla sua frequenza di risonanza.
Quando la punta viene spostata attorno a una superficie, vengono registrate le variazioni nell'ampiezza (o nella frequenza) dell'oscillazione del cantilever, risultante dall'interazione della punta con la superficie del campione, poiché forniscono una misura per il valore z locale. L'AFM non coinvolge lenti, quindi la sua risoluzione non è limitata dal cosiddetto limite di diffrazione come, ad esempio, nella diffrazione dei raggi X.
HS-AFM produce un video, in cui l'intervallo di tempo tra i fotogrammi dipende dalla velocità con cui una singola immagine può essere generata (mediante la scansione xy del campione).
I ricercatori del Nano Life Science Institute (WPI-NanoLSI), dell'Università di Kanazawa, negli ultimi anni hanno sviluppato ulteriormente HS-AFM, in modo che possa essere applicato per studiare molecole biochimiche e processi biomolecolari in tempo reale. Mikihiro Shibata e colleghi hanno ora applicato il metodo per studiare in dettaglio la dinamica dei nucleosomi e in particolare il ruolo delle terminazioni molecolari degli istoni, proteine che svolgono un ruolo cruciale nell'accessibilità del DNA.
Ulteriori informazioni: Shin Morioka et al, La microscopia a forza atomica ad alta velocità rivela lo scorrimento dei nucleosomi e le dinamiche di scarto/avvolgimento del DNA dei nucleosomi senza coda, nanolettere (2024). DOI:10.1021/acs.nanolett.4c00801
Informazioni sul giornale: Nanolettere
Fornito dall'Università di Kanazawa