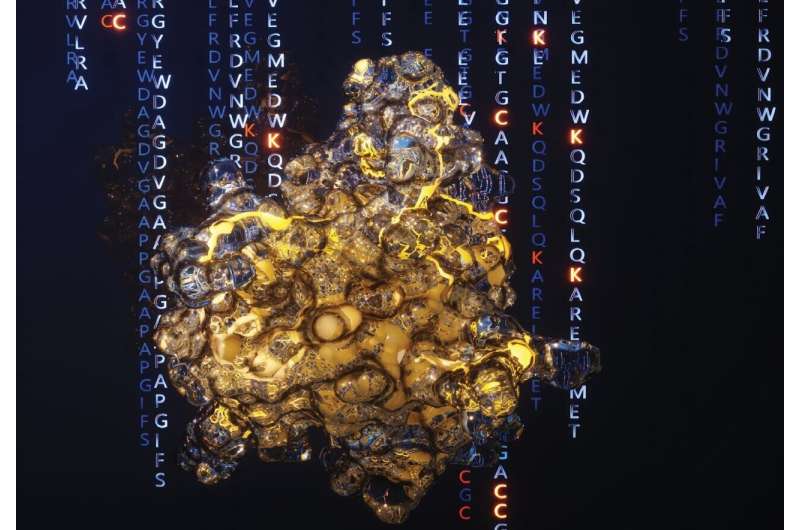
In uno studio pubblicato su Nature Nanotechnology , gli scienziati dell'Università di Tecnologia di Delft presentano una nuova tecnica per identificare le proteine. Le proteine svolgono funzioni essenziali nelle nostre cellule, mentre svolgono un ruolo cruciale in malattie come il cancro e l’infezione da COVID-19. I ricercatori identificano le proteine leggendo l'impronta digitale e confrontando l'impronta digitale con i modelli di un database.
Usando questa nuova tecnologia, i ricercatori possono identificare proteine individuali, intatte e a lunghezza intera, preservandone tutte le informazioni. Ciò può far luce sui meccanismi alla base di molte malattie diverse e consentire una diagnosi precoce.
"Lo studio delle proteine all'interno delle cellule è stato un argomento caldo per decenni e ha fatto enormi progressi, consentendo ai ricercatori di avere un'idea molto migliore su che tipo di proteine esistono e quale funzione svolgono", afferma Mike Filius, primo autore dell'articolo.
Attualmente, gli scienziati utilizzano un metodo chiamato spettrometria di massa per identificare le proteine. L'approccio più comune della spettrometria di massa è l'approccio "dal basso verso l'alto", in cui le proteine a lunghezza intera vengono tagliate in frammenti più piccoli, chiamati peptidi, che vengono poi misurati dallo spettrometro di massa. Sulla base dei dati di questi piccoli frammenti, un computer ricostruisce la proteina.
Filius afferma:"Questo è un po' simile al tipico progetto IKEA, in cui ti restano sempre alcuni pezzi di ricambio in cui non sei sicuro di come inserirli. Ma nel caso delle proteine, questi pezzi di ricambio possono effettivamente contenere molto informazioni preziose, ad esempio se una tale proteina ha o meno una struttura dannosa che causa una malattia."
L'impronta digitale delle proteine
"Per identificare una proteina, non è necessario conoscere tutti gli aminoacidi, gli elementi costitutivi di qualsiasi proteina. Si cerca invece di ottenere informazioni sufficienti in modo da poter identificare la proteina utilizzando un database come riferimento, simili a come la polizia può scoprire l'identità di un sospettato attraverso un'impronta digitale", spiega Filius.
"In lavori precedenti, abbiamo dimostrato che ogni proteina ha un'impronta digitale unica, proprio come l'analogo umano. Ci siamo resi conto che abbiamo solo bisogno di conoscere la posizione di alcuni di tutti gli amminoacidi di una proteina per generare un'impronta digitale unica da che possiamo identificare la proteina," aggiunge Raman van Wee, Ph.D. candidato coinvolto nella ricerca.
"Possiamo rilevare questi amminoacidi attraverso molecole che si illuminano al microscopio e sono attaccate a piccoli pezzi di DNA che si legano in modo molto specifico a un determinato amminoacido", spiega Van Wee. In questo modo, il team può determinare molto rapidamente la posizione dell'amminoacido con grande precisione.
"Poiché la sensibilità di questa nuova tecnica, chiamata FRET X, è superiore a quella dei metodi convenzionali come la spettrometria di massa, possiamo rilevare concentrazioni di proteine molto più basse in una miscela di molte altre biomolecole e richiedere solo una piccola quantità di campione," Filius dice. Questo è importante perché rende a portata di mano la misurazione dei campioni dei pazienti in caso di malattia.
"Nel nostro articolo dimostriamo che possiamo rilevare piccole quantità di proteine caratteristiche del morbo di Parkinson o dell'infezione da COVID-19", afferma Filius.
"Mentre ci sono altri approcci allo studio per identificare le proteine, il nostro si concentra sull'identificazione di proteine intatte e singole in una miscela complessa. Possiamo cercare un ago in un pagliaio", aggiunge Van Wee.
Sebbene promettente, la ricerca richiede ancora uno sviluppo sostanziale, su cui il Chirlmin Joo Lab non vede l’ora di lavorare. Il gruppo di ricerca ha parlato con diverse parti interessate nei laboratori clinici e nell'industria biofarmaceutica e ha appreso che sono davvero entusiasti del potenziale rivoluzionario della tecnologia.
Stanno anche lavorando al lancio di una start-up per sviluppare FRET X in una piattaforma per il rilevamento di proteine altamente sensibili. Questa piattaforma può diagnosticare le malattie nelle prime fasi, migliorando l'efficacia del potenziale trattamento.
"Questa tecnica innovativa decifra il codice delle proteine e apre interessanti possibilità per il rilevamento precoce delle malattie", afferma Chirlmin Joo, supervisore del progetto.
Ulteriori informazioni: Mike Filius et al, Impronta digitale completa di proteine a singola molecola, Nature Nanotechnology (2024). DOI:10.1038/s41565-023-01598-7
Informazioni sul giornale: Nanotecnologia naturale
Fornito dall'Università della Tecnologia di Delft