In una nuova Nature Communications studio, i ricercatori hanno esplorato la costruzione di circuiti genetici su singole molecole di DNA, dimostrando la sintesi proteica localizzata come principio guida per i nanodispositivi dissipativi, offrendo approfondimenti sulla progettazione delle cellule artificiali e sulle applicazioni nanobiotecnologiche.
Il termine "circuito genetico" è una descrizione metaforica della complessa rete di elementi genetici (come geni, promotori e proteine regolatrici) all'interno di una cellula che interagiscono per controllare l'espressione genica e le funzioni cellulari.
Nel campo della progettazione delle cellule artificiali, gli scienziati mirano a replicare e progettare questi circuiti genetici per creare unità funzionali e autonome. Questi circuiti fungono da macchinario molecolare responsabile dell'orchestrazione dei processi cellulari regolando con precisione la produzione di proteine e altre molecole.
Comprendendo e manipolando questi circuiti, i ricercatori possono progettare cellule artificiali con comportamenti programmabili, imitando le funzionalità delle cellule naturali.
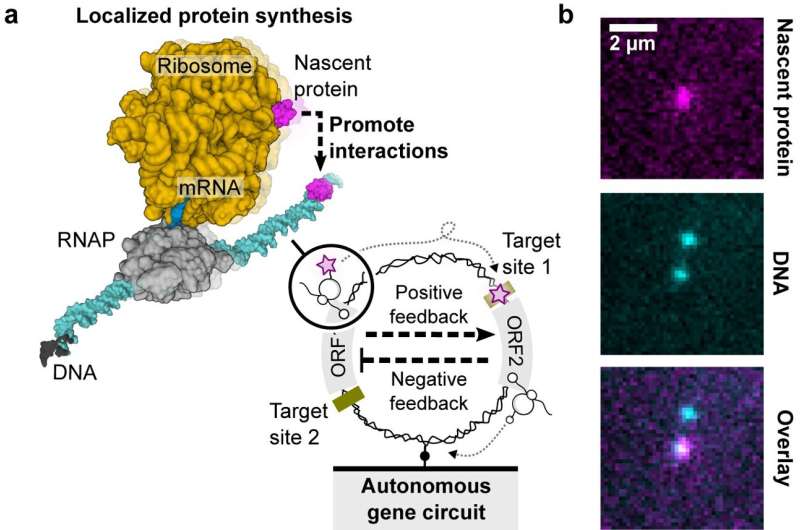
Nell'ambito dello studio citato l'attenzione si concentra sulla costruzione di circuiti genetici su singole molecole di DNA. Questo rappresenta un approccio nuovo in quanto si allontana dal contesto cellulare tradizionale ed esplora la possibilità di creare circuiti genetici in condizioni prive di cellule.
Il primo autore, il dottor Ferdinand Greiss del Weizmann Institute of Science in Israele, ha spiegato a Phys.org la motivazione dei ricercatori:"Stiamo cercando di ricostituire i processi biologici al di fuori dei complessi circuiti delle cellule viventi, si spera di migliorare la nostra comprensione dei principi guida della natura. la ricerca è diretta alla costruzione di future cellule artificiali e singole molecole di DNA potrebbero costituirne la base genetica."
La regolazione genetica è il processo mediante il quale le cellule controllano l'espressione dei geni, determinando quando e in che misura l'informazione di un gene viene utilizzata nella sintesi di molecole funzionali come proteine o RNA. Svolge un ruolo cruciale nel mantenimento delle funzioni cellulari, nella risposta ai cambiamenti ambientali e nel garantire un corretto sviluppo.
La regolazione dell'espressione genica coinvolge la trascrizione e la traduzione. Durante la trascrizione, un segmento specifico di DNA funge da modello per la sintesi di molecole di mRNA complementari da parte della RNA polimerasi. Questo mRNA trasporta il codice genetico dal nucleo al citoplasma, dove avviene la traduzione.
La traduzione comporta la conversione dell'mRNA in proteine. I ribosomi leggono la sequenza dell'mRNA, facilitando l'assemblaggio degli amminoacidi in una catena polipeptidica, formando la proteina codificata dal gene.
"Nei sistemi procariotici, i processi di trascrizione e traduzione sono accoppiati. Ciò significa che una volta che la RNA polimerasi produce mRNA dal DNA, il ribosoma può trovare il sito di legame ribosomiale sull'mRNA nascente per iniziare a sintetizzare la proteina. La proteina nascente può ripiegarsi e funzionare mentre è ancora legata al DNA dal complesso RNA polimerasi-mRNA-ribosoma Dopo la conclusione della trascrizione o della traduzione, la proteina nascente cade dal DNA e si disperde nella soluzione sfusa," ha spiegato la coautrice Dr. Shirley Shulman Daube del progetto. Istituto Weizmann delle Scienze in Israele.
Il significato risiede nell’aumento della concentrazione locale di proteine nascenti, che è circa 1.000 volte superiore rispetto alla massa della soluzione circostante. Questa organizzazione spaziale e questo aumento della concentrazione potrebbero avere implicazioni per le funzioni cellulari e potenzialmente svolgere un ruolo nella costruzione di cellule artificiali utilizzando singole molecole di DNA.
"I circuiti genetici si basano su molecole geneticamente codificate, come i fattori di trascrizione, che sono prodotti dal DNA e si legano al DNA per regolare la produzione propria e di altre molecole", ha affermato il coautore Dr. Vincent Noireaux dell'Università del Minnesota .
Per costruire il circuito genetico su una singola molecola di DNA, i ricercatori hanno progettato sequenze specifiche con i geni del batteriofago lambda (E. coli).
Il circuito genetico prevedeva una cascata negativa, guidata dal gene repressore CI e dal suo sito di legame con l'operatore, che controllava in modo complesso il gene HT. Questo gene HT codificava la proteina HaloTag (HT), un elemento cruciale per visualizzare le proteine nascenti su singole molecole di DNA.
Lo studio ha implementato condizioni rigorose, inclusa la bassa densità superficiale del DNA, per garantire una sintesi proteica localizzata precisa.
Contemporaneamente, si è svolta una cascata positiva con la fusione dell'RNA polimerasi del batteriofago T7 (HT-T7 RNAP) e della proteina HT, consentendo il monitoraggio in tempo reale dell'espressione genica attraverso un gene reporter a valle, GFP.
Un colorante fluorogenico lontano rosso (MaP655-Halo) ha migliorato il rilevamento delle proteine nascenti, fornendo una visione completa delle dinamiche del circuito genetico.
La cascata negativa, o soppressione, regola e inibisce la produzione di proteine specifiche in determinate condizioni. D'altra parte, le cascate positive contribuiscono all'attivazione e all'espressione di geni specifici all'interno del circuito genetico.
La ricerca è andata oltre la semplice osservazione, incorporando un circuito di feedback dotato di un repressore dCro sintetico. Questo componente era cruciale nella regolazione dell'espressione genetica attraverso un promotore sintetico meticolosamente progettato.
I ricercatori hanno scoperto che la sintesi proteica localizzata su una singola molecola di DNA può guidare i circuiti genetici in condizioni prive di cellule senza il confinamento dei compartimenti cellulari. La dinamica dei circuiti genetici è stata osservata meticolosamente in condizioni molto diluite.
L'autore principale, il dottor Roy Bar-Ziv del Weizmann Institute of Science in Israele, ha sottolineato l'importanza delle loro scoperte:"La regolazione dell'espressione genetica dipende dalle proteine che si legano al DNA, bloccando o aumentando l'attività di un gene. Il legame richiede un'elevata concentrazioni di proteine per trovare e legare sequenze specifiche sulla molecola di DNA. Inaspettatamente, scopriamo che la sintesi proteica localizzata può aumentare transitoriamente la concentrazione abbastanza a lungo da consentire alle proteine di fare lo stesso senza confinamento cellulare."
In sostanza, la scoperta sfida l'idea convenzionale secondo cui alte concentrazioni sono essenziali per la regolazione genetica, introducendo un nuovo aspetto della sintesi proteica localizzata come mezzo per influenzare i circuiti genetici in condizioni prive di cellule.
Per il lavoro futuro, i ricercatori prevedono di sfruttare la sintesi proteica localizzata come principio guida per migliorare la funzionalità delle cellule artificiali costruite da singole molecole di DNA, affrontando le sfide a basse concentrazioni. Prevedono inoltre potenziali applicazioni in nanodispositivi autocodificati e intendono esplorare le correlazioni tra la struttura del DNA, la dinamica dell'espressione genetica e la sintesi proteica.
La ricerca ha coinvolto anche il contributo di Nicolas Lardon con il Prof. Kai Johnsson dell'MPI for Medical Research, che ha sviluppato il colorante fluorogenico (MaP655-Halo); Yoav Barak, che ha contribuito a ottimizzare la preparazione del DNA; e Leonie Schütz con il Prof. Elmar Weinhold, che ha aperto la strada allo sviluppo di metiltransferasi per modifiche del DNA sito-specifiche con biotine.
Ulteriori informazioni: Ferdinand Greiss et al, Un circuito genetico su una singola molecola di DNA come nanodispositivo dissipativo autonomo, Nature Communications (2024). DOI:10.1038/s41467-024-45186-2
Informazioni sul giornale: Comunicazioni sulla natura
© 2024 Rete Scienza X