Un gruppo di ricerca guidato dall'Università di Scienza e Tecnologia di Hong Kong (HKUST) ha sviluppato una piattaforma di spettroscopia Raman ottimizzata per superficie (SERS) controllata da pinzette plasmoniche ottiche che utilizza il controllo on-off della luce per sondare varie specie di amilina nelle miscele a livello di singola molecola, svelando le strutture eterogenee delle specie di amilina dipendenti dal pH e i segreti dietro i meccanismi di aggregazione dell'amiloide associati al diabete di tipo 2.
Rimuovendo la media d'insieme, le tecniche a singola molecola discernono il segnale delle singole molecole per svelare dettagli nascosti e rivoluzionare la nostra comprensione di sistemi molecolari complessi ed eterogenei. Gli attuali approcci a singola molecola sono limitati all'ultra-diluizione e/o all'immobilizzazione molecolare perché il volume di rilevamento limitato dalla diffrazione non può essere ulteriormente ridotto.
Alcune biomolecole, invece, intraprendono varie interazioni che sono significativamente influenzate dalle concentrazioni. Ad esempio, essendo una tipica proteina intrinsecamente disordinata, il polipeptide amiloide delle isole umane (amilina, hIAPP) è privo di strutture secondarie stabili ma possiede una propensione all'aggregazione governata da fattori ambientali, come concentrazione e pH, per formare diversi intermedi oligomerici e fibrille amiloidi nel diabete di tipo II pazienti.
Il meccanismo molecolare rimane poco chiaro, a causa delle difficoltà legate al rilevamento delle specie di amilina rare, transitorie ed eterogenee in una miscela dinamica, che richiedono lo sviluppo di metodi avanzati a singola molecola.
In una recente svolta, il gruppo di ricerca guidato dal Prof. Huang Jinqing, Professore assistente presso il Dipartimento di Chimica di HKUST, ha sviluppato con successo una nuova piattaforma a molecola singola che combina la manipolazione plasmonica ottica e la misurazione SERS per ridurre il volume di rilevamento ed aumentare il miglioramento del segnale, consentendo un'efficienza e Caratterizzazione di singole molecole ad alto rendimento per studiare specie di amilina dipendenti dal pH a concentrazioni fisiologiche.
Nello specifico, il team ha costruito una giunzione plasmonica tra due microsfere di silice rivestite con nanoparticelle di Ag per intrappolare un'ulteriore nanoparticella di Ag per formare una nanocavità dinamica dopo l'irradiazione laser, che potrebbe incapsulare una o poche molecole per caratterizzazioni SERS sensibili.
Poiché sia l'intrappolamento plasmonico ottico che i fenomeni SERS sono confinati spazialmente su scala nanometrica, supera il limite di diffrazione ottica per consentire un controllo preciso della posizione, ridurre al minimo il volume di rilevamento e aumentare contemporaneamente il miglioramento SERS.
Inoltre, i dimeri di microsfere di silice rivestite con nanoparticelle di Ag costruiti sono più stabili rispetto alle nanoparticelle di Ag convenzionali assemblate in soluzioni, rendendo più facile osservare e localizzare la giunzione plasmonica sui normali microscopi per migliorare l'efficienza e la riproducibilità. Commutando la luce laser tra gli stati "acceso" e "spento", i ricercatori possono controllare l'intrappolamento ottico plasmonica per modulare l'assemblaggio e il disassemblaggio della nanocavità dinamica per il campionamento ad alto rendimento e misurazioni SERS simultanee.
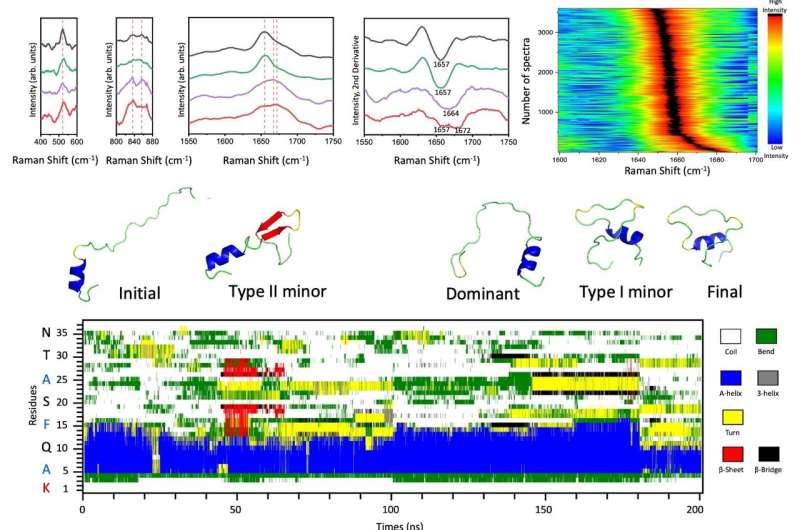
Utilizzando questa efficiente piattaforma a molecola singola, il gruppo di ricerca ha sfruttato una quantità statisticamente significativa di spettri SERS alla base delle caratteristiche strutturali di varie specie di amilina in due distinte condizioni fisiologiche:i granuli secretori delle cellule β pancreatiche a pH 5,5 e i compartimenti extracellulari a pH 7.4, rispettivamente.
Sono stati identificati due tipi di specie di amilina a bassa popolazione a partire dai loro monomeri predominanti nella fase iniziale dell'aggregazione dell'amiloide a pH neutro, contenenti una struttura di svolta critica o una corta forcina β con terminale C vincolato, come supportato da simulazioni di dinamica molecolare (MD) .
Uno spostamento così leggero nell’equilibrio tra le diverse specie di amilina potrebbe portare allo sviluppo irreversibile dell’amiloide anche dopo la post-aggiustamento del pH da 7,4 a 5,5. Pertanto, la caratterizzazione strutturale diretta di queste specie di amilina all'interno di miscele eterogenee indica l'impatto del pH sulle loro interazioni intra e intermolecolari e fa luce sul meccanismo alla base dell'aggregazione amiloide regolata dal pH per comprendere il diabete di tipo 2.
"Presentiamo una strategia facile da usare che riduce il volume di rilevamento, migliora il segnale molecolare e aumenta l'efficienza del turnover", ha spiegato il prof. Huang. "La nostra piattaforma a singola molecola può acquisire una grande quantità di spettri SERS come istantanee molecolari, paragonabili a quelle ottenute attraverso simulazioni MD. Analizzando statisticamente i dettagli strutturali a livello di singola molecola, siamo in grado di ricostruire le proprietà di massa e ottenere risultati unici approfondimenti sulla popolazione e sulla probabilità di tipi specifici di molecole all'interno della miscela eterogenea. Ha il potenziale per scoprire misteri nascosti in sistemi complessi."
Lo studio è stato recentemente pubblicato su Nature Communications .
Ulteriori informazioni: Wenhao Fu et al, Caratterizzazione SERS a singola molecola controllata da pinzette plasmoniche ottiche efficienti di specie di amilina pH-dipendenti in ambienti acquosi, Nature Communications (2023). DOI:10.1038/s41467-023-42812-3
Informazioni sul giornale: Comunicazioni sulla natura
Fornito dall'Università della Scienza e della Tecnologia di Hong Kong