
In un nuovo studio, i ricercatori del Dipartimento di Biochimica dell’Indian Institute of Science (IISc), hanno utilizzato una nuova tecnica di imaging per individuare quanto fortemente le basi adiacenti, gli elementi costitutivi del DNA, si accumulano l’una sull’altra in un singolo filamento. . I risultati aprono possibilità per costruire nanodispositivi complessi di DNA e svelare aspetti fondamentali della struttura del DNA.
Dietro il funzionamento ininterrotto di ogni cellula vivente si trova il DNA, il veicolo ereditario che trasporta le informazioni per la sua crescita, funzionamento e riproduzione. Ogni filamento di DNA è solitamente costituito da quattro basi nucleotidiche:adenina (A), guanina (G), timina (T) e citosina (C). Le basi su un filamento si accoppiano con quelle sul filamento opposto per formare il DNA a doppio filamento (A si accoppia con T e G si accoppia con C).
Due tipi di interazioni stabilizzano la struttura a doppia elica del DNA. L'accoppiamento delle basi (interazione tra basi su filamenti opposti) è più ampiamente conosciuto, mentre l'impilamento delle basi, interazione tra basi sullo stesso filamento, non è molto ben studiato. Immagina una cerniera in cui l'accoppiamento delle basi è come la cerniera che tiene insieme i due fili, mentre l'impilamento delle basi agisce come i denti della cerniera, garantendo una connessione salda e sicura.
Le interazioni di impilamento delle basi sono in genere più forti dell'accoppiamento di basi, afferma Mahipal Ganji, professore assistente presso il Dipartimento di Biochimica, IISc e autore corrispondente dell'articolo pubblicato su Nature Nanotechnology .
Per studiare tutte le 16 possibili combinazioni di impilamento delle basi, i ricercatori hanno utilizzato DNA-PAINT (Point Accumulation in Nanoscale Topography). DNA-PAINT è una tecnica di imaging che funziona secondo il principio secondo cui due filamenti di DNA progettati artificialmente, ciascuno terminante su una base diversa, quando messi insieme in una soluzione tampone a temperatura ambiente, si legano e si legano l'uno all'altro in modo casuale per un tempo molto breve .
Il team ha contrassegnato uno dei fili (filo dell'imager) con un fluoroforo che emetteva luce durante il legame e ha testato l'impilamento di questo filo sopra un altro filo ancorato. Il legame e lo scioglimento di diverse combinazioni di filamenti (in base alle basi terminali) sono stati catturati come immagini al microscopio a fluorescenza.
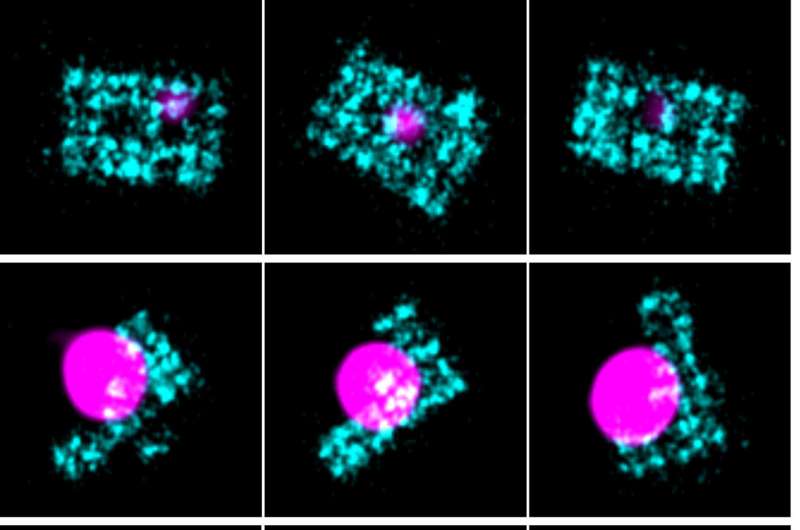
È stato riscontrato che il tempo impiegato per legare e sciogliere i fili aumentava se l'interazione tra le basi impilate era forte, spiega Abhinav Banerjee, primo autore e Ph.D. studente presso il Dipartimento di Biochimica. Pertanto, utilizzando i dati ottenuti da DNA-PAINT, i ricercatori hanno costruito un modello che collegava i tempi di legame e distacco con la forza dell'interazione tra le basi impilate.
Usando questa tecnica, il team è stato in grado di scoprire interessanti spunti sullo stacking di base. Ad esempio, l’aggiunta di un’ulteriore interazione di accumulo di basi a un filamento di DNA sembra aumentarne la stabilità fino a 250 volte. Hanno anche scoperto che ciascuna coppia di nucleotidi aveva la propria forza di impilamento unica. Queste informazioni hanno permesso al team di progettare una nanostruttura di DNA a tre bracci altamente efficiente che potrebbe potenzialmente essere incorporata in un veicolo a forma di poliedro per applicazioni biomediche, come il targeting di specifici marcatori di malattie e la fornitura di terapie mirate.
I ricercatori stanno anche lavorando per migliorare la tecnica stessa del DNA-PAINT. Banerjee afferma che, sfruttando le interazioni di accumulo, intendono progettare nuove sonde che amplierebbero le potenziali applicazioni di DNA-PAINT.
Inoltre, secondo gli scienziati, la ricerca ha applicazioni più ampie che vanno oltre l'imaging e la nanotecnologia. Ganji spera che queste scoperte possano essere utilizzate per studiare le proprietà fondamentali del DNA a singolo e doppio filamento che, a sua volta, potrebbe far luce sui meccanismi di riparazione del DNA, il cui fallimento porta a molte malattie incluso il cancro.
Ulteriori informazioni: Banerjee A, Anand M, Kalita S, Ganji M, Analisi di una singola molecola dell'energia di impilamento delle basi del DNA utilizzando nanostrutture di DNA modellate, Nature Nanotechnology (2023). www.nature.com/articles/s41565-023-01485-1
Informazioni sul giornale: Nanotecnologia naturale
Fornito dall'Indian Institute of Science