I ricercatori della Griffith University hanno svolto un ruolo chiave nell'utilizzo di modelli "origami" di DNA per controllare il modo in cui vengono assemblati i virus.
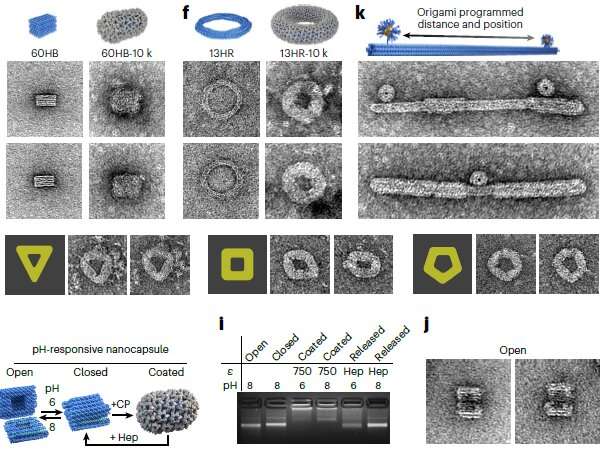
Il team globale dietro la ricerca, intitolata "DNA-origami-directed virus capsid polymorphism", pubblicata su Nature Nanotechnology , ha sviluppato un modo per dirigere l'assemblaggio dei capsidi virali, l'involucro proteico dei virus, in condizioni fisiologiche in modo preciso e programmabile.
Il dottor Frank Sainsbury e la dottoressa Donna McNeale del Griffith Institute for Drug Discovery facevano parte del gruppo di ricerca e hanno affermato che forzare i virus ad assemblarsi sul DNA piegato in forme diverse "come gli origami" è stata una domanda a cui questo progetto ha risposto.
"Abbiamo ottenuto il controllo sulla forma, dimensione e topologia delle proteine virali utilizzando nanostrutture di origami di DNA definite dall'utente come piattaforme di legame e assemblaggio, che sono state incorporate nel capside", ha affermato il dott. Sainsbury.
"I rivestimenti proteici del virus potrebbero proteggere dalla degradazione gli origami di DNA incapsulati.
"Questa attività è più simile all'avvolgimento di un regalo:le proteine del virus si depositano sopra la diversa forma definita dalla forma dell'origami del DNA.
"E le diverse proteine virali sono come diverse carte da imballaggio, il che sarebbe rilevante per diversi usi degli origami di DNA rivestiti."
Un controllo preciso sulle dimensioni e sulla forma delle proteine virali comporterebbe vantaggi nello sviluppo di nuovi vaccini e sistemi di somministrazione.
"Ma gli attuali strumenti per controllare il processo di assemblaggio in modo programmabile erano sfuggenti", ha affermato il dott. McNeale.
"Inoltre, il nostro approccio non si limita a un singolo tipo di unità proteica del capside virale e può essere applicato anche a strutture di origami RNA-DNA per aprire la strada alla protezione del carico e alle strategie di targeting di prossima generazione."
Attualmente, il dottor Sainsbury e il suo team stanno lavorando per acquisire una comprensione più approfondita di come i diversi virus si autoassemblano e di come possono essere utilizzati per incapsulare carichi diversi.
Ciò consentirà loro di progettare e modificare ulteriori particelle simili a virus per una vasta gamma di usi. Ad esempio, hanno scoperto che un virus trovato nei topi è in grado di trasportare carichi proteici attraverso ambienti inospitali e in uno specifico compartimento subcellulare nelle cellule umane.
"Con l'enorme spazio di progettazione esistente tra i virus che potrebbero essere utilizzati come vettori, c'è ancora molto da imparare dal loro studio. Continueremo a espandere i confini di come le particelle simili ai virus possono assemblarsi e cosa si può imparare dal loro utilizzo" come trasportatori di medicinali, vaccini e recipienti per reazioni biochimiche", ha affermato il dottor Sainsbury.
La fase successiva della ricerca del team GRIDD utilizzerà questo approccio per capire perché i virus non si assemblano essi stessi in forme diverse.
Ulteriori informazioni: Polimorfismo del capside del virus diretto da origami del DNA, Nature Nanotechnology (2023). DOI:10.1038/s41565-023-01443-x, https://www.nature.com/articles/s41565-023-01443-x
Informazioni sul giornale: Nanotecnologia naturale
Fornito dalla Griffith University