Giocare ai videogiochi non è esattamente scienza missilistica ma, grazie a Foldit, può essere biologia molecolare.
Una specie di.
Sviluppato nel 2008 dai ricercatori dell'Università di Washington, il gioco multiplayer online aumenta l'abilità della mente umana per il ragionamento spaziale grazie alle capacità intellettuali di decine di migliaia di giocatori. Solo e in squadra, questi analisti dilettanti fanno a gara per risolvere i più sconcertanti enigmi che oggi assillano i biologi molecolari:come si ripiegano le singole proteine e gli amminoacidi che li compongono.
La risposta vale più che vantarsi o sbloccare obiettivi (i giochi parlano per quando un giocatore completa un compito particolarmente fantastico); fornisce i mezzi per fornire farmaci che potrebbero un giorno fermare malattie come il virus dell'immunodeficienza umana (HIV) che muoiono di colpo.
Non c'è da meravigliarsi se un tale brusio è emerso quando una pubblicazione del 2011 sulla rivista Nature Structural &Molecular Biology ha riferito che i giocatori di Foldit avevano svelato una proteina chiave in Virus della scimmia Mason-Pfizer ( MPMV ), la versione scimmiesca dell'HIV, che aveva ostacolato i ricercatori per più di un decennio.
Come John Henry contro il martello a vapore o Garry Kasparov contro Deep Blue, I giocatori di Foldit hanno dimostrato che gli umani hanno ancora qualcosa da insegnare alle macchine; a differenza di Enrico, chi è morto, o Kasparov, che ha perso in una rivincita, i giocatori che ripiegano le proteine hanno ancora un vantaggio rispetto allo sgranocchiare numeri a forza bruta dei supercomputer.
Per comprendere la portata di questo risultato e cosa potrebbe significare per il futuro dell'HIV, diamo un'occhiata al motivo per cui è così importante capire come si ripiega una proteina.
Le proteine sono responsabili di numerose funzioni nel corpo, tutto, dalla conversione del cibo in energia alla consegna di messaggi chimici. L'origami peculiare di ogni proteina determina sia il suo ruolo che la sua capacità di connettersi con altre molecole. È come se una proteina fosse una catena composta da mille ciocche, tutti raggruppati in una palla:se volessi progettare un farmaco per influenzarlo, avresti bisogno di sapere quali serrature sono state rivolte verso l'esterno, e con quale schema, in modo da poter tagliare un mazzo di chiavi per adattarli.
Particolari proteine svolgono un ruolo fondamentale nelle catene chiave degli eventi. I ricercatori apprezzano queste proteine perché rappresentano una vulnerabilità che possono sfruttare per rallentare o fermare una malattia, Compreso retrovirus come HIV e MPMV. UN retrovirus è un virus che trasporta le sue informazioni genetiche come acido ribonucleico (RNA) anziché DNA. Questi virus trascrivono il loro RNA in DNA, invece che viceversa, intrappolando permanentemente il loro codice genetico nel genoma della cellula infetta e trasformandolo in una fabbrica per produrre più retrovirus.
I retrovirus si basano su un catalizzatore proteico chiamato enzima proteasi come parte del loro processo riproduttivo. L'inibizione di quella proteina getta una chiave inglese nel meccanismo di distruzione di un retrovirus. Infatti, tali inibitori della proteasi sono già usati per trattare l'infezione da HIV nei pazienti con AIDS [fonte:Britannica].
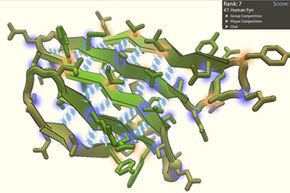
Sfortunatamente, scoprire la struttura di tali proteine è uno degli enigmi più difficili che conosciamo. Immagina di riempire una scatola gigante con luci aggrovigliate dell'albero di Natale, giocattoli Slinky in disuso, filo spinato, nastro adesivo ed elettromagneti, poi scuotendolo e rigirandolo, e infine cercando di indovinare che forma avevi fatto. Hai appena iniziato a grattare la superficie della complessità di questo compito. Le proteine possono essere costituite da più di 10, 000 atomi a testa, formare catene, sottocatene e legami in innumerevoli combinazioni; infatti, ci sono più modi per piegare una proteina di quanti atomi ci siano nell'universo [fonte:Bohannon].
Tale complessità è più di quanto anche un supercomputer a volte possa gestire, soprattutto perché i computer non sono particolarmente bravi a lavorare con forme tridimensionali. Così, gli scienziati hanno iniziato a cercare un mezzo più rapido ed efficace per rompere le strutture proteiche. La loro soluzione? Usa le capacità innate di analisi spaziale del cervello umano. Nasce Foldit. Quasi immediatamente, ha cominciato a pagare dividendi.
In questa prossima sezione, daremo un'occhiata più da vicino a come funziona Foldit, ciò che i giocatori hanno ottenuto con esso e se hanno curato o meno l'HIV.
Una parte essenziale del mestiere dell'orologiaio consiste nell'assemblare una collezione di parti delicate all'interno di uno spazio il più compatto possibile, garantendo nel contempo che i quarti stretti non interferiscano con la funzione dell'orologio.
In Foldit, i giocatori usano una semplice scatola di strumenti per manipolare la forma di una proteina. L'idea è di piegarsi, intrecciare, muovere e scuotere le catene laterali della proteina e le spine dorsali degli amminoacidi in modo tale che l'intera struttura sia compattata nella sua forma ottimale. I giocatori sanno che la loro soluzione funziona quando eliminano le collisioni tra le catene laterali di atomi, nascondere le catene idrofobiche all'interno della proteina, affrontare le catene idrofile verso l'esterno e rimuovere grandi spazi vuoti che minacciano la stabilità della proteina, il che si riflette nel loro punteggio.
Il punteggio, insieme alle regole che disciplinano le mosse consentite, deriva dalle leggi della fisica che regolano il ripiegamento delle proteine. La termodinamica ci dice che i sistemi naturali tendono verso stati di energia più bassa. Altre leggi fisiche, come l'attrazione reciproca di cariche opposte, repulsione di cariche simili e limitazioni su come i legami atomici possono essere organizzati e ruotati, sono anche incorporati.
Il programma Foldit astrae i dettagli in una forma che l'occhio può percepire e il cervello può afferrare. La fisica viene gestita dietro le quinte, liberando i giocatori di manipolare le forme attraverso un'analisi meticolosa, istinto o qualsiasi altro metodo gli si addice.
Entro un anno dalla sua introduzione, I giocatori di Foldit hanno prodotto soluzioni per il ripiegamento delle proteine che hanno superato quelle presentate dai biologi molecolari. Ispirato dai primi successi, I creatori di Foldit hanno applicato il programma ad altre proteine e hanno incaricato i giocatori di progettare nuove proteine per combattere il cancro, AIDS e morbo di Alzheimer. Per esempio, la proteina soppressore del tumore p53 è danneggiata in molti pazienti oncologici. Se riparato o sostituito, una tale proteina potrebbe fermare la crescita del tumore.
Sconcertare con successo l'enzima proteasi MPMV è la pietra angolare della carriera di Foldit finora. Prima che arrivassero lì, però, i giocatori hanno sfornato decine di migliaia di prototipi in continuo miglioramento fino a quando, meno di tre settimane dopo l'inizio, avevano risolto questo particolare puzzle proteico [fonte:Niemeyer]. Non era una cura per l'HIV ma, grazie ad una somiglianza di famiglia retrovirale, Il catalizzatore proteico di MPMV aiuterà i ricercatori a creare farmaci antiretrovirali migliori con cui combattere l'HIV.
Foldit non è privo di limiti, né è una stele di Rosetta per tutte le proteine. Tuttavia, ha permesso a una folla di giocatori di prevedere la struttura di una proteina che aveva sfidato tutti gli approcci tradizionali, e questo da solo giustifica il suo valore come strumento per l'analisi molecolare.
Crowdsourcing e calcolo distribuitoSempre più, gli scienziati stanno sfruttando le collaborazioni di massa per generare idee a basso costo e portare un'ampia varietà di prospettive per affrontare le questioni di ricerca. Foldit è una forma assistita da computer di tale crowdsourcing , ma le folle possono anche aiutare i computer. Per esempio, in calcolo distribuito , le persone offrono volontariamente il tempo di inattività del proprio computer per essere utilizzato nella risoluzione di un problema. Individualmente, questi cicli di lavorazione contano poco, ma combinato, si sommano a un supercomputer virtuale. Reso famoso dalla ricerca del programma SETI@home dell'intelligenza extraterrestre, il calcolo distribuito aiuta anche a modellare le strutture proteiche. Rosetta@casa, sviluppato anche presso l'Università di Washington, è stato installato su centinaia di migliaia di macchine host, fornendo una valida alternativa alle più tradizionali tecniche di analisi delle proteine, come Cristallografia a raggi X e spettroscopia di risonanza magnetica nucleare ( NMR ).