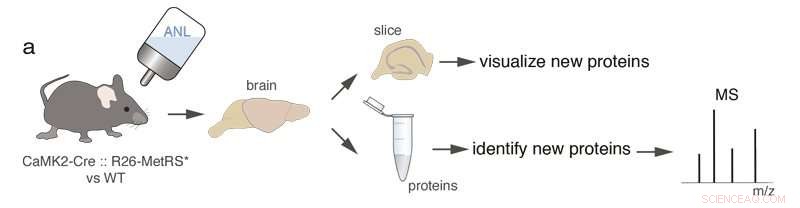
Un topo transgenico che esprime una metionil tRNA sintetasi mutante in cellule specifiche (cellule CamK2a-positive) viene nutrito con un amminoacido non canonico (ANL) che viene incorporato nelle proteine di nuova sintesi solo nelle cellule specifiche. Usando la chimica dei clic, le proteine di nuova sintesi sono contrassegnate per la visualizzazione o l'identificazione. Credito:Brain Research
La complessità degli esseri viventi è guidata, in gran parte, dall'enorme diversità di tipi cellulari. Poiché tutte le cellule di un organismo condividono gli stessi geni, la diversità delle cellule deve provenire dalle particolari proteine che sono espresse. Le cellule del cervello sono generalmente divise in neuroni e glia. All'interno di queste due categorie, però, si trova una grande diversità di tipi di cellule che stiamo appena iniziando a scoprire. La diversità dei tipi di cellule nel cervello e in altri tessuti è stata recentemente ampliata da nuove tecniche, come il sequenziamento dell'RNA, che identificano e misurano gli mRNA presenti in una cellula, il cosiddetto trascrittoma. Sebbene gli mRNA siano lo stampo per le proteine, il trascrittoma è un povero proxy per le proteine che una cellula effettivamente produce, il proteoma. Gli scienziati dell'Istituto Max Planck per la ricerca sul cervello di Francoforte hanno ora sviluppato nuovi metodi per rilevare i cambiamenti in tempo reale nel proteoma.
Basandosi sulla tecnologia precedente, sviluppato dal Lab di Erin Schuman al Max Planck Institute e dai collaboratori David Tirrell del Caltech e Daniela Dieterich dell'Università di Magdeburgo, Beatriz Alvarez-Castelao e colleghi hanno sfruttato un sistema di etichettatura "metabolico" delle proteine. In questo sistema le proteine durante la sintesi sono "marcate" con un amminoacido, che è, in condizioni normali, non presente in queste cellule. Per etichettare esclusivamente le proteine in un particolare tipo di cellula, il team di ricerca ha utilizzato una metionil tRNA sintetasi mutante (MetRS) che riconosce l'aminoacido modificato. Hanno quindi creato una linea di topi in cui MetRS può essere espresso in specifici tipi di cellule. Quando l'amminoacido non canonico viene somministrato ai topi MetRS mutanti attraverso l'acqua da bere, solo le proteine nelle cellule che esprimono la metRS mutante sono etichettate.
Le proteine marcate nelle cellule possono essere visualizzate e riconosciute con anticorpi o possono essere estratte e identificate mediante spettrometria di massa. Alvarez-Castelao:"Abbiamo usato la tecnica per identificare due diversi set di proteine cerebrali, quelli presenti nei neuroni eccitatori dell'ippocampo, una struttura cerebrale importante per la navigazione degli animali, l'apprendimento e la memoria, e neuroni inibitori nel cervelletto, una struttura coinvolta nel comportamento motorio".
Una caratteristica particolarmente sorprendente di questa tecnologia è che è possibile rilevare direttamente i cambiamenti nelle proteine del cervello in risposta a un ambiente modificato. Topi allevati in un ambiente sensoriale arricchito con un labirinto, ruota da corsa, e giocattoli di varia consistenza hanno mostrato cambiamenti significativi nel proteoma nell'ippocampo, in particolare nelle proteine che lavorano nelle sinapsi neuronali. Schuman:"Pensiamo che, combinando questo mouse con altri modelli di mouse "malattia", questo metodo può essere utilizzato per scoprire le proteine in particolari tipi di cellule e come cambiano i proteomi durante lo sviluppo del cervello, apprendimento, memoria e malattia».