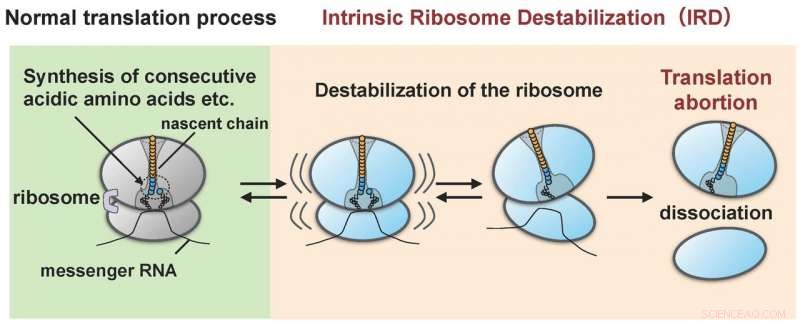
Il ribosoma viene destabilizzato dalla proteina (catena nascente) da essa stessa sintetizzata (IRD) e si divide nelle subunità grandi e piccole. Di conseguenza, la sintesi proteica termina a metà ciclo senza raggiungere il punto finale. Credito:Hidekii Taguchi
Un gruppo di ricerca congiunto del Tokyo Institute of Technology (Tokyo Tech) e della Kyoto Sangyo University ha scoperto che una proteina, durante la sua sintesi, può destabilizzare la struttura del ribosoma e terminare prematuramente la propria sintesi, e ha scoperto che questo fenomeno viene utilizzato per adattare la cellula al suo ambiente.
Sfondo
Le proteine sono molecole funzionali, che supportano la vita e sono strutture tridimensionali di catene polipeptidiche, catene di amminoacidi legati. La configurazione degli amminoacidi su questa catena (la sequenza) è scritta nella sequenza del DNA. Le catene polipeptidiche si formano dove il ribosoma, il macchinario per la sintesi proteica, risiede su una copia della sequenza del DNA (RNA messaggero). Inizia a polimerizzare gli amminoacidi uno ad uno selezionando tra 20 tipi di amminoacidi da aggiungere al legame secondo il codice genetico. Si completa nel punto finale dove la catena si stacca dal ribosoma.
Il processo di sintesi di una proteina in un ribosoma è chiamato "traduzione, " e tutte le proteine di tutti gli organismi viventi, compresi gli umani, sono prodotti attraverso la traduzione. Si era pensato che, nel processo di collegamento degli amminoacidi al ribosoma, la catena polipeptidica neoformata (catena nascente) è stata sintetizzata continuamente.
In studi recenti di questo gruppo di ricerca e di altri, è diventato evidente che la fluttuazione della velocità si verifica in modo significativo nella traduzione, che alcune catene nascenti, a seconda della sequenza amminoacidica, agire sul ribosoma che lo sta formando per rallentare il tempo di traslazione.
Panoramica dei risultati della ricerca
Il gruppo di ricerca guidato da Hideki Taguchi alla Tokyo Tech e Koreaki Ito alla Kyoto Sangyo University ha scoperto che, quando un sistema di traduzione privo di cellule ricostituito di E. coli è stato fatto tradurre proteine aventi sequenze di una decina di amminoacidi acidi (aspartico e glutammico) o sequenze in cui sono collegati alternativamente un amminoacido acido e un amminoacido chiamato prolina, la traduzione si interrompe a metà quando tale sequenza è stata tradotta. Questo evento abortivo si verifica quando i ribosomi che sintetizzano la sequenza amminoacidica si destabilizzano a causa dell'azione della catena nascente (i ricercatori designano questo "IRD":"destabilizzazione intrinseca del ribosoma"). Risulta nella scissione del ribosoma nelle subunità grandi e piccole (Figura 1). Il ribosoma, con il suo ruolo di sintetizzare diverse migliaia o decine di migliaia di tipi di proteine all'interno della cellula, si pensava fosse in grado di collegare qualsiasi combinazione di sequenze di amminoacidi. Però, la scoperta dell'IRD, in cui il ribosoma viene destabilizzato durante la traduzione da catene nascenti esso stesso si sta sintetizzando, indica che la traduzione è qualcosa che progredisce con il potenziale rischio di essere interrotta. Le proteine che includono sequenze di amminoacidi che attivano l'IRD non sono in grado di completare la loro sintesi.
A prima vista, questo fenomeno di IRD può sembrare un difetto del ribosoma, ma cosa indica per gli organismi viventi?
I ricercatori avevano scoperto che gli organismi viventi possiedono anche un meccanismo per contrastare l'IRD. Questa scoperta li ha portati a utilizzare un mutante di E. coli privo di questo meccanismo, con ribosomi quindi leggermente destabilizzati e soggetti a IRD, e analizzare tutte le proteine all'interno di una cellula (il proteoma). Di conseguenza, la squadra ha scoperto che, nel mutante, le quantità cellulari di un certo numero di proteine variavano rispetto al ceppo selvatico. In particolare, MgtA, una proteina di membrana che trasporta lo ione magnesio nelle cellule, è risultato essere espresso più di dieci volte di più. interessante, il gene chiamato mgtL e che regola l'espressione di MgtA possedeva una sequenza IRD. Il risultato dell'analisi ha mostrato che E. coli regola l'espressione di MgtA, utilizzando uno speciale meccanismo caratterizzato dal fenomeno IRD dovuto alla sequenza di mgtL, in risposta ai cambiamenti nelle concentrazioni di magnesio nella cellula.
Gli ioni di magnesio sono necessari per molti processi vitali all'interno della cellula, ed è necessario per la traduzione, in particolare, per stabilizzare il ribosoma. Così, i risultati di questa ricerca suggeriscono che E. coli ha acquisito un meccanismo per mantenere la concentrazione di ioni magnesio cellulare utilizzando la capacità IRD di mgtL di esprimere un'abbondanza di MgtA quando il suo ambiente di crescita diventa povero di magnesio. In altre parole, un organismo vivente ha un meccanismo per monitorare i cambiamenti nell'ambiente intracellulare sfruttando il fenomeno dell'IRD (Figura 2).
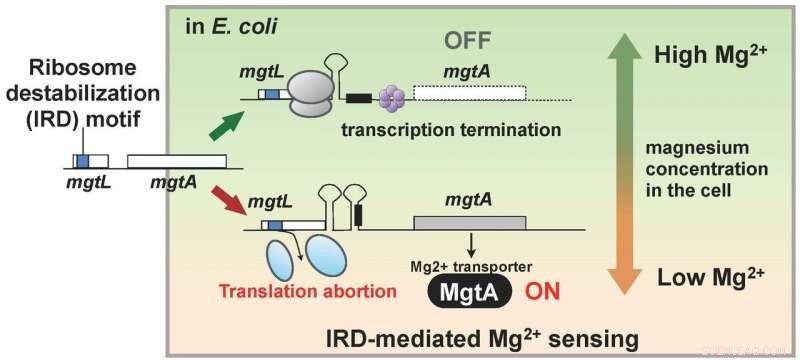
Il gene, mgtL, il controllo dell'espressione della proteina di membrana MgtA che trasporta il magnesio nelle cellule ha una sequenza destabilizzante dei ribosomi, e la traduzione viene interrotta quando la concentrazione di magnesio all'interno della cellula è bassa. Esiste un meccanismo che attiva la sintesi di MgtA quando la traduzione di mgtL viene interrotta, e si pensa che l'abbondante espressione di MgtA che ne risulta aumenti la concentrazione di ioni magnesio all'interno della cellula. Credito:Hideki Taguchi
Sviluppi futuri
Questo studio ha rivelato che, oltre a contenere informazioni che determinano le strutture tridimensionali delle proteine, sequenze di amminoacidi hanno anche la capacità di influenzare la stabilità del macchinario che lo sintetizza e di interrompere la traduzione a metà ciclo, infine. Questo favorisce la nostra comprensione del fondamento dei processi vitali, che sono supportati dall'espressione regolata dell'informazione genetica. Presto, la cessazione prematura della traduzione non era stata considerata fino ad oggi un processo vitale nelle scienze della vita. Taguchi e i membri del team credono che la loro realizzazione possa essere ampliata, in concomitanza con l'attuale prosperità delle scienze della vita, a varie applicazioni come la produzione di nuove proteine utili o lo sviluppo di biofarmaci.