Il percorso appena scoperto coinvolge due geni che facilitano il legame di L-Glu a UDP-MurNAc-L-Ala e quindi epimerizzare (isomerizzare) L-Glu a D-Glu. Credito:Dairi T. et al., Giornale dell'American Chemical Society, 15 marzo, 2017
Gli scienziati hanno scoperto un percorso biosintetico unico per il D-glutammato, un importante componente della parete cellulare nei batteri patogeni, che potrebbe portare allo sviluppo di medicinali e prodotti chimici per l'agricoltura.
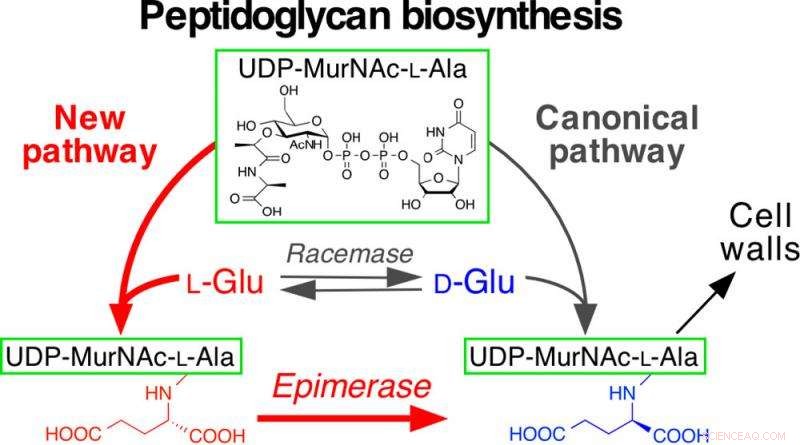
Le pareti cellulari (peptideglicano) sono essenziali per i microrganismi per mantenere l'integrità cellulare. Il team guidato dal professor Toru Dairi, dalla Graduate School of Engineering dell'Università di Hokkaido, focalizzato sulla biosintesi del D-Glutammato (Glu), che è un componente delle pareti cellulari. Generalmente, D-Glu è fornito da un enzima chiamato Glu racemasi. Ma il team ha scoperto attraverso la sua genomica comparativa di vari microrganismi che Xanthomonas oryzae, un batterio patogeno che fa diventare bianche le foglie delle piante di riso prima di uccidere la pianta, non ha i geni Glu racemasi.
Per capire cosa sta compensando il pezzo mancante, i ricercatori hanno condotto esperimenti di clonazione in cui i geni di X. oryzae sono stati introdotti in mutanti di Escherichia coli che non possono produrre D-Glu. Il team ha scoperto che i due geni hanno creato un percorso che aggirava il difetto, consentendo la produzione di D-Glu. Generalmente, D-Glu si lega con un complesso molecolare chiamato UDP-MurNAc-L-Ala, ma in questo microrganismo patogeno, L-Glu si è legato al complesso mediato da uno dei due geni identificati. L'altro gene quindi epimerizza (isomerizza) L-Glu in D-Glu.
"Come X.oryzae, altri batteri patogeni come Stenotrophomonas che innescano infezioni in ospedale potrebbero anche prendere il percorso appena scoperto. Ci auguriamo che il percorso fornisca nuovi obiettivi per lo sviluppo di farmaci e prodotti chimici agricoli contro i batteri, "dice Toru Dairi.