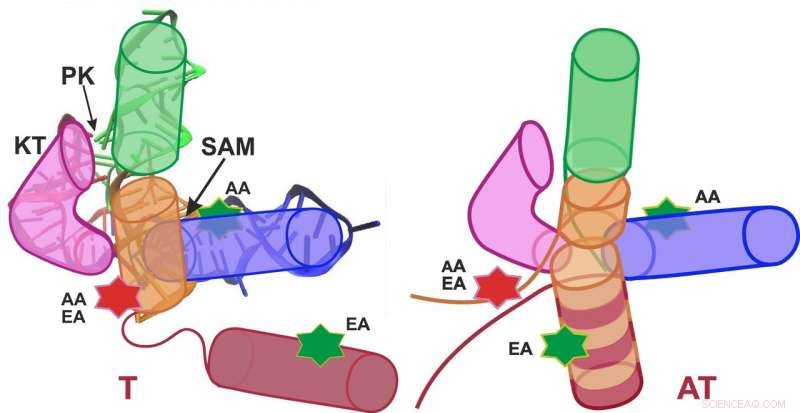
Rappresentazione schematica del riboswitch SAM-I nella conformazione terminatore (spegnimento; sinistra) e anti-terminatore (accensione; destra). Credito:Grafica:APH/KIT
Molti batteri hanno elementi di controllo molecolare attraverso i quali possono attivare e disattivare i geni. Questi riboswitch aprono anche nuove opzioni nello sviluppo di antibiotici o nel rilevamento e nella decomposizione di tossine ambientali. Ricercatori del Karlsruhe Institute of Technology (KIT), Università di Heidelberg, e Freie Universität Berlin hanno ora utilizzato la microscopia ottica leggera di singole molecole per studiare in modo fondamentale il modo in cui funzionano i riboswitch. Questo è riportato in Natura chimica biologia .
I riboswitch si trovano sull'acido ribonucleico messaggero (mRNA) che trasporta le informazioni genetiche al sito di biosintesi proteica. Un riboswitch è costituito da un sensore che misura la concentrazione di una piccola molecola metabolica e da un effettore che controlla l'espressione genica e, quindi, sintesi di una proteina. Poiché i riboswitch esistono in molti patogeni batterici, rappresentano obiettivi importanti nello sviluppo di nuovi antibiotici. Altre applicazioni sono possibili in biologia sintetica. Per esempio, i batteri possono essere geneticamente modificati con riboswitch per rilevare e decomporre le tossine ambientali a basso peso molecolare, come gli erbicidi. Però, è richiesta una conoscenza fondamentale dei processi alla base della funzione dei riboswitch. Il lavoro presentato in Natura chimica biologia è un contributo essenziale in questo senso.
I gruppi di ricerca del professor Gerd Ulrich Nienhaus del KIT e del professor Andres Jäschke dell'Università di Heidelberg hanno studiato il riboswitch S-adenosil-L-metionina (SAM)-I. "L'attaccamento della molecola SAM a questo riboswitch provoca la conformazione, questa è la disposizione spaziale degli atomi, per passare dalla struttura anti-terminatore (AT) alla struttura terminatore (T), " Nienhaus spiega. "Di conseguenza, l'espressione genica è disattivata."
Primo, gli scienziati di Heidelberg hanno sintetizzato i riboswitch SAM-I e li hanno specificamente contrassegnati con due coloranti fluorescenti ciascuno in punti diversi. I ricercatori di KIT hanno quindi studiato queste molecole di RNA ad alta risoluzione spaziale e temporale utilizzando microscopi ottici altamente sensibili che misurano l'emissione fluorescente di molecole a colorante singolo. Mediante esperimenti di trasferimento di energia per risonanza (FRET) di Förster, le dinamiche di conformazione sono state determinate direttamente. Per questo scopo, la radiazione laser viene utilizzata per fare in modo che un colorante verde emetta luce. Se nelle vicinanze si trova un colorante rosso, può assumere l'energia di eccitazione del colorante verde ed emettere luce stessa.
La probabilità di trasferimento di energia dipende fortemente dalla distanza dei coloranti l'uno dall'altro. I cambiamenti strutturali di una molecola a cui i coloranti sono specificamente attaccati possono essere osservati direttamente tramite l'emissione del colorante rosso. L'emissione di luce è estremamente debole, che richiedono metodi di analisi dei dati complessi basati sulla modellazione di Markov nascosta. La professoressa Bettina Keller dell'Istituto di chimica e biochimica della Freie Universität di Berlino ha sviluppato i metodi appositamente per questo tipo di esperimento al fine di distinguere i segnali di emissione di luce dipendenti dal tempo dal rumore.
Nella loro analisi, i ricercatori hanno distinto due conformazioni (T e AT) del riboswitch SAM-I, e un totale di quattro conformazioni (T1, T2, AT1, e AT2). Sorprendentemente, il riboswitch non è passato completamente tra le strutture T e AT in presenza e assenza di SAM, come ci si aspettava, ma fluttuava in modo permanente tra tutti gli stati:solo le ponderazioni venivano spostate. Un risultato importante per la funzione biologica era che le fluttuazioni della struttura osservate con un SAM collegato erano molto più veloci che senza SAM. Poiché la sequenza riboswitch sull'RNA messaggero si trova direttamente di fronte al gene da controllare, la molecola di RNA deve formare una struttura a T (spegnimento) il più rapidamente possibile dopo la sintesi in presenza di SAM per impedire la successiva trascrizione del gene da controllare. L'accelerazione delle fluttuazioni della struttura mediante l'attacco SAM garantisce quindi una formazione sufficientemente rapida di una struttura a T. "Di conseguenza, la dinamica del riboswitch SAM-I gioca un ruolo importante per la sua funzione, " dice Nienhaus. "Queste intuizioni dettagliate sul funzionamento di una biomolecola sono i risultati di un approccio interdisciplinare della fisica, biotecnologia, e chimica teorica."