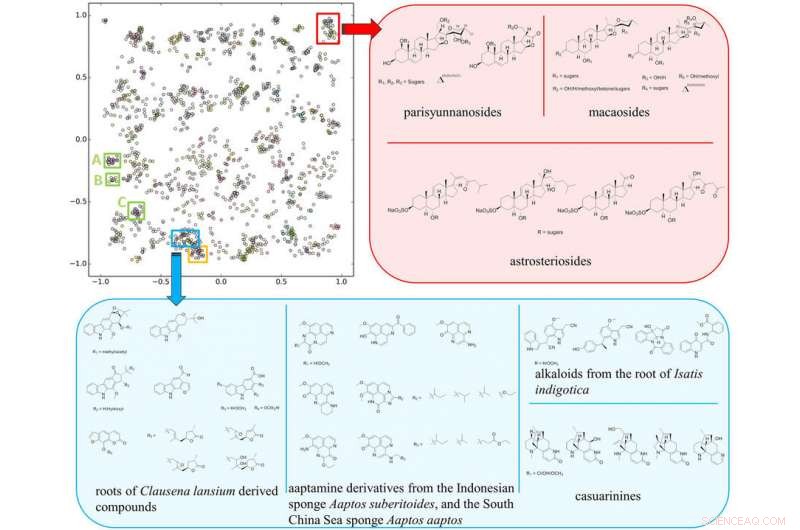
La mappa del cluster SMART basata sul risultato della formazione di 2, 054 spettri HSQC su 83, 000 iterazioni, con riquadri che rappresentano diverse classi di composti discusse nel testo. Credito:Università della California - San Diego
Un team interdisciplinare di ricercatori dell'Università della California a San Diego ha sviluppato un metodo per identificare le strutture molecolari dei prodotti naturali che è significativamente più veloce e preciso rispetto ai metodi esistenti. Il metodo funziona come il riconoscimento facciale per le strutture molecolari:utilizza un dato spettrale unico per ogni molecola e quindi lo esegue attraverso una rete neurale di apprendimento profondo per posizionare la molecola sconosciuta in un cluster di molecole con strutture simili.
Il nuovo sistema si chiama "SMART, " che sta per Small Molecule Accurate Recognition Technology, e ha il potenziale per accelerare di dieci volte il processo di identificazione della struttura molecolare. Questo sviluppo potrebbe rappresentare un cambio di paradigma nell'analisi chimica, campi farmaceutici e di scoperta di farmaci poiché il 70% di tutti i farmaci approvati dalla Food and Drug Administration (FDA) si basa su prodotti naturali come i microrganismi del suolo, piante terrestri e, sempre più, forme di vita marina come le alghe.
"La struttura di una molecola è l'informazione abilitante, " ha detto Bill Gerwick, professore di oceanografia e scienze farmaceutiche presso la Scripps Institution of Oceanography della UC San Diego. "Devi avere la struttura per qualsiasi approvazione della FDA. Se vuoi avere la proprietà intellettuale, devi brevettare quella struttura. Se vuoi fare degli analoghi di quella molecola, devi sapere qual è la molecola di partenza. È un'informazione fondamentale".
Chen Zhang, un dottorato di ricerca in nanoingegneria. studente alla UC San Diego che collabora con Gerwick e primo autore dell'articolo pubblicato in Rapporti scientifici sulla natura , ha detto che determinare la struttura di una molecola può essere un collo di bottiglia nel processo di ricerca del prodotto naturale, impiegando agli esperti mesi e persino anni per determinare con precisione la struttura corretta e completa. Mentre ogni molecola e la sua sequenza temporale di identificazione sono diverse, l'approccio SMART offre ai ricercatori un primo indizio su quale famiglia rientri in una nuova molecola, riducendo drasticamente il tempo necessario per caratterizzare un nuovo prodotto naturale.
"Il modo in cui siamo stati in grado di accelerare il processo è essenzialmente utilizzando un software di riconoscimento facciale per esaminare le informazioni chiave che otteniamo sulle molecole, " ha detto Gerwick. L'informazione chiave che il team usa è chiamata risonanza magnetica nucleare a coerenza quantistica singolare eteronucleare, o HSQC NMR, spettro. Produce una mappa topologica di punti che rivelano quali protoni nella molecola sono attaccati direttamente a quali atomi di carbonio, una disposizione unica per ogni molecola.
Zhang e Gerwick hanno collaborato con Gary Cottrell, un professore di informatica e ingegneria presso la UC San Diego Jacobs School of Engineering, sviluppare un sistema di deep learning addestrato con migliaia di spettri HSQC estratti da ricerche precedenti. Questa rete neurale convoluzionale prende un'immagine 2-D dello spettro HSQC NMR di una molecola sconosciuta e la mappa in uno spazio 10-dimensionale raggruppato vicino a molecole simili, rendendo più facile per i ricercatori chiarire la struttura di una molecola sconosciuta.
"Chen ha adottato questo approccio per ottenere spettri NMR di oltre 4, 000 composti dalla letteratura ritagliando letteralmente le immagini dai PDF dei giornali, " Ha detto Cottrell. "E 'stato uno sforzo impressionante! Comunque, normalmente questi dati non sono sufficienti per addestrare una rete profonda, ma abbiamo usato una tecnologia chiamata rete siamese, in cui ti alleni su coppie di immagini. Questo amplifica il tuo training set di circa il quadrato del numero di composti in una famiglia, ed è ciò che ha reso questo progetto fattibile."
Questa collaborazione è la prima volta che Gerwick ha fatto da mentore a uno studente di ingegneria, e lo scambio di idee si è rivelato fruttuoso.
"È stata un'interazione meravigliosa. L'UC San Diego ha qualcosa di davvero magico, e questa è la profondità della collaborazione che si verifica tra i dipartimenti:è fenomenale, " ha detto Gerwick. "Quando cerchi di prendere premurosamente da un'altra disciplina qualcosa che forse è anche comune in quella disciplina e applicarlo in un modo nuovo e unico nella nostra disciplina, è un'opportunità per avere davvero questo tipo di cambiamento di paradigma. E penso che questa tecnologia, con qualche progresso, potrebbe essere un vero cambiamento di paradigma nel modo in cui facciamo tutti i tipi di chimica e analisi chimiche".