All'inizio, in qualche modo i mattoni genetici di base sono stati tradotti in proteine per condurre alla vita complessa come la conosciamo. Credito:Christ-claude Mowandza-ndinga
Tutti gli esseri viventi usano il codice genetico per "tradurre" le informazioni genetiche basate sul DNA in proteine, che sono le principali molecole di lavoro nelle cellule. Proprio come il complesso processo di traduzione sia sorto nelle prime fasi della vita sulla Terra più di quattro miliardi di anni fa è stato a lungo misterioso, ma due biologi teorici hanno ora compiuto un significativo passo avanti nella risoluzione di questo mistero.
Carlo Carter, dottorato di ricerca, professore di biochimica e biofisica presso la UNC School of Medicine, e Peter Wills, dottorato di ricerca, professore associato di biochimica presso l'Università di Auckland, ha utilizzato metodi statistici avanzati per analizzare come le moderne molecole traslazionali si adattano insieme per svolgere il loro lavoro, collegando brevi sequenze di informazioni genetiche ai mattoni proteici che codificano.
L'analisi degli scienziati pubblicato in Ricerca sugli acidi nucleici , rivela regole precedentemente nascoste con le quali le molecole traslazionali chiave interagiscono oggi. La ricerca suggerisce come gli antenati molto più semplici di queste molecole abbiano iniziato a lavorare insieme agli albori della vita.
"Penso che abbiamo chiarito le regole sottostanti e la storia evolutiva del codice genetico, " Carter ha detto. "Questo era rimasto irrisolto per 60 anni".
Wills ha aggiunto, "Le coppie di modelli molecolari che abbiamo identificato potrebbero essere le prime che la natura abbia mai usato per trasferire informazioni da una forma all'altra negli organismi viventi".
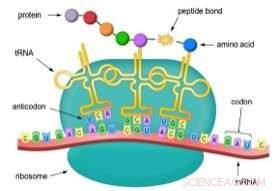
Le scoperte sono incentrate su una molecola a forma di quadrifoglio chiamata RNA di trasferimento (tRNA), un attore chiave nella traduzione. Un tRNA è progettato per trasportare un semplice elemento costitutivo proteico, noto come amminoacido, sulla catena di montaggio della produzione di proteine all'interno di minuscole fabbriche molecolari chiamate ribosomi. Quando una copia o "trascrizione" di un gene chiamato RNA messaggero (mRNA) emerge dal nucleo cellulare ed entra in un ribosoma, è legato ai tRNA che trasportano i loro carichi di amminoacidi.
L'mRNA è essenzialmente una stringa di "lettere" genetiche che spiegano le istruzioni per la produzione di proteine, e ciascun tRNA riconosce una specifica sequenza di tre lettere sull'mRNA. Questa sequenza è chiamata "codone". Quando il tRNA si lega al codone, il ribosoma lega il suo amminoacido all'aminoacido che lo ha preceduto, allungamento del peptide in crescita. Una volta completato, la catena di amminoacidi viene rilasciata come proteina appena nata.
Le proteine nell'uomo e nella maggior parte delle altre forme di vita sono costituite da 20 diversi amminoacidi. Quindi ci sono 20 tipi distinti di molecole di tRNA, ciascuno in grado di legarsi a un particolare amminoacido. La partnership con questi 20 tRNA sono 20 enzimi helper corrispondenti noti come sintetasi (aminoacil-tRNA sintetasi), il cui compito è caricare i tRNA dei loro partner con l'amminoacido corretto.
"Puoi pensare a queste 20 sintetasi e 20 tRNA collettivamente come un computer molecolare che l'evoluzione ha progettato per far accadere la traduzione da gene a proteina, " disse Carter.
Tutti gli esseri viventi usano il codice genetico per "tradurre" le informazioni genetiche basate sul DNA in proteine, che sono le principali molecole di lavoro nelle cellule. Proprio come il complesso processo di traduzione sia sorto nelle prime fasi della vita sulla Terra più di quattro miliardi di anni fa è stato a lungo misterioso, ma due biologi teorici hanno ora compiuto un significativo passo avanti nella risoluzione di questo mistero. Credito:Carter e Wills
I biologi sono stati a lungo incuriositi da questo computer molecolare e dall'enigma di come sia nato miliardi di anni fa. Negli ultimi anni, Carter e Wills hanno fatto di questo puzzle il loro principale obiettivo di ricerca. hanno mostrato, Per esempio, come le 20 sintetasi, che esistono in due classi strutturalmente distinte di 10 sintetasi, probabilmente è nato solo da due più semplici, enzimi ancestrali.
Una divisione in classi simile esiste per gli amminoacidi, e Carter e Wills hanno sostenuto che la stessa divisione di classe deve applicarsi ai tRNA. In altre parole, propongono che all'alba della vita sulla Terra, gli organismi contenevano solo due tipi di tRNA, che avrebbe funzionato con due tipi di sintetasi per eseguire la traduzione da gene a proteina utilizzando solo due diversi tipi di amminoacidi.
L'idea è che nel corso degli eoni questo sistema è diventato sempre più specifico, come ciascuno dei tRNA originali, sintetasi, e gli amminoacidi sono stati aumentati o raffinati da nuove varianti fino a quando non c'erano classi distinte di 10 al posto di ciascuno dei due tRNA originali, sintetasi, e aminoacidi.
Nel loro studio più recente, Carter e Wills hanno esaminato i moderni tRNA alla ricerca di prove di questa antica dualità. Per farlo hanno analizzato la parte superiore della molecola di tRNA, noto come radice accettore, dove si legano le sintetasi partner. La loro analisi ha mostrato che solo tre basi di RNA, o lettere, nella parte superiore della radice accettore portano un codice altrimenti nascosto che specifica le regole che dividono i tRNA in due classi, che corrispondono esattamente alle due classi di sintetasi." Sono semplicemente le combinazioni di queste tre basi che determinano quale classe di sintetasi si lega a ciascun tRNA , " disse Carter.
Lo studio ha trovato casualmente prove per un'altra proposta sui tRNA. Ogni tRNA moderno ha alla sua estremità inferiore un "anticodone" che utilizza per riconoscere e aderire a un codone complementare su un mRNA. L'anticodone è relativamente distante dal sito di legame della sintetasi, ma gli scienziati fin dai primi anni '90 hanno ipotizzato che i tRNA una volta fossero molto più piccoli, combinando le regioni di legame dell'anticodone e della sintetasi in una. L'analisi di Wills e Carter mostra che le regole associate a una delle tre basi che determinano la classe - base numero 2 nella molecola complessiva di tRNA - implicano effettivamente una traccia dell'anticodone in un antico, versione troncata del tRNA.
"Si tratta di una conferma del tutto inaspettata di un'ipotesi che circola da quasi 30 anni, " disse Carter.
Questi risultati rafforzano l'argomento che il sistema di traduzione originale aveva solo due tRNA primitivi, corrispondenti a due sintetasi e due tipi di amminoacidi. Poiché questo sistema si è evoluto per riconoscere e incorporare nuovi amminoacidi, sarebbero emerse nuove combinazioni di basi di tRNA nella regione di legame della sintetasi per stare al passo con la crescente complessità, ma in un modo che lasciava tracce rilevabili della disposizione originale.
"Queste tre basi che definiscono le classi nei tRNA contemporanei sono come un manoscritto medievale i cui testi originali sono stati cancellati e sostituiti da testi più recenti, " disse Carter.
I risultati restringono le possibilità per le origini della codifica genetica. Inoltre, restringono il campo dei futuri esperimenti che gli scienziati potrebbero condurre per ricostruire le prime versioni del sistema traslazionale in laboratorio e forse anche far evolvere questo semplice sistema in più complesso, forme moderne dello stesso sistema di traduzione. Ciò mostrerebbe ulteriormente come la vita si sia evoluta dalla più semplice delle molecole in cellule e organismi complessi.