Credito:Università di Oxford
I ricercatori dell'Università di Oxford hanno costruito una "tramoggia molecolare, "capace di spostare singoli filamenti di DNA attraverso un nanotubo proteico.
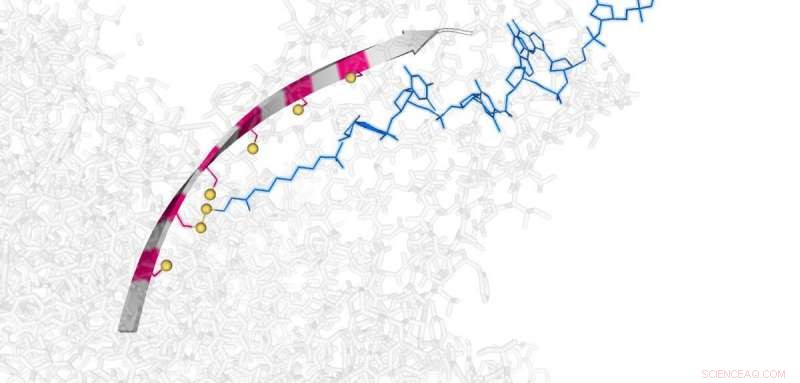
La minuscola tramoggia funziona creando e rompendo in sequenza semplici legami chimici che la collegano a una pista su scala nanometrica. Questo può essere attivato, spento o invertito da un piccolo potenziale elettrico, che alla fine potrebbe renderlo adatto all'uso in dispositivi di sequenziamento del DNA a nanopori.
Professor Hagan Bayley del Dipartimento di Chimica dell'Università di Oxford, che ha condotto la ricerca, ha dichiarato:"Essere in grado di controllare il movimento molecolare è il Santo Graal della costruzione di macchine su scala nanometrica. Essere in grado di elaborare singole molecole di DNA sotto un preciso controllo chimico può fornire un'alternativa all'uso degli enzimi nelle tecnologie di sequenziamento del DNA, migliorando la loro velocità e il numero di molecole che possono essere analizzate in parallelo."
Il Premio Nobel 2016 è stato assegnato in parte per la costruzione di molecole con elementi scorrevoli e rotanti, dimostrando l'importanza di questa tecnologia in molti campi. Il team di Oxford ha notevolmente migliorato questa tecnologia producendo molecole che effettuano passaggi sub-nanometrici che possono essere rilevati uno alla volta e sono soggetti a controllo esterno.
La tramoggia attualmente impiega alcuni secondi per ogni passaggio, e i ricercatori ora cercano di aumentare la velocità della chimica e la lunghezza della pista, che è attualmente limitato a sei punti d'appoggio.
Come funziona la tramoggia
Il movimento saltellante utilizza una chimica molto semplice basata su 3 atomi di zolfo [scambio tiolo/disolfuro], che avviene in acqua a temperatura ambiente. La tramoggia effettua passi sub-nanometrici (0,7 nm), ed è alimentato e controllato da un campo elettrico; la direzione del salto può essere cambiata invertendo il campo elettrico. Tutto questo viene monitorato in tempo reale a livello di singola molecola.
Per il sequenziamento dei nanopori è necessario un movimento a cricchetto, che attualmente si ottiene utilizzando un enzima. Il movimento di salto nel dispositivo appena pubblicato è un cricchetto chimico e questo principio potrebbe essere applicato al sequenziamento del DNA e dell'RNA perché la dimensione del gradino è simile alla distanza internucleotidica nel DNA a filamento singolo.
Il documento completo, "Controllo direzionale di una tramoggia molecolare processiva, " è pubblicato sulla rivista Scienza .