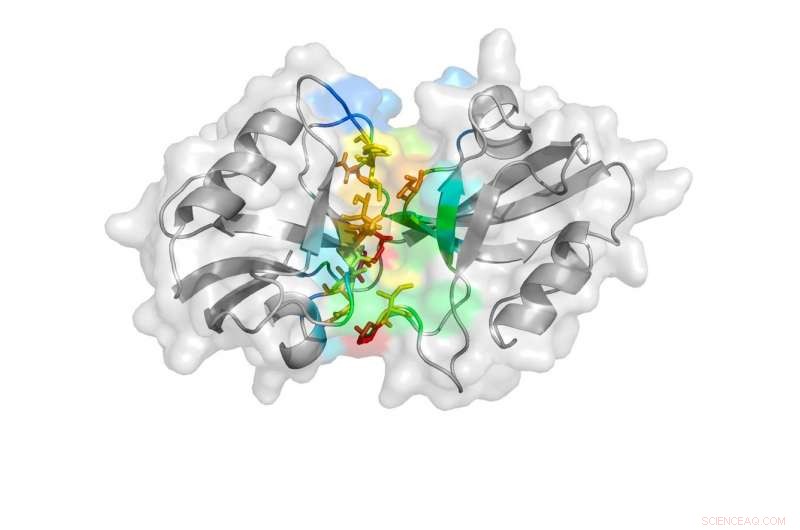
Una rappresentazione a fumetti svelata dallo studio mostra lo stato di chiusura dei domini PDZ. Credito: Comunicazioni sulla natura , doi:10.1038/s41467-018-06133-0
In un'estensione della ricerca pubblicata un mese fa in Metodi della natura , un nuovo approccio ibrido eseguito dai ricercatori del dipartimento di fisica e astronomia della Clemson University e della Stony Brook University ha rivelato una struttura 3-D di un frammento proteico che potrebbe fungere da bersaglio di farmaci nel trattamento dei pazienti con ictus.
La proteina chiamata "proteina di densità postsinaptica di 95 kDa (PSD-95)" è posizionata sui neuroni del cervello che ricevono messaggi chimici - neurotrasmettitori - dai neuroni adiacenti. Reclutando recettori e altre proteine ausiliarie, PSD-95 lavora per mantenere l'integrità delle connessioni neurali nel tempo, facilitando così la comunicazione neurale, apprendimento e memoria.
PSD-95 è composto da cinque parti, o domini, che ciascuno svolge un ruolo diverso nella funzione complessiva della proteina. Due di questi domini, chiamati PDZ-1 e PDZ-2, hanno dimostrato di influenzare i sintomi associati all'ictus ischemico, come paralisi o disturbi del linguaggio.
"Una delle idee che è stata postulata in letteratura è quella di creare un farmaco multivalente che prenda di mira entrambi i domini PDZ perché sono di natura molto simile. Se riesci a impedire ai domini PDZ di legarsi a particolari proteine o enzimi, puoi ridurre gli effetti debilitanti di un ictus, " disse Hugo Sanabria, autore principale dello studio.
La sfida, però, è che è quasi impossibile progettare un inibitore di farmaci senza prima conoscere l'esatta struttura dei domini PDZ di PSD-95. Sarebbe come guidare attraverso il paese senza avere una mappa degli Stati Uniti.
"Le funzioni biologiche delle biomolecole sono determinate dalle loro strutture, quindi abbiamo bisogno di approfondimenti strutturali e dinamici dettagliati di PDZ-1 e -2 per aiutare a comprendere meglio i loro ruoli funzionali e aiutare nella progettazione di nuovi inibitori, " ha detto Feng Ding, Il collega di Sanabria qui alla Clemson.
Esiste una manciata di approcci per rendere la struttura delle biomolecole. Ma nel caso di PSD-95, ogni approccio:spettroscopia NMR, La cristallografia a raggi X e il trasferimento di energia per risonanza di Förster (FRET) hanno fornito un modello strutturale diverso. Il collaboratore dei ricercatori della Stony Brook University, professore associato Mark Bowen nel dipartimento di fisiologia e biofisica, ha stabilito una partnership con Sanabria su questo progetto dopo aver scoperto uno dei modelli strutturali inconsistenti del frammento PSD-95.
Il laboratorio di Sanabria ha affrontato questa discrepanza modellando prima il frammento PSD-95 usando FRET, un approccio che identifica possibili configurazioni di biomolecole. Con questo metodo, Sanabria ha attaccato due molecole fotosensibili, chiamati cromofori, in due diverse posizioni sul frammento PSD-95. Ha poi scoperto la distanza tra i cromofori visualizzando il frammento al microscopio. Questo è stato ripetuto più volte da diversi punti di attacco.
"Per l'aspetto modellistico, FRET ti dà le distanze tra i cromofori, ma non è abbastanza per riempire tutti i vincoli geometrici della molecola, quindi dobbiamo fare affidamento su qualcos'altro, qualche altra metodologia. È qui che entra in gioco il professor Ding, ", ha detto Sanabrio.
Ding dirige un laboratorio di biofisica computazionale presso la Clemson University, dove utilizza un software per misurare l'aspetto delle biomolecole, muoversi e funzionare. Il suo approccio alla modellazione utilizza una simulazione al computer nota come dinamica molecolare discreta (DMD) che mappa il paesaggio di una biomolecola, predire le traiettorie delle proteine mentre si ripiegano e interagiscono con altre molecole. La simulazione successiva può essere riprodotta come un film, aiutando i ricercatori a visualizzare i comportamenti delle proteine nel tempo.
"Se fai simulazioni molecolari tradizionali, di solito si campiona una regione molto piccola dello spazio, in particolare per le molecole più grandi, quindi non avrai una buona panoramica di come apparirà l'intera molecola anche in condizioni fisiologiche, " Sanabria ha detto. "La dinamica molecolare discreta è un modo molto più veloce e meno costoso dal punto di vista computazionale per campionare con precisione e rapidità lo spazio conformazionale delle proteine".
Per farlo, Sanabria ha prima ottenuto una serie di distanze misurando PSD-95 con FRET. In quell'esperimento, Sanabria aveva 10 campioni del frammento PSD-95 che ciascuno stava riproducendo distanze diverse e sono state osservate tre forme o conformazioni comuni di PSD-95. Ancora, senza una simulazione DMD, non c'era modo per i ricercatori di sapere quale distanza corrispondesse a quale conformazione del frammento. Quindi immettono ogni possibile distanza rispetto a ogni possibile forma e lasciano che la simulazione faccia il resto.
"Una volta che abbiamo fatto la prima simulazione, abbiamo visto che c'erano tre stati principali che PDZ-1 e -2 stavano prendendo. Uno ha mostrato un contatto molto stretto tra i due, uno mostrava una serie di contatti intermedi e uno non aveva alcun contatto, "Ding ha detto.
I ricercatori hanno quindi eseguito nuovamente una simulazione DMD senza considerare le distanze FRET per confermare che i tre stati osservati esistono in natura e non sono semplicemente un colpo di fortuna imposto dalle distanze FRET. Hanno ulteriormente sondato le strutture osservando il modo in cui i singoli amminoacidi, che costituiscono i domini PDZ, legame l'uno con l'altro. Da queste analisi, Ding, Bowen e Sanabria sono stati in grado di confermare che i domini PDZ assumono due dei tre stati osservati nella simulazione DMD:quello con qualche contatto e quello senza alcun contatto.
"Ora, abbiamo due potenziali obiettivi per la progettazione di nuovi farmaci che saranno più efficienti di quelli attualmente disponibili, " Sanabria ha detto. "Le prospettive per i pazienti con ictus sono promettenti".
Senza dinamica molecolare discreta, che può catturare i cambiamenti conformazionali che si verificano sulla scala temporale del microsecondo, questi due stati sarebbero stati persi come lo erano negli studi passati.
"La maggior parte delle persone che fanno la modellazione strutturale guidata da FRET sta lavorando con una molecola rigida, come il DNA. Se hai una molecola rigida, è facile da modellare:hai un solo stato da catturare. Puoi assegnare le distanze FRET e non c'è davvero nessun problema, — disse Sanabria. — In questo caso, abbiamo superato questo approccio in molti modi."
Negli studi futuri, il team sta cercando di analizzare il potenziale per il frammento PSD-95 di auto-inibirsi in base alla struttura del frammento.