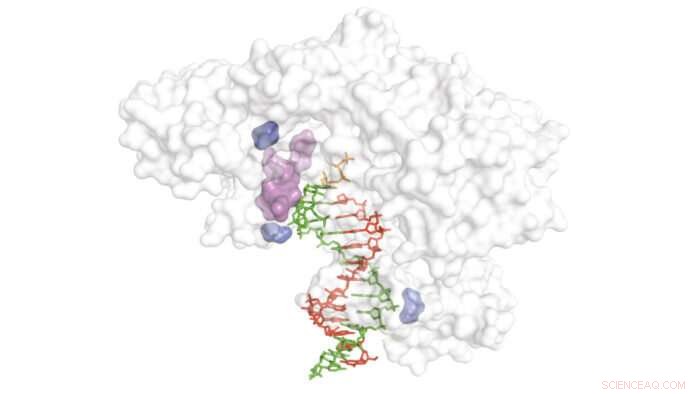
Diagramma che mostra la struttura della trascrittasi inversa (bianco) con il filamento di cDNA nascente (rosso), Modello XNA (verde) e regioni critiche per la trascrizione inversa dei modelli XNA (blu e viola). Credito:MRC Laboratorio di Biologia Molecolare
Oltre ad essere richiesto per alcune delle tecniche fondamentali della biologia molecolare, gli enzimi della trascrittasi inversa (RT) hanno svolto un ruolo chiave nella genetica sintetica consentendo la sintesi, replica, ed evoluzione degli acidi xeno-nucleici (XNA). Però, per la maggior parte delle sostanze chimiche XNA, non sono disponibili enzimi RT o gli enzimi esistenti hanno una bassa attività. Il gruppo di Philipp Holliger, nella divisione PNAC di LMB, hanno sviluppato un nuovo metodo di evoluzione diretta per migliorare l'attività RT per qualsiasi chimica degli acidi nucleici e hanno scoperto un nuovo gruppo di enzimi RT ottimali.
Gli XNA sono polimeri genetici come il DNA o l'RNA, ma con anelli di zucchero alterati, basi, o spine dorsali. Nonostante queste diverse chimiche, sono ancora in grado di immagazzinare e trasmettere informazioni genetiche e possono svolgere funzioni enzimatiche molto simili agli enzimi RNA, noti anche come ribozimi. Possono anche funzionare come aptameri e legarsi a proteine con elevata specificità e affinità, proprio come fanno gli anticorpi. Queste funzioni e le varie proprietà risultanti dalle loro diverse sostanze chimiche significano che gli XNA potrebbero avere un'ampia gamma di applicazioni in biotecnologia e medicina. Però, la generazione di aptameri XNA e XNAzymes è stata limitata dalla mancanza di enzimi RT ad alta fedeltà.
Che cos'è la trascrizione inversa?
Il primo passo del cosiddetto dogma centrale della biologia molecolare è la trascrizione del DNA per produrre RNA. L'RNA può anche essere retrotrascritto per produrre DNA e gli enzimi RT sintetici consentono l'accesso agli XNA.
Consentendo agli scienziati di convertire l'RNA in DNA, Gli enzimi RT consentono agli scienziati di studiare più facilmente quali geni vengono trascritti all'interno delle cellule, e quindi quali geni sono "on, " attraverso tecniche di base come RT-PCR e RNAseq. Accanto alle applicazioni nella ricerca, questa capacità viene utilizzata anche per esami medici, come testare la presenza di RNA virale, anche nei test COVID-19.
Per affrontare la mancanza di enzimi RT ad alta fedeltà, Gillian Houlihan e altri del gruppo di Philipp hanno sviluppato una nuova tecnica di evoluzione diretta che ha portato alla scoperta di un nuovo gruppo di enzimi RT ottimali in grado di decodificare le informazioni genetiche in modo più accurato ed efficiente. È importante sottolineare che questo nuovo metodo è compatibile con qualsiasi chimica dell'acido nucleico e la loro scoperta include nuovi enzimi RT per le chimiche XNA per le quali non esisteva alcun enzima RT in precedenza. Tra i nuovi enzimi RT ci sono i primi enzimi in grado di correggere attivamente le bozze durante la trascrizione inversa di XNA, migliorare la precisione.
Gli enzimi RNA RT ad alta fedeltà avranno applicazioni immediate nella ricerca e nella biotecnologia in quanto forniranno una maggiore precisione di sequenziamento durante l'analisi di RNA cellulari o virali. L'attività migliorata di XNA RT probabilmente aiuterà lo sviluppo di nuovi aptameri XNA che potrebbero essere utili nella diagnostica e nella terapia per un'ampia gamma di malattie. Come esempio specifico, questo lavoro include il primo enzima RT per la chimica XNA utilizzato nel farmaco oligo antisenso nusinersen che ha l'approvazione della FDA e dell'EMA per il trattamento dell'atrofia muscolare spinale. La scoperta di questo enzima RT apre la possibilità di quantificare il livello e l'emivita di questo farmaco all'interno dei pazienti, che potrebbe aiutare il trattamento.