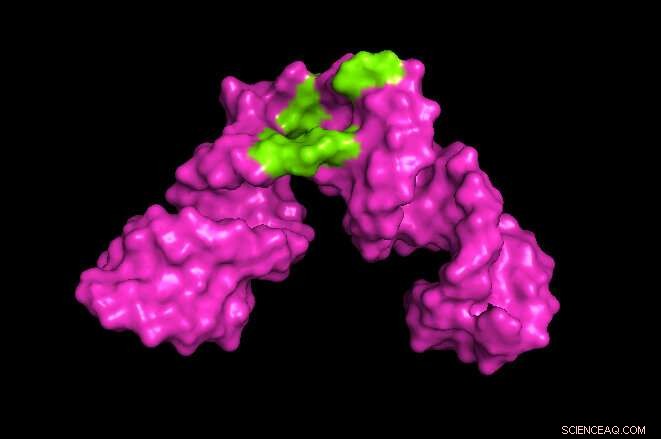
I ricercatori dell'Università del Maryland hanno sviluppato un metodo per ampliare l'ambito della spettroscopia di risonanza magnetica nucleare (NMR). Nell'esempio sopra, i ricercatori sono stati in grado di creare un'immagine 3D che rivela il sito su un pezzo di RNA dell'epatite a cui potrebbero legarsi piccole molecole come un farmaco (mostrato in verde). Credito:Kwaku Dayie/Università del Maryland
Gli scienziati dell'Università del Maryland hanno sviluppato un metodo per determinare le strutture di grandi molecole di RNA ad alta risoluzione. Il metodo supera una sfida che ha limitato l'analisi 3D e l'imaging dell'RNA solo a piccole molecole e pezzi di RNA negli ultimi 50 anni.
Il nuovo metodo, che amplia la portata della spettroscopia di risonanza magnetica nucleare (NMR), consentirà ai ricercatori di comprendere la forma e la struttura delle molecole di RNA e di apprendere come interagiscono con altre molecole. Le intuizioni fornite da questa tecnologia potrebbero portare a trattamenti terapeutici mirati a RNA per la malattia. Il documento di ricerca su questo lavoro è stato pubblicato sulla rivista Progressi scientifici il 7 ottobre, 2020.
"Il campo della spettroscopia di risonanza magnetica nucleare è stato bloccato guardando cose che sono piccole, diciamo 35 blocchi di RNA o nucleotidi. Ma la maggior parte delle cose interessanti che sono biologicamente e clinicamente rilevanti sono molto più grandi, 100 nucleotidi o più, " disse Kwaku Dayie, un professore di chimica e biochimica all'UMD e autore senior dell'articolo. "Così, essere in grado di abbattere l'inceppamento del registro e guardare le cose che sono grandi è molto eccitante. Ci permetterà di sbirciare in queste molecole e vedere cosa sta succedendo in un modo che non siamo stati in grado di fare prima".
Nella spettroscopia NMR, gli scienziati dirigono le onde radio su una molecola, eccitando gli atomi e "illuminando" la molecola. Misurando i cambiamenti nel campo magnetico attorno agli atomi eccitati, la risonanza magnetica nucleare, gli scienziati possono ricostruire caratteristiche come la forma, struttura e movimento della molecola. I dati che questo produce possono quindi essere utilizzati per generare immagini, proprio come le immagini MRI viste in medicina.
ordinariamente, I segnali NMR dai molti atomi in una molecola biologica come l'RNA si sovrappongono tra loro, rendendo l'analisi molto difficile. Però, negli anni '70, gli scienziati hanno imparato a ingegnerizzare biochimicamente le molecole di RNA per lavorare meglio con l'NMR sostituendo gli atomi di idrogeno con atomi di fluoro magneticamente attivi. In molecole relativamente piccole di RNA costituite da 35 o meno nucleotidi, gli atomi di fluoro si illuminano facilmente quando vengono colpiti dalle onde radio e rimangono eccitati abbastanza a lungo per l'analisi ad alta risoluzione. Ma man mano che le molecole di RNA diventano più grandi, gli atomi di fluoro si accendono solo brevemente, quindi perdere rapidamente il segnale. Ciò ha impedito l'analisi 3D ad alta risoluzione di molecole di RNA più grandi.
Lavori precedenti di altri avevano dimostrato che il fluoro continuava a produrre un segnale forte quando era vicino a un atomo di carbonio contenente sei protoni e sette neutroni (C-13). Così, Dayie e il suo team hanno sviluppato un metodo relativamente semplice per cambiare il C-12 presente in natura nell'RNA (che ha 6 protoni e 6 neutroni) in C-13 e installare un atomo di fluoro (F-19) direttamente accanto ad esso.
Dayie e il suo team hanno dimostrato per la prima volta che il loro metodo potrebbe produrre dati e immagini uguali ai metodi attuali applicandolo a pezzi di RNA dell'HIV contenenti 30 nucleotidi, che era stato precedentemente ripreso. Hanno quindi applicato il loro metodo a pezzi di RNA dell'epatite B contenenti 61 nucleotidi, quasi il doppio della precedente spettroscopia NMR possibile per l'RNA.
Il loro metodo ha consentito ai ricercatori di identificare i siti sull'RNA dell'epatite B in cui piccole molecole si legano e interagiscono con l'RNA. Ciò potrebbe essere utile per comprendere l'effetto di potenziali farmaci terapeutici. Il prossimo passo per i ricercatori è analizzare molecole di RNA ancora più grandi.
"Questo lavoro ci consente di espandere ciò che può essere messo a fuoco, " disse Dayie. "I nostri calcoli ci dicono che, in teoria, possiamo guardare cose davvero grandi, come una parte del ribosoma, che è la macchina molecolare che sintetizza le proteine all'interno delle cellule."
Comprendendo la forma e la struttura di una molecola, gli scienziati possono comprendere meglio la sua funzione e come interagisce con il suo ambiente. Cosa c'è di più, questa tecnologia consentirà agli scienziati di vedere la struttura 3D mentre cambia, perché le molecole di RNA in particolare cambiano forma frequentemente. Questa conoscenza è la chiave per sviluppare terapie che colpiscano in modo ristretto le molecole specifiche della malattia senza influenzare le funzioni delle cellule sane.
"La speranza è che se i ricercatori conoscono gli angoli e le fessure in una molecola che è disfunzionale, quindi possono progettare farmaci che riempiono gli angoli e le fessure per metterli fuori uso, " disse Dayie. "E se possiamo seguire queste molecole mentre cambiano forma e struttura, allora la loro risposta a potenziali farmaci sarà un po' più prevedibile, e progettare farmaci efficaci può essere più efficiente".