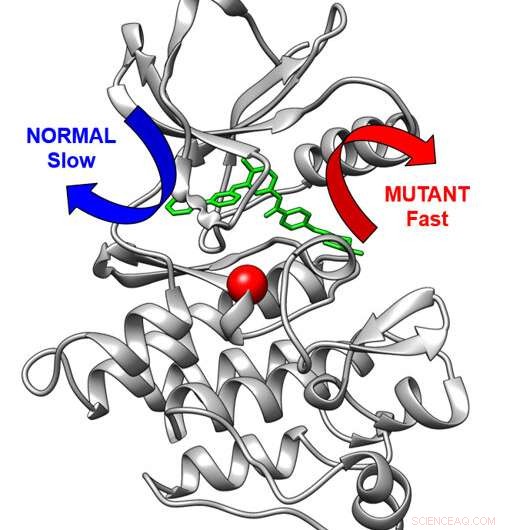
Struttura tridimensionale della chinasi Abl bersaglio del farmaco antitumorale (grigio) legata al farmaco antitumorale imatinib. Normalmente, il farmaco esce rallentando tramite la freccia blu. Una modifica della chinasi (sfera rossa) fa uscire il farmaco attraverso un percorso veloce (freccia rossa). Credito:Aziz M. Rangwala
Capire perché e come si verifica la resistenza alla chemioterapia è un passo importante verso l'ottimizzazione dei trattamenti per il cancro. Un team di scienziati, tra cui Markus Seeliger, Ph.D., dello Stony Brook Cancer Center e della Renaissance School of Medicine della Stony Brook University, crede di aver trovato un nuovo processo attraverso il quale avviene la resistenza ai farmaci. Stanno usando un modello di simulazione al computer che li sta aiutando a capire esattamente come le molecole interagiscono con il farmaco antitumorale Imatinib (noto come Gleevec) nel processo resistente alla chemioterapia. Imatinib tratta la leucemia mieloide cronica (LMC) in modo molto efficace, tuttavia molti pazienti in fase avanzata sperimentano resistenza ai farmaci, il che rende il farmaco minimamente efficace in quella fase.
La ricerca è evidenziata in un articolo pubblicato su Angewandte Chemie e si basa su ricerche precedenti dettagliate nel 2021 in PNAS .
Imatinib inibisce la protein chinasi BCR-Abl, un meccanismo di segnalazione cellulare eccessivamente attivo nella LMC. Nel PNAS studio, i ricercatori hanno dimostrato che le variazioni nel piano di costruzione della chinasi possono rendere più difficile per Imatinib legarsi alla chinasi e anche accelerare il rilascio del farmaco dalla chinasi. Nella Angewandte Chemie documento, il team di ricerca ha adottato la metodologia computazionale, sviluppata dal coautore Pratyush Tiwary dell'Università del Maryland, che ha consentito loro di studiare il rilascio molto lento di Imatinib dalla chinasi.
"Questo metodo in sé è un importante risultato tecnico che estende le capacità computazionali per la ricerca sulla resistenza ai farmaci e, soprattutto, ci ha permesso di prevedere quanto rapidamente proteine sane e mutanti rilascerebbero questo farmaco", afferma Seeliger, professore associato presso il Dipartimento di farmacologia Scienze. "Per la prima volta, abbiamo potuto vedere il rilascio di un farmaco da una proteina in modo così dettagliato e accurato. Inoltre, potremmo dimostrare che la mutazione cambia fondamentalmente all'interno della via di uscita del farmaco dalla proteina.
"Questo è importante poiché la velocità di rilascio del farmaco può essere altrettanto importante per l'effetto terapeutico di un farmaco quanto il legame stretto di un farmaco con la proteina".
Seeliger spiega inoltre che il metodo potrebbe fornire una base per comprendere i meccanismi molecolari alla base della resistenza alla chemioterapia.
Più in generale, le implicazioni di ciò che hanno scoperto sono che se gli scienziati possono capire come i farmaci vengono rilasciati dalle loro proteine, potrebbero essere in grado di progettare farmaci con un rilascio più lento e un maggiore impatto terapeutico. Inoltre, se il rilascio rapido del farmaco può causare resistenza ai farmaci e i medici possono dimostrare che ciò sta accadendo, potrebbero essere in grado di riattivare l'efficacia del farmaco chiedendo al paziente di assumerlo più frequentemente.
Le basi per il test di mutazione tramite il metodo computazionale sono state delineate nel PNAS carta. Seeliger e colleghi hanno testato come imatinib si lega alle mutazioni in pazienti con leucemia mieloide cronica resistente a imatinib. Hanno scoperto che la maggior parte delle mutazioni si lega prontamente a imatinib, quindi questo ha posto la domanda in che modo queste mutazioni causano resistenza nei pazienti? I ricercatori hanno quindi identificato diversi mutanti che legavano prontamente imatinib ma rilasciano il farmaco molto più velocemente.
Dopo aver identificato questi mutanti con un rilascio più rapido del farmaco, il team ha utilizzato la risonanza magnetica nucleare (NMR) e la dinamica molecolare per collegare la proteina alla dissociazione del farmaco, alla base dell'importanza della cinetica di dissociazione del farmaco per l'efficacia del farmaco. Ciò ha consentito loro di identificare un nuovo meccanismo di resistenza a imatinib.
Il lavoro risultante nel paper pubblicato su PNAS ha coinvolto gli sforzi di collaborazione di Seeliger e dei suoi colleghi allo Stony Brook e dei ricercatori del Memorial Sloan Kettering Cancer Center e dell'Università Goethe di Francoforte, in Germania.
La ricerca che ha portato al documento più recente è stata condotta da Tiwary e colleghi dell'Università del Maryland, in collaborazione con Seeliger e scienziati del Broad Institute del MIT e dell'Università di Harvard. + Esplora ulteriormente