I ricercatori del Boston College hanno utilizzato una leggera carica elettrica per modificare con precisione le proteine, un nuovo strumento che può essere utilizzato per sviluppare nuovi strumenti bioterapeutici e di ricerca basati sulle proteine, ha riferito recentemente il team sulla rivista Nature Chemistry .
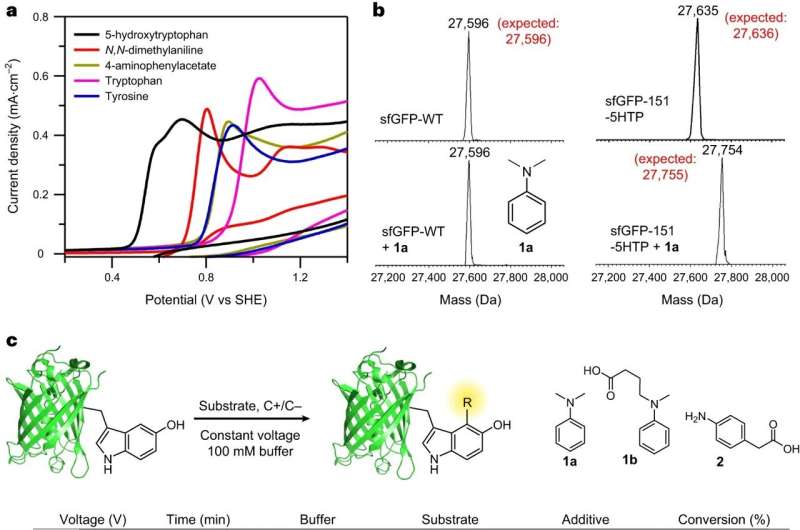
Il team, guidato dai professori di chimica della Columbia Britannica Abhishek Chatterjee e Eranthie Weerapana, ha sviluppato e ottimizzato una nuova reazione elettrochimica di etichettatura delle proteine chiamata "eCLIC" che consente la modifica precisa dei residui di 5-idrossitriptofano (5HTP) incorporati specificatamente nel sito su molte proteine diverse, tra cui l'intero anticorpi terapeutici di lunga durata.
"Abbiamo utilizzato questa strategia per generare molti coniugati proteici sito-specifici, incluso un coniugato anticorpo-farmaco citotossico che entra selettivamente e uccide le cellule tumorali, ma non le cellule non tumorali", ha affermato Chatterjee. "Un vantaggio chiave di eCLIC è che i reagenti necessari per questo metodo sono davvero economici, costando meno di 10 dollari al grammo."
Il successo del team ha segnato la prima volta in cui l'elettrocatalisi è stata utilizzata per ottenere la modificazione delle proteine in modo sito-specifico, come riportato nel loro articolo "Etichettatura elettrochimica di idrossiindoli con chemoselettività per bioconiugazione di proteine sito-specifiche".
Le proteine sono molecole di grandi dimensioni, tipicamente composte da centinaia di monomeri di amminoacidi, ha osservato Chatterjee. La capacità di modificare selettivamente le proteine in siti predefiniti è importante per molte applicazioni. Ad esempio, legando in modo covalente farmaci tossici agli anticorpi è stato possibile somministrarli selettivamente alle cellule tumorali, il che ha portato sia a una migliore efficacia terapeutica che a una ridotta tossicità fuori bersaglio.
Molte applicazioni di ricerca richiedono anche il collegamento di sonde biofisiche su varie proteine. La capacità di definire il sito di modificazione sulle proteine è fondamentale per garantire che importanti funzioni proteiche non vengano danneggiate, ha affermato Chatterjee.
"La sfida nasce dal fatto che tutte le proteine sono costituite da 20 aminoacidi in varie combinazioni", ha affermato. "Identificare una funzionalità modificabile nel sito desiderato, non ripetuta altrove, è generalmente impegnativo, il che rende difficile ottenere la specificità del sito nella modificazione delle proteine."
Per superare queste sfide, il team ha cercato di sviluppare un metodo per incorporare un amminoacido non naturale in qualsiasi sito scelto di una proteina. Il team è riuscito a raggiungere questo obiettivo riprogettando il sistema di traduzione delle cellule per accogliere il nuovo amminoacido 5HTP.
Inoltre, i ricercatori volevano progettare reazioni chimiche che potessero essere utilizzate per modificare selettivamente questo amminoacido non naturale in presenza di tutti gli amminoacidi naturali, ha affermato Chatterjee.
"Se potessimo farlo, potremmo fornire un metodo generale per generare proteine con un 'manico di attacco' incorporato in un sito predefinito. In particolare, eravamo interessati a sviluppare una reazione che utilizzasse l'elettricità per catalizzare la modificazione della proteina reazione invece della catalisi chimica poiché la prima è economica, rispettosa dell'ambiente e delicata sulle proteine delicate."
Chatterjee ha affermato che il team è stato in grado di superare una sfida insolita quando ha provato per la prima volta a modellare la reazione. In genere, i ricercatori iniziano con piccole molecole, in questo caso 5HTP e anilina, per poi passare alle proteine più grandi.
Ma i primi tentativi di reazione tra 5HTP e aniline a livello di piccole molecole sono stati complicati, poiché le molecole 5HTP reagivano preferenzialmente tra loro. Ma quando il 5HTP è stato incorporato in una grande proteina, non è stato più in grado di reagire con un altro 5HTP legato alla proteina e ha invece reagito in modo pulito con un'anilina, ha riferito il team.
"Se fossimo rimasti fedeli alla progressione tradizionale, dal piccolo al grande, non avremmo mai perseguito eCLIC, pensando che fosse troppo complicato", ha affermato Chatterjee. "Invece, abbiamo sviluppato la reazione in modo non tradizionale, direttamente su una proteina, il che ci ha aiutato a capire quanto fosse pulita e selettiva in questo contesto."
Per far avanzare ulteriormente la strategia eCLIC per la modifica su larga scala di importanti target proteici, questa tecnologia è stata concessa in licenza aBrickBio, Inc., di cui Chatterjee è co-fondatore. La ricerca futura si concentrerà sullo sviluppo di prodotti bioterapeutici e reagenti di ricerca a base di proteine modificate specificatamente per il sito di prossima generazione.
Ulteriori informazioni: Conor Loynd et al, Etichettatura elettrochimica di idrossiindoli con chemoselettività per la bioconiugazione di proteine sito-specifiche, Nature Chemistry (2023). DOI:10.1038/s41557-023-01375-y
Fornito dal Boston College