Un gruppo di ricerca del Dipartimento di Chimica dell'Università di Amburgo è riuscito per la prima volta a identificare a livello molecolare il meccanismo dinamico utilizzato dall'enzima RNasi R per degradare la subunità ribosomiale 30S. I risultati dello studio sono stati pubblicati sulla rivista scientifica Nature .
La sintesi proteica è un processo vitale e ad alta intensità energetica nella cellula in cui i ribosomi svolgono un ruolo cruciale. Queste molecole relativamente grandi si trovano in tutti gli organismi viventi e agiscono come "fabbriche di proteine" delle cellule.
Per fare ciò, i ribosomi leggono il progetto di una proteina specifica su una molecola messaggera, l’RNA messaggero (mRNA), e quindi convertono queste informazioni in una nuova proteina. I ribosomi sono costituiti da due subunità. La subunità piccola è responsabile della lettura e del controllo degli errori nell'mRNA, mentre la subunità grande è responsabile della polimerizzazione degli amminoacidi per formare proteine.
Per la sintesi proteica sono necessari una produzione controllata e un turnover regolato dei ribosomi. Sebbene l'assemblaggio dei ribosomi sia stato compreso sempre meglio negli ultimi anni, non sono state ottenute informazioni strutturali sulla degradazione dei ribosomi.
Questo è importante perché in situazioni di stress come la mancanza di cibo, o alla fine del loro ciclo di crescita, le cellule riducono il loro metabolismo per sopravvivere più a lungo. Questo stato è noto come fase stazionaria. Durante questa fase, la sintesi proteica ad alta intensità energetica viene ridotta e alcuni ribosomi vengono degradati per rilasciare l'energia investita in essi per garantire la sopravvivenza cellulare.
Per le loro indagini, i ricercatori hanno studiato il Bacillus subtilis, un batterio del suolo a forma di bastoncino che si trova nell'aria, nella polvere e nell'acqua, nonché nell'intestino dell'uomo e degli animali. "A differenza degli studi precedenti, abbiamo preso cellule che erano ancora in crescita e non nella fase stazionaria. Volevamo sapere quali processi avvengono nel passaggio alla fase stazionaria", afferma il dott. Helge Paternoga del Dipartimento di Chimica dell'Università Universität Hamburg, ultimo autore dello studio.
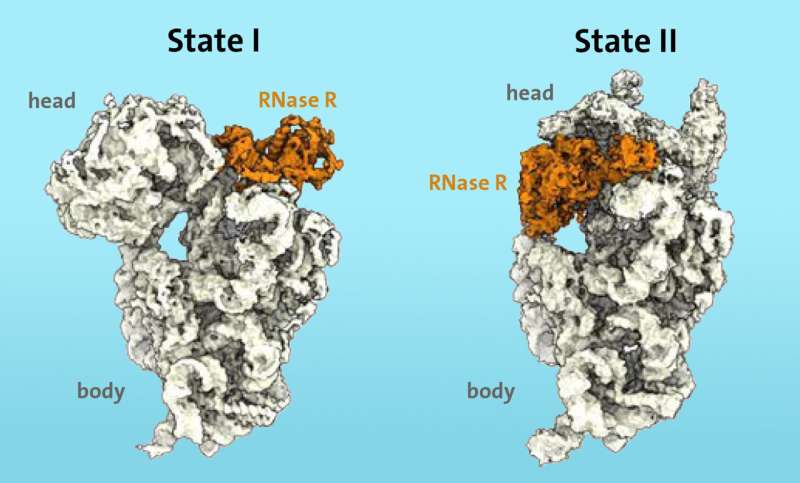
I ricercatori sapevano da lavori precedenti che alcuni enzimi, come la ribonucleasi R (RNasi R), sono coinvolti nel processo di degradazione dei ribosomi in situazioni di stress. Utilizzando la microscopia crioelettronica, sono stati in grado di dimostrare per la prima volta che l'enzima RNasi R si lega alla piccola subunità 30S del ribosoma. La "S" sta per "unità Svedberg" e si riferisce alla massa della subunità ribosomiale.
La RNasi R non taglia arbitrariamente la subunità 30S, ma si attacca ad un'area libera, che i ricercatori chiamano "collo", e poi stacca la "testa", la zona superiore della subunità, in due fasi consecutive. /P>
"Nella prima fase, l'enzima RNasi R incontra un ostacolo al 'collo' e destabilizza la zona del collo, rendendola più flessibile. Nella seconda fase, la 'testa' viene girata, il che rimuove l'ostacolo e consente all'enzima di continuare senza ostacoli il processo di degradazione della subunità 30S," spiega Paternoga.
"I nostri esperimenti di degradazione in vitro indicano che l'interruttore della 'testa' costituisce una barriera cinetica significativa per l'RNasi R. Inoltre, siamo stati in grado di dimostrare che l'enzima da solo è sufficiente per realizzare l'intero processo di degradazione 30S", afferma il Prof. Dr. Daniel Wilson, capo del gruppo di ricerca presso il Dipartimento di Chimica dell'Università di Amburgo e coautore dello studio.
Ulteriori informazioni: Lyudmila Dimitrova-Paternoga et al, Basi strutturali della degradazione della subunità ribosomiale 30S da parte di RNasi R, Natura (2024). DOI:10.1038/s41586-024-07027-6
Fornito dall'Università di Amburgo