I lipidi sono una classe di biomolecole che svolgono un ruolo importante in molti processi cellulari. Le analisi che cercano di caratterizzare tutti i lipidi in un campione, chiamate lipidomica, sono fondamentali per studiare sistemi biologici complessi.
Una sfida importante nella lipidomica è collegare la varietà di strutture dei lipidi con le loro funzioni biologiche. La posizione dei doppi legami all'interno delle catene di acidi grassi è particolarmente importante. Questo perché possono influenzare le proprietà fisiche delle membrane cellulari e modulare le vie di segnalazione cellulare.
Queste informazioni non vengono misurate di routine negli studi lipidomici perché richiedono un complicato apparato sperimentale che produce dati complessi. Pertanto, gli scienziati del Pacific Northwest National Laboratory (PNNL) hanno sviluppato un flusso di lavoro semplificato per determinare le posizioni dei doppi legami. Questo flusso di lavoro utilizza approcci sia di automazione che di machine learning.
Il loro nuovo metodo, LipidOz, semplifica l’analisi dei dati per determinare le posizioni dei doppi legami. Affrontando questa parte fondamentale dell'analisi dei lipidi, LipidOz offre ai ricercatori un metodo più efficiente e accurato per la caratterizzazione dei lipidi. Lo studio è pubblicato sulla rivista Communications Chemistry .
L'identificazione univoca dei lipidi è complicata dalla presenza di parti molecolari che hanno la stessa formula chimica ma diverse configurazioni fisiche. Nello specifico, le differenze in queste parti molecolari includono la lunghezza della catena acilica grassa, la posizione numerata stereospecificamente (sn) e la posizione/stereochimica dei doppi legami.
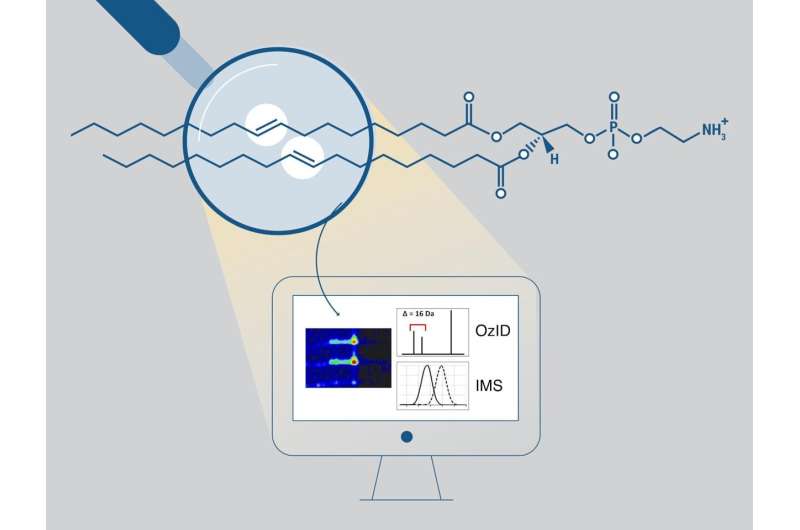
Le analisi convenzionali possono determinare la lunghezza delle catene aciliche grasse, il numero di doppi legami e, in alcuni casi, la posizione sn ma non le posizioni dei doppi legami carbonio-carbonio. Le posizioni di questi doppi legami possono essere determinate con maggiore sicurezza utilizzando una reazione di ossidazione in fase gassosa chiamata dissociazione indotta da ozono (OzID), che produce frammenti caratteristici.
Tuttavia, l'analisi dei dati ottenuti da questa reazione è complessa e ripetitiva e manca il supporto di strumenti software. Lo strumento Python open source, LipidOz, determina e assegna automaticamente le posizioni dei doppi legami dei lipidi utilizzando una combinazione di automazione tradizionale e approcci di deep learning. Una nuova ricerca dimostra questa capacità per miscele lipidiche standard ed estratti lipidici complessi, consentendo l'applicazione pratica di OzID per futuri studi lipidomici.
Ulteriori informazioni: Dylan H. Ross et al, LipidOz consente la delucidazione automatizzata delle posizioni dei doppi legami carbonio-carbonio dei lipidi dai dati della spettrometria di massa di dissociazione indotta dall'ozono, Chimica delle comunicazioni (2023). DOI:10.1038/s42004-023-00867-9
Informazioni sul giornale: Chimica delle comunicazioni
Fornito dal Pacific Northwest National Laboratory