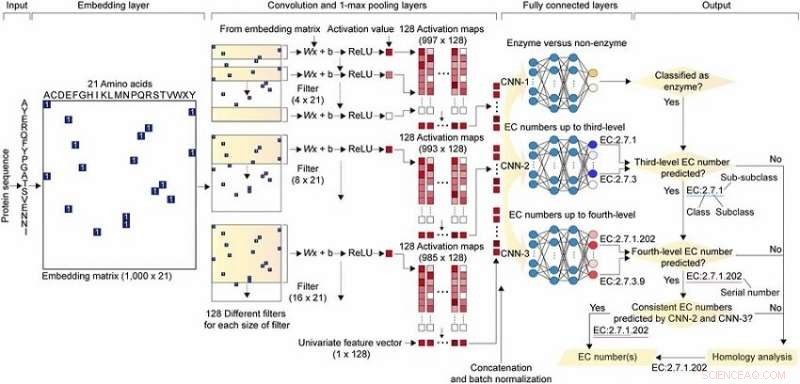
Schema generale di DeepEC. Attestazione:KAIST
Un framework computazionale basato sul deep learning, 'DeepEC, ' consentirà la previsione di alta qualità e ad alto rendimento dei numeri delle commissioni enzimatiche, che è essenziale per la comprensione accurata delle funzioni degli enzimi.
Un team composto dal Dr. Jae Yong Ryu, Professor Hyun UK Kim, e il distinto professor Sang Yup Lee al KAIST hanno riportato il quadro computazionale alimentato dal deep learning che prevede i numeri della commissione enzimatica (EC) con alta precisione in un modo ad alto rendimento.
DeepEC prende una sequenza proteica come input e prevede con precisione i numeri EC come output. Gli enzimi sono proteine che catalizzano reazioni biochimiche e numeri CE, che consistono di quattro numeri di livello (cioè, un, B, C, d) indicare reazioni biochimiche. Così, l'identificazione dei numeri CE è fondamentale per comprendere con precisione le funzioni e il metabolismo degli enzimi.
I numeri EC vengono solitamente assegnati a una sequenza proteica che codifica un enzima durante una procedura di annotazione del genoma. Data l'importanza dei numeri CE, sono stati sviluppati diversi strumenti di previsione del numero CE, ma hanno margini di ulteriore miglioramento per quanto riguarda i tempi di calcolo, precisione, copertura, e la dimensione totale dei file necessari per la previsione del numero CE.
DeepEC utilizza tre reti neurali convoluzionali (CNN) come motore principale per la previsione dei numeri EC, e implementa anche l'analisi dell'omologia per i numeri EC se le tre CNN non producono numeri EC affidabili per una data sequenza proteica. DeepEC è stato sviluppato utilizzando un set di dati gold standard che copre 1, 388, 606 sequenze proteiche e 4, 669 numeri CE.
In particolare, studi di benchmarking di DeepEC e di altri cinque strumenti rappresentativi di previsione dei numeri EC hanno mostrato che DeepEC ha effettuato le previsioni più precise e veloci per i numeri EC. DeepEC richiedeva anche lo spazio su disco più piccolo per l'implementazione, che lo rende un componente software di terze parti ideale.
Per di più, DeepEC è stato il più sensibile nel rilevare la perdita della funzione enzimatica a causa di mutazioni nei domini/sito di legame residuo delle sequenze proteiche; in questa analisi comparativa, tutti i domini o residui del sito di legame sono stati sostituiti con residui di L-alanina per rimuovere la funzione proteica, che è noto come metodo di scansione L-alanina.
Questo studio è stato pubblicato online nel Atti dell'Accademia Nazionale delle Scienze ( PNAS ) il 20 giugno, 2019, intitolato "L'apprendimento approfondito consente una previsione di alta qualità e ad alto rendimento dei numeri delle commissioni enzimatiche".
"DeepEC può essere utilizzato come strumento indipendente e anche come componente software di terze parti in combinazione con altre piattaforme computazionali che esaminano le reazioni metaboliche. DeepEC è disponibile gratuitamente online, ", ha detto il professor Kim.
Il distinto professor Lee ha detto, "Con DeepEC, è diventato possibile elaborare volumi sempre crescenti di dati di sequenza proteica in modo più efficiente e accurato".