
Pepsi-SAXS:nuovo metodo di analisi delle proteine 50 volte più veloce degli analoghi. Credito:MIPT
Pepsi-SAXS è un nuovo, metodo altamente efficiente per il calcolo dei profili di diffusione dei raggi X, che sono necessari per l'analisi della molecola proteica allo stato di soluzione. Il metodo è stato creato da scienziati dell'Université Grenoble Alpes e del MIPT, guidato da Sergei Grudinin. Il team ha testato il loro metodo, e i risultati sono stati pubblicati dalla International Union of Crystallography nella sua rivista Acta Crystallographica Sezione D:Biologia Strutturale .
Le proteine hanno una struttura complessa e dimensioni estremamente ridotte, dell'ordine di alcuni nanometri. Per studiarli, i ricercatori devono trovare metodi insoliti, perché i campioni di proteine vengono distrutti troppo facilmente e le loro proprietà alterate negli esperimenti. La conoscenza delle strutture e dei meccanismi funzionali delle biomolecole consente di sviluppare nuovi farmaci non per tentativi ed errori, tecnicamente chiamati screening ad alto rendimento, ma in modo più mirato.
Una delle tecniche utilizzate per studiare le proteine è l'analisi dei raggi X diffusi da esse. I ricercatori devono utilizzare i raggi X e non la luce ordinaria per ingrandire i singoli atomi con una dimensione caratteristica dell'ordine di 0,1 nanometri. Più piccolo è l'oggetto, minore è la lunghezza d'onda della luce che deve essere utilizzata per osservarla. La luce visibile comprende lunghezze d'onda comprese tra 400 e 700 nanometri. raggi X, d'altra parte, hanno una lunghezza d'onda molto più corta e quindi possono essere usati per esaminare le strutture molecolari.
"Il nuovo metodo ci consente di tracciare curve di dispersione in modo efficiente e preciso, e analizzare la struttura tridimensionale di un campione, " dice la studentessa del MIPT Maria Garkavenko, un coautore del documento. "Tra l'altro, Pepsi-SAXS aumenta l'efficienza della modellazione e l'accuratezza della previsione tridimensionale della struttura delle macromolecole".
Diffusione di raggi X a piccolo angolo, o SAXS, è una tecnica sperimentale che prevede la diffusione di raggi X da un campione e la loro raccolta ad angoli molto piccoli. Di conseguenza, si ottiene un grafico dell'intensità del fascio di raggi X diffusi in funzione dell'angolo di incidenza. Usando questa trama, un campione proteico può essere confrontato con altri campioni nel database sperimentale per determinarne la struttura e le proprietà.
Rispetto ad altre tecniche utilizzate per determinare la struttura del campione, SAXS è molto più semplice ed economico. Richiede solo una preparazione minima del campione, e le proteine non devono essere congelate o cristallizzate. I campioni vengono studiati in soluzione e nel loro stato funzionale. Questo rende i risultati molto più affidabili, perché la preparazione del campione a volte può alterare lo stato e le proprietà di una proteina. Un altro importante vantaggio del metodo è che non è distruttivo, il che significa che il campione sperimentale rimane in gran parte inalterato dai raggi X.
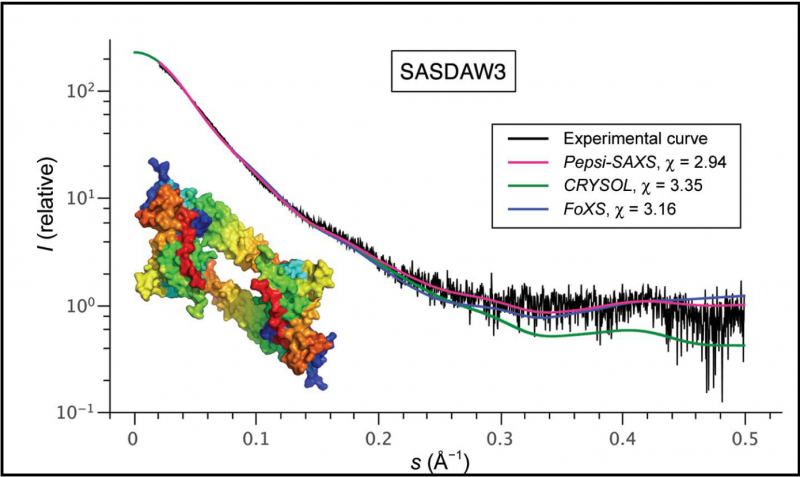
La figura 1 mostra i risultati di una serie di esperimenti, che ha confrontato Pepsi-SAXS con due dei metodi computazionali attualmente utilizzati applicandoli allo stesso campione (SASDAW3) dal database SASBDB. L'intensità media dispersa viene tracciata in funzione dell'angolo di diffusione. L'errore χ² del modello computazionale è il più basso nel caso di Pepsi-SAXS, che risulta da una rappresentazione più precisa del guscio di idratazione. Credito:S. Grudinin, M. Garkavenko e A. Kazennov
Ma fino a poco tempo fa SAXS aveva un grosso inconveniente:il metodo era computazionalmente intensivo, il che significava che non poteva essere utilizzato se il numero di esperimenti era sostanziale. Ci sono volute ore per elaborare i risultati di un solo esperimento. Inizialmente, il numero di calcoli era direttamente proporzionale al quadrato del numero degli atomi nel campione, quest'ultimo numero di solito superiore a mille. Però, negli anni '70, Heinrich Stuhrmann, un ricercatore tedesco, ha avuto un'idea che ha semplificato i calcoli. Propose che lo scattering sui composti molecolari fosse descritto in termini di funzioni di un tipo particolare chiamato armoniche sferiche. Questo approccio si è rivelato un successo. Negli anni, sono stati creati una serie di strumenti di calcolo per l'analisi dei dati SAXS. Importanti contributi al loro sviluppo sono stati dati da ricercatori con un background scientifico sovietico tra cui Dmitri Svergun (che attualmente lavora ad Amburgo), che ha scritto la suite di software ATSAS per l'analisi dei dati SAXS nella ricerca sulle macromolecole biologiche. I ricercatori dello studio qui riportato hanno esaminato diversi metodi computazionali e li hanno confrontati con la propria tecnica.
"Pepsi-SAXS sta per "espansioni polinomiali di strutture e interazioni proteiche" e "diffusione di raggi X a piccolo angolo". È un metodo adattivo per il calcolo rapido e accurato di profili di diffusione di raggi X a piccolo angolo, " spiega il dottorando del MIPT Andrei Kazennov, un coautore del documento. "Pepsi-SAXS può essere adattato alla dimensione di un dato campione e alla risoluzione dei dati sperimentali".
I ricercatori hanno anche creato un modello efficiente del guscio di idratazione, uno strato di molecole d'acqua che circonda le proteine in soluzione, e lo hanno incorporato nel loro software, aumentando la precisione del metodo.
"Il nostro metodo è stato convalidato su un ampio set di dati da BioIsis e SASBDB, le due più grandi banche dati biologiche, "dice Sergei Grudinin, che ha curato la ricerca. "Abbiamo dimostrato che Pepsi-SAXS è da cinque a 50 volte più veloce dei metodi utilizzati in precedenza, vale a dire CRYSOL, FoXS, e la tecnica Zernike tridimensionale implementata nel pacchetto SAStbx. Allo stesso tempo, la precisione è alla pari con loro."
I ricercatori hanno prestato particolare attenzione all'analisi dei risultati ottenuti, che sono stati confrontati con i dati sperimentali.
La ricerca sulle proteine ha un significato fondamentale per la nostra comprensione dei processi di base alla base della vita, così come per lo sviluppo di farmaci, trattamenti, e materiali organici, compresi gli organi artificiali. Il nuovo strumento presentato dagli autori potrebbe significare progressi 50 volte più rapidi in questi settori.