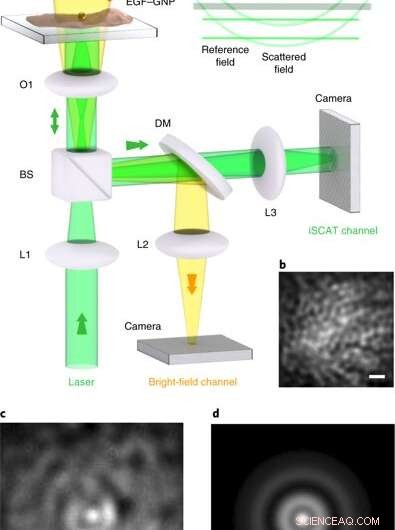
Microscopia iSCAT su cellule vive. un, Disposizione sperimentale del microscopio iSCAT per l'imaging di cellule vive. Le cellule vengono piastrate in un piatto con fondo di vetro sotto mezzo Leibowitz. (a) la micropipetta fornisce le sonde EGF-GNP direttamente sulla coltura cellulare, dove mirano specificamente alla proteina EGFR nella membrana cellulare. Il canale di illuminazione a campo chiaro dall'alto aiuta a ispezionare la coltura ma non è necessario per l'imaging iSCAT. L1–L3, lenti a contatto; O1, ×100 obiettivo; BS, divisore di raggio 90:10; DM, Specchio dicroico a passaggio corto da 590 nm. L'imaging iSCAT è stato eseguito con intensità di illuminazione di 1-8 kW cm-2, che sono noti per essere vitali per HeLa alla lunghezza d'onda di interesse. inserto, fronti d'onda dei campi che contribuiscono al segnale iSCAT. (B), Una sezione della membrana della cellula HeLa prima della marcatura, visualizzato tramite riflessione iSCAT. (C), Immagine iSCAT della membrana cellulare inclusa una sonda EGF-GNP legata. (D), Il PSF estratto da c. Le barre della scala in b–d sono 1 μm. Credito: Fotonica della natura , doi:10.1038/s41566-019-0414-6
Le funzioni cellulari sono dettate dall'intricato movimento delle proteine nelle membrane che si estendono su una scala da nanometri a micrometri, entro un lasso di tempo da microsecondi a minuti. Però, questo ricco parametro dello spazio è inaccessibile utilizzando la microscopia a fluorescenza, sebbene sia alla portata del tracciamento delle particelle a dispersione interferometrica (iSCAT). La nuova tecnica iSCAT è, però, altamente sensibile alle proteine singole e non etichettate, causando così una colorazione di fondo non specifica come una sfida sostanziale durante l'imaging cellulare.
In un recente studio, Richard W. Taylor e colleghi dei dipartimenti interdisciplinari di Fisica e Biologia in Germania hanno sviluppato un nuovo approccio di elaborazione delle immagini per superare questa difficoltà. Hanno usato il metodo per tracciare il recettore del fattore di crescita epidermico transmembrana (EGFR) con precisione su scala nanometrica in tre dimensioni (3-D). La tecnica ha consentito l'imaging da microsecondi a minuti. Gli scienziati hanno fornito esempi di movimento e confinamento su scala nanometrica utilizzando il metodo per visualizzare processi ubiquitari come la diffusione nelle membrane plasmatiche, trasporto in filopodia e movimento rotatorio durante l'endocitosi. I risultati sono ora pubblicati in Fotonica della natura .
Mentre i costanti progressi nella microscopia a fluorescenza hanno permesso agli scienziati di monitorare gli eventi cellulari su scala nanometrica, resta ancora molto da fare con i sistemi di imaging avanzati. Le sfide della microscopia a fluorescenza si sono verificate a causa del tasso di emissione finito di una sorgente fluorescente (molecola di colorante o punto quantico di semiconduttore), dove troppo poche emissioni di fotoni durante un intervallo di tempo molto piccolo hanno impedito un'imaging efficace o prolungato. La difficoltà centrale della microscopia basata sulla dispersione è relativa alla sonda nanoscopica, che compete con il rumore di fondo e un basso rapporto segnale-rumore (SNR); limitando il potenziale dell'imaging a pochi nanometri negli esperimenti di tracciamento ad alta velocità.

Video grezzo di una nanoparticella di fattore di crescita epidermico-oro (EGFR-GNP) che si diffonde su una membrana cellulare HeLa. Credito: Fotonica della natura , doi:10.1038/s41566-019-0414-6
Nel presente lavoro, Taylor et al. ha utilizzato la microscopia a dispersione interferometrica (iSCAT) per tracciare le proteine nelle membrane cellulari vive. Il metodo potrebbe visualizzare le interazioni sonda-cella per comprendere le dinamiche tra diffusione e topologia locale. Durante gli esperimenti, gli scienziati hanno utilizzato nanoparticelle d'oro (GNP) per etichettare i recettori del fattore di crescita epidermico (EGFR) nelle cellule HeLa. Gli EGFR sono proteine transmembrana di tipo I in grado di rilevare e rispondere a segnali extracellulari, la cui segnalazione aberrante è legata a una varietà di malattie. Taylor et al. ha mostrato la proteina marcata con GNP come un "nano-rover" che ha mappato la nano-topologia delle caratteristiche cellulari come i terreni della membrana, strutture filopodi e clatrina. Hanno fornito esempi di subdiffusione e movimento di confinamento nanoscopico di una proteina in 3-D ad alta risoluzione temporale e tempi lunghi.
Negli esperimenti, Taylor et al. ha introdotto le sonde del fattore di crescita epidermico-nanoparticelle d'oro (EGF-GNP) nella camera del campione del microscopio utilizzando una micropipetta per etichettare gli EGFR (recettori del fattore di crescita epidermico) sulle cellule HeLa e verificato che le sonde stimolassero gli EGFR. Precedenti studi avevano già indicato che la dimensione della sonda potrebbe influenzare i tassi di diffusione dei lipidi nelle membrane sintetiche, anche se non hanno influenzato la modalità di diffusione. Inoltre, nelle cellule vive, l'affollamento molecolare era trascurabile per particelle uguali o inferiori a 50 nm.
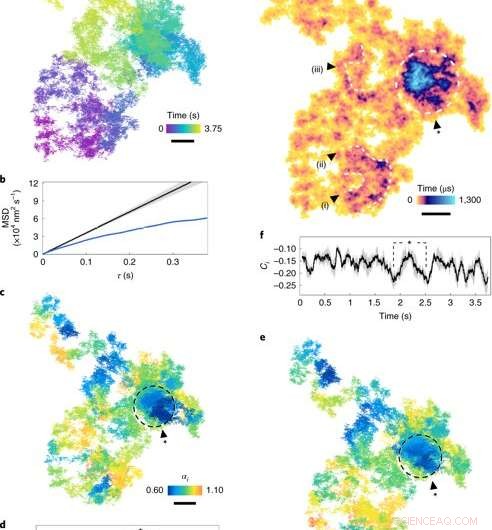
Diffusione sulla membrana plasmatica. (un), Una traiettoria di diffusione laterale (tempo di esposizione 17,5 μs, vedere la scala dei colori per la cronologia). (B), MSD (spostamento quadratico medio) rispetto a . La curva blu mostra l'MSD di a. La curva nera è una diffusione normale simulata (α= 1), con la busta grigia che indica l'incertezza. (C), L'esponente di diffusione delle finestre rotanti (scala dei colori) sulla traiettoria. Le regioni di subdiffusione (α<1) sono indicate da sfumature più scure. (D), αi attraverso il tempo. L'ombreggiatura grigia rappresenta un'incertezza media del 7 ± 4%, corrispondente a un intervallo di confidenza del 95% per una finestra di 100 ms (1, 000 fotogrammi) e τ= 250 μs. I punti contrassegnati dall'asterisco corrispondono al cerchio in c. (e), La direzione del passo Ci per le finestre avvolgibili lungo la traiettoria. (F), Il passo-direzione Ci tracciato nel tempo, con l'ombreggiatura che denota incertezza. (G), Grafico di occupazione ATOM con tempo di residenza (scala di colori). La dimensione del contenitore corrisponde all'errore di localizzazione. Regioni degne di nota di occupazione estesa, contrassegnati come loop e vortici (i)-(iii), sono indicativi di strutture nanoscopiche persistenti. La regione racchiusa rappresenta una fitta macchia di notevole subdiffusione. Barre della scala, 100 nm. Credito: Fotonica della natura , doi:10.1038/s41566-019-0414-6
Taylor et al. verificato questi due casi concreti nel presente lavoro confrontando GNP di vari diametri a 48 nm e 20 nm. Gli scienziati hanno quindi condotto studi fluorescenti e biochimici per suggerire che i GNP rivestiti di EGF attivassero la segnalazione di EGFR, proprio come gli EGF disponibili gratuitamente, indicando che l'etichetta non ha ostacolato le funzioni biologiche. Per superare il rumore di fondo correlato all'imaging molecolare, gli scienziati hanno implementato un nuovo algoritmo, che ha estratto l'intera funzione di diffusione del punto iSCAT (iSCAT-PSF) direttamente da ciascun fotogramma per chiarezza. Poiché le tecniche esistenti non sono in grado di visualizzare le caratteristiche ad alta risoluzione spaziale e temporale, molti dettagli sull'attività intracellulare rimangono oggetto di dibattito. In risposta, il nuovo metodo di Taylor et al. ha offerto una vasta gamma di eterogeneità dinamiche in 3-D per far luce sul movimento delle proteine intracellulari.
Gli scienziati hanno prima studiato quantitativamente la subdiffusione nella membrana plasmatica considerando un esempio 2-D del viaggio dell'EGFR sulla membrana di una cellula HeLa vivente. Per questo, hanno calcolato lo spostamento quadratico medio (MSD) per l'intera traiettoria del movimento. Taylor et al. non ha avuto bisogno di fare ipotesi sulla natura della diffusione o sul suo paesaggio geografico durante il calcolo. Hanno misurato la presenza di barriere e confinamenti diffrattivi biologici osservando il grado di correlazione direzionale tra due passaggi vettoriali in un intervallo di tempo.

Diffusione su filopodio. Credito: Fotonica della natura , doi:10.1038/s41566-019-0414-6
Gli scienziati hanno quindi valutato la popolarità di ciascun pixel della traiettoria nello spazio introducendo una mappa di occupazione temporale accumulata (ATOM). In questa tecnica, hanno diviso il piano laterale della traiettoria in contenitori di dimensioni nanometriche e hanno contato l'occorrenza della particella in ciascun contenitore. I risultati hanno indicato la disposizione delle nanostrutture in loop e vortici entro una durata minima di 250 nanosecondi (5000 frame) per rappresentare potenzialmente una fase pre-endocitica. In totale, le osservazioni simulate hanno mostrato come la diffusione delle proteine fosse influenzata dalla sottostruttura della cellula.
La tecnica di microscopia iSCAT ha permesso agli scienziati di registrare gli effetti per un periodo di tempo molto lungo, che hanno usato insieme alle capacità di imaging 3D per seguire gli EGFR su un filopodio. I filopodi sono sporgenze cellulari biologicamente simili a bastoncelli contenenti fasci di filamenti di actina fino a 100-300 nm di diametro e 100 µm di lunghezza. Le nanostrutture possono rilevare stimoli meccanici per la chemioattrazione o repulsione nel microambiente cellulare fornendo al contempo siti per l'attaccamento cellulare. Il legame del ligando e l'attivazione di EFGR sui filopodi si sono verificati a basse concentrazioni di EGF, seguita dalla sua associazione con i filamenti di actina e il trasporto retrogrado di EFGR al corpo cellulare.
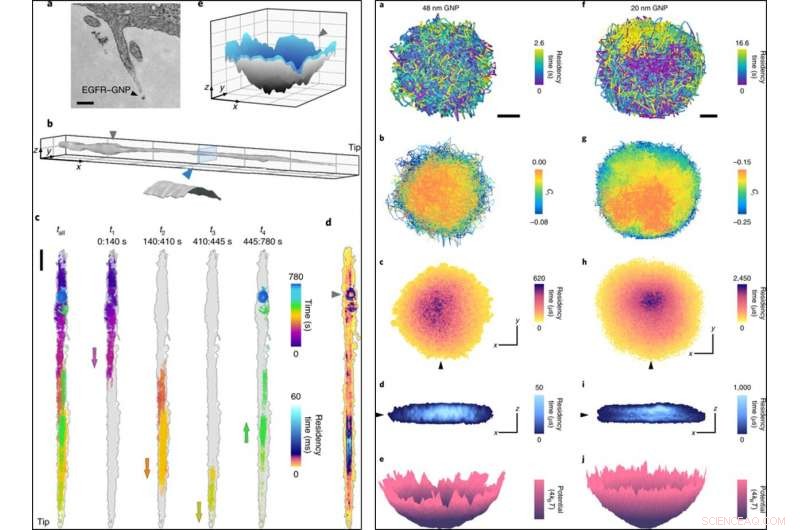
A SINISTRA:(a) Un'immagine TEM (microscopio elettronico a trasmissione) di un filopodio che include un EGFR-GNP. (B), Una superficie filopodio ricostruita dal 780, 000 punti di traiettoria con un errore di localizzazione di x, y = 2 nm registrato a 1, 000fps. inserto, sezione trasversale che raffigura una superficie cilindrica di diametro 150 nm dopo aver tenuto conto delle dimensioni del PNL. (C), Una traiettoria grezza di 13 min (a sinistra) spezzata in quattro pezzi successivi che rivelano il viaggio da e verso la punta, con frecce che indicano la direzione di movimento della rete. (D), Un diagramma ATOM di c, corretto per la deriva del filopodio. (e), Un'interpolazione di superficie degli ultimi anni '80. Il confinamento ad anello nella fase finale (contrassegnato da un triangolo) è una fossa 3D. Le barre della scala sono 200 nm (a), 1μm (x, y) e 200 nm (z) (b), 1, 000 nm (c) e 100 nm (x, y) e 50 nm (z) (e). DESTRA:(a), Una traiettoria laterale di una sonda GNP da 48 nm. Barra della scala, 100 nm. Un campionamento temporale inferiore di questo confinamento avrebbe sottovalutato l'estensione del confine. (B), Ci della traiettoria (usando un intervallo di cinque fotogrammi), che mostra una diffusione parzialmente ostacolata con una propensione a una diffusione più libera al centro. (C), Un diagramma ATOM di a. (D), Un taglio attraverso il grafico 3D-ATOM lungo la linea del triangolo nero in c mostra che l'occupazione favorisce una regione più interna simile a un disco. Gli assi indicano 100 nm sia in c che d. (e), Conversione dell'occupazione temporale 2D da c in un'effettiva distribuzione potenziale di energia. (f-j), Equivalente a a–e, ma per una sonda GNP da 20 nm. Credito: Fotonica della natura , doi:10.1038/s41566-019-0414-6
Gli scienziati hanno così acquisito informazioni sui dettagli nanoscopici della diffusione lungo il filopodio e hanno registrato i dati per 13 minuti. Hanno analizzato la traiettoria 3D per creare la topografia del filopodio utilizzando nanoparticelle d'oro come "nano rover" e mappato la topologia superficiale delle strutture cellulari per un esame più approfondito. Hanno tracciato la traiettoria ATOM (mappa di occupazione temporale accumulata) e hanno scoperto che la rappresentazione 3D era coerente con la fase biologica dell'invaginazione della membrana pre-endocitica.
Tecniche di microscopia ad alta velocità come iSCAT sono necessarie per ottenere informazioni temporali ad alta risoluzione e prevenire effetti di sfocatura durante l'imaging basato sulla localizzazione delle nanoparticelle. Gli scienziati hanno dimostrato questa caratteristica registrando la diffusione confinata a 30, 000 fps (fotogrammi al secondo) con GNP di 48 nm e 20 nm. Hanno seguito gli esperimenti con il tracciamento 3D ad altissima velocità delle proteine a 66, 000 fps utilizzando un breve tempo di esposizione di 10 µs in una durata di 3,5 secondi. L'imaging al microscopio iSCAT veloce ha fornito ulteriori prove per rivelare le complesse caratteristiche degli eventi endocitici relativi all'endocitosi mediata da clatrina nelle cellule HeLa quando simulati da basse concentrazioni di EGF.
In questo modo, Taylor et al. ha osservato che la nuova tecnica potrebbe registrare fedelmente le informazioni nano-topografiche. I risultati corrispondevano alle osservazioni registrate con la microscopia elettronica a trasmissione (TEM) senza differenze significative sulla riduzione delle dimensioni della sonda da 48 nm a 20 nm, fornendo allo stesso tempo nuovi spunti. Le nuove intuizioni includevano dettagli sulla subdiffusione, confinamento nanoscopico, Contorni 3-D di strutture filopodi e clatrina su scala nanometrica. Gli scienziati intendono combinare iSCAT con la microscopia a fluorescenza a super risoluzione in situ per comprendere le traiettorie delle proteine, virus e altre entità biologiche nanoscopiche. Taylor et al. mirano a far progredire i metodi di analisi delle immagini per tracciare GNP inferiori a 20 nm in futuro e credono che la nuova tecnologia e l'ottimizzazione aggiuntiva consentiranno loro di comprendere in modo specifico il ciclo di vita dei virus senza utilizzare un'etichetta esterna per il tracciamento.
© 2019 Scienza X Rete