
Progettazione e realizzazione dell'imaging MATRIEX:(a) Schema sperimentale del sistema di imaging MATRIEX. I due oggetti 3D rotondi nell'angolo in basso a sinistra sono le viste dall'alto e dal basso della camera della testa del mouse utilizzata per l'imaging in vivo. (Ti:Sa):Laser a impulsi ultraveloce Ti:Zaffiro; PC:cellula di Pockels; BE:espansore di travi; SM1 e SM2:specchi di scansione x–y; SL:lente di scansione; TL:lente a tubo; DM:specchio dicroico; CL:lente di raccolta; PMT:tubo fotomoltiplicatore; DO:obiettivo asciutto; MO:obiettivi miniaturizzati. (b) Fotografia che mostra una panoramica obliqua dell'attuale sistema di imaging MATRIEX. (c) La fotografia nell'immagine in alto mostra una vista ingrandita dei tre MO attaccati alle barre di manipolazione sopra la camera di testa; la foto in basso è stata scattata direttamente sopra gli MO con la fotocamera di uno smartphone. Tutti gli MO utilizzati in questa figura sono dello stesso modello:"versione standard". (d, e) Illustrazioni dell'ingrandimento a due stadi e dell'accoppiamento multiasse. Le immagini quadrate sono reali immagini a due fotoni scattate con perline da 20 μm. Ogni cerchio rosso indica un FOV. Il modello di DO utilizzato nei pannelli (d-f) è l'Olympus MPlan ×4/0.1, e tutti gli MO in questa figura sono dello stesso modello personalizzato. (f) Illustrazione che mostra l'assenza di diafonia inter-FOV sotto MO adiacenti. Le immagini sono state scattate su una lastra fluorescente uniforme. I cerchi rossi indicano le aree di analisi utilizzate per confrontare il contrasto dell'immagine tra due condizioni; la condizione a sinistra mostra la piastra fluorescente sotto entrambi i MO, e la condizione del lato destro mostra la piastra di fluorescenza sotto un solo MO. (g) Testare la risoluzione ottica dell'assieme composto con perline da 0,51 μm. Curve:raccordi gaussiani di punti dati grezzi. I profili di intensità di fluorescenza in asse o fuori asse sono stati misurati quando l'asse del MO era allineato con l'asse del DO o lontano dall'asse del DO (2 mm per il DO di ×4 o ×5, 3 mm per il DO di ×2,5, e 4 mm per il DO di ×2), rispettivamente. Credito:Luce:scienza e applicazioni, doi:10.1038/s41377-019-0219-x
L'imaging al microscopio a scansione laser a due fotoni viene comunemente applicato per studiare l'attività neuronale a risoluzioni cellulari e subcellulari nel cervello dei mammiferi. Tali studi sono ancora confinati a una singola regione funzionale del cervello. In un recente rapporto, Mengke Yang e colleghi del Brain Research Instrument Innovation Center, Istituto di Neuroscienze, Centro per la neuroscienza dei sistemi e la tecnologia di produzione avanzata dei sistemi ottici in Cina, La Germania e il Regno Unito hanno sviluppato una nuova tecnica denominata MATRIEX (esploratore in vitro multiarea a due fotoni in tempo reale). Il metodo ha permesso all'utente di mirare a più regioni del cervello funzionale con un campo visivo (FOV) di circa 200 µm di diametro per eseguire Ca a due fotoni 2+ imaging con risoluzione a singola cella contemporaneamente in tutte le regioni.
Yang et al. condotto imaging funzionale in tempo reale delle attività del singolo neurone nella corteccia visiva primaria, corteccia motoria primaria e regione CA1 dell'ippocampo durante gli stati anestetizzati e svegli nei topi. La tecnica MATRIEX può configurare in modo univoco più FOV microscopici utilizzando un singolo dispositivo di scansione laser. Di conseguenza, la tecnica può essere implementata come modulo ottico aggiuntivo all'interno della convenzionale scansione a raggio singolo esistente, microscopi a due fotoni senza ulteriori modifiche. Il MATRIEX può essere applicato per esplorare l'attività neuronale multiarea in vivo per la funzione del circuito neurale dell'intero cervello con risoluzione a cellula singola.
La microscopia laser a due fotoni è nata negli anni '90 per diventare popolare tra i neuroscienziati interessati allo studio delle strutture e delle funzioni neurali in vivo. Uno dei principali vantaggi dell'imaging a due e tre fotoni per i cervelli viventi include la risoluzione ottica ottenuta attraverso tessuti cerebrali densamente etichettati che diffondono fortemente la luce, durante la quale i pixel dell'immagine sezionati otticamente possono essere scansionati e acquisiti con una diafonia minima. Però, i vantaggi comportavano anche notevoli inconvenienti al metodo impedendo la visione simultanea di due oggetti entro una determinata distanza. I ricercatori avevano precedentemente implementato molte strategie per estendere i limiti, ma i metodi erano difficili da implementare nei laboratori di ricerca delle neuroscienze. Tuttavia, esiste una richiesta sempre più elevata nelle neuroscienze per studiare le funzioni neuronali a livello del cervello con risoluzione di singole cellule in vivo.

A SINISTRA:Schema sperimentale del sistema di imaging MATRIEX. I due oggetti 3D rotondi nell'angolo in basso a sinistra sono le viste dall'alto e dal basso della camera della testa del mouse utilizzata per l'imaging in vivo. (Ti:Sa):Laser a impulsi ultraveloce Ti:Zaffiro; PC:cellula di Pockels; BE:espansore di travi; SM1 e SM2:specchi di scansione x–y; SL:lente di scansione; TL:lente a tubo; DM:specchio dicroico; CL:lente di raccolta; PMT:tubo fotomoltiplicatore; DO:obiettivo asciutto; MO:obiettivi miniaturizzati. A DESTRA:illustrazioni dell'ingrandimento a due stadi e dell'accoppiamento multiasse. Le immagini quadrate sono reali immagini a due fotoni scattate con perline da 20 μm. Ogni cerchio rosso indica un FOV. Credito:Luce:scienza e applicazioni, doi:10.1038/s41377-019-0219-x
In un approccio diretto, gli scienziati possono posizionare due microscopi sopra lo stesso cervello animale per visualizzare simultaneamente la corteccia e il cervelletto. Ma tali sforzi possono portare a sostanziali aumenti di complessità e costi. Le elevate aspettative esistenti in termini di prestazioni e fattibilità pongono quindi una domanda ingegneristica molto impegnativa su come un singolo sistema di imaging possa ottenere simultaneamente immagini microscopiche dal vivo da più regioni del cervello in vivo. Per rispondere alla domanda, Yang et al. ha introdotto un nuovo metodo che combinava l'ingrandimento a due stadi e l'accoppiamento ottico multiasse.
Hanno realizzato il metodo utilizzando un obiettivo secco a basso ingrandimento (DO), con multiplo immerso in acqua, obiettivi miniaturizzati (MO) nell'ambito dell'obiettivo secco. Gli scienziati hanno posizionato ciascuno degli MO nella posizione e nella profondità desiderate nel tessuto cerebrale. Il team ha utilizzato il nuovo assemblaggio dell'oggetto composto in modo simile all'obiettivo del microscopio a immersione originale senza ulteriori modifiche al sottosistema di scansione e acquisizione delle immagini.
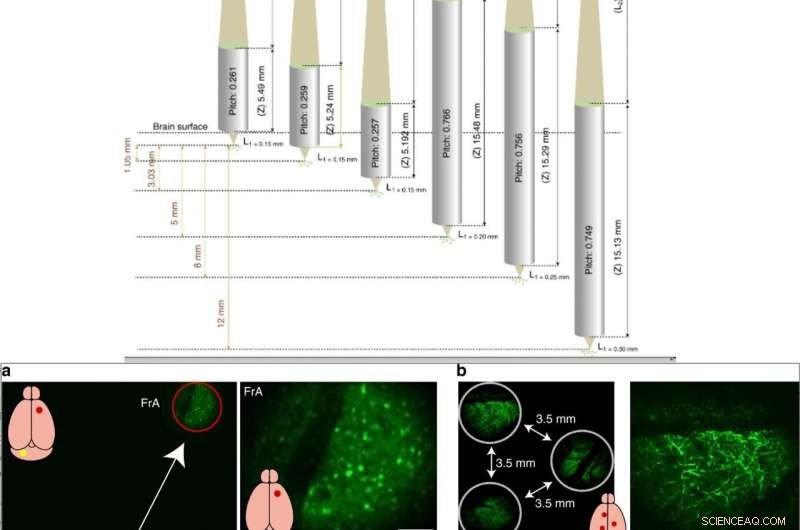
TOP:Configurazione degli MO con parametri diversi per mirare a piani di oggetti a diverse profondità per poi essere coniugati sullo stesso piano dell'immagine. Ogni cilindro grigio rappresenta una lente con un valore di passo, distanza di lavoro anteriore (L1), distanza di lavoro posteriore (L2) e lunghezza (Z). BOTTOM:Dimostrazione dell'imaging MATRIEX:imaging strutturale in più aree cerebrali in vivo. a Immagine a sinistra:un'immagine full frame che include due FOV nella corteccia associativa frontale (FrA) e nel cervelletto. I cerchi rosso e giallo indicano due FOV ingranditi digitalmente e mostrati nelle immagini in alto a destra e in basso a destra. È stato utilizzato un topo transgenico GAD67-GFP (con gli interneuroni etichettati a livello cerebrale). Due MO ("versione standard") sono stati posizionati alla stessa profondità sotto un DO (Mitutoyo ×2/0.055). b Esempio di configurazione di tre FOV nella corteccia di un topo transgenico Thy1-GFP (con neuroni corticali di strato 5 specificamente etichettati e con dendriti a ciuffo visibili vicino alla superficie corticale). Tre MO ("versione standard") sono stati posizionati alla stessa profondità sotto un DO (Olympus ×4/0.1). Credito:Luce:scienza e applicazioni, doi:10.1038/s41377-019-0219-x
Il team di ricerca ha prima assemblato l'obiettivo composto MATRIEX. Per questo, hanno sostituito il convenzionale obiettivo del microscopio a immersione in acqua con un gruppo obiettivo composto personalizzato, all'interno di un microscopio a scansione laser a due fotoni dotato di un dispositivo di scansione raster convenzionale a raggio singolo. L'assemblaggio composto conteneva più MO (obiettivi miniaturizzati) inseriti attraverso più craniotomie durante le quali gli scienziati hanno incollato una camera di plastica stampata in 3D al cranio del modello murino. La camera ha allineato approssimativamente i MO con lo stesso spazio per regolare la posizione laterale e la profondità. Yang et al. manipolato con precisione i singoli MO per visualizzare gli oggetti sotto tutti gli MO contemporaneamente nello stesso piano dell'immagine.
Hanno implementato il metodo MATRIEX utilizzando due principi; ingrandimento a due stadi e accoppiamento multiasse. Per esempio, utilizzando l'ingrandimento a due stadi con il solo obiettivo a secco (DO), hanno osservato perline da 20 µm come minuscoli punti sfocati mentre osservavano nitidi, cerchi circolari attraverso l'assieme composto. Durante l'accoppiamento multiasse, gli scienziati hanno accoppiato un singolo DO con più MO sullo stesso piano dell'immagine. Utilizzando una semplice scansione raster in un'unica cornice rettangolare, il team di ricerca ha acquisito un'immagine rettangolare contenente più FOV circolari (Field of Views) – dove ogni FOV corrispondeva a un MO con diafonia di pixel inter-FOV minima.
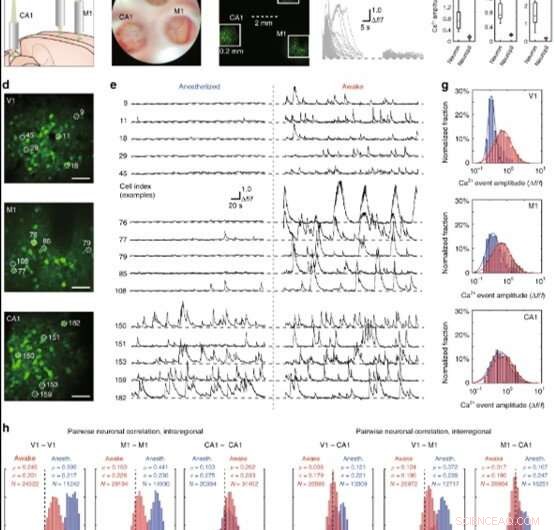
Dimostrazione dell'imaging MATRIEX:acquisizione simultanea di modelli di attività neuronale in tempo reale in V1, M1, e CA1 ippocampale nei topi nello stato anestetizzato o nello stato di veglia. I neuroni sono stati marcati da un indicatore fluorescente di Ca2+ codificato geneticamente, GCaMP6f (a) Illustrazione che mostra il posizionamento di tre MO sulla V1, M1 e regioni CA1 dell'ippocampo in un cervello di topo modello. (b) Una fotografia della fotocamera scattata attraverso la lente oculare del microscopio con illuminazione in campo chiaro a luce bianca, in cui tre FOV sono facilmente visibili. La regione superiore è V1, la regione in basso a sinistra è CA1, e la regione in basso a destra è M1. (c) Un'immagine a due fotoni, che è una media di 100 fotogrammi, acquisita mediante semplice scansione raster full frame con un microscopio a due fotoni. Le caselle bianche piene mostrano le tre parti dell'immagine che sono ingrandite nel pannello (d). (d) FOV individuali ingranditi digitalmente che mostrano i neuroni in V1, M1, e CA1, da cima a fondo. Barra della scala:40μm. (e) Tracce del segnale Ca2+ time-lapse di cinque cellule di esempio da ciascuna regione, con ciascuno etichettato dall'indice di cella. Vengono mostrate le registrazioni della stessa cellula nello stesso animale nello stato anestetizzato (lato sinistro) e nello stato di veglia (lato destro). (f) Sinistra:tracce che mostrano eventi di segnale di Ca2+ individuali (divisi da ciascun tempo di insorgenza e sovrapposti) da celle di esempio selezionate casualmente. Al centro:tracce di segnale di Ca2+ di ciascuna delle zone di neuropilo direttamente adiacenti a ciascuna delle cellule di esempio. A destra:tre box plot che confrontano l'ampiezza dell'evento del segnale Ca2+ neuronale con l'ampiezza del segnale Ca2+ del neurone adiacente; test della somma dei ranghi di Wilcoxon accoppiato, ***P < 0,001. (g) Adattamento log-normale degli istogrammi di distribuzione dell'ampiezza dell'evento spontaneo del Ca2+ per i dati raccolti da tutti gli animali. Le barre rosse e la curva adattata mostrano la distribuzione dei dati registrati nello stato di veglia, e le barre blu e la curva adattata mostrano la distribuzione dei dati registrati nello stato anestetizzato. (h) Correlazione dell'attività neuronale a coppie (coefficienti di correlazione di Pearson) per i dati raccolti da tutti gli animali. Le barre rosse mostrano la distribuzione dei dati registrati nello stato di veglia, e le barre blu mostrano la distribuzione dei dati registrati nello stato anestetizzato. Credito:Luce:scienza e applicazioni, doi:10.1038/s41377-019-0219-x
Gli scienziati hanno attribuito all'ingrandimento dell'apertura numerica (NA) il merito di aver consentito una migliore risoluzione con l'assieme composto. Gli obiettivi associati erano anche flessibili e progettati su misura per la produzione di massa a basso costo per aiutare la progettazione sperimentale. La caratteristica principale di MATRIEX era la sua capacità di visualizzare più oggetti contemporaneamente a grandi intervalli di profondità. Per evidenziare questo, Yang et al. progettato diversi MO con parametri diversi, posizionandoli ad una profondità specifica dove i corrispondenti piani dell'oggetto si coniugano sullo stesso asse. In pratica, il team di ricerca ha compensato le discrepanze minori tra la profondità dell'oggetto desiderata e quella effettiva regolando i MO individualmente lungo ciascuno degli assi z.
Tipicamente, sotto il DO (obiettivo asciutto) la dimensione laterale massima della zona target è limitata dalla dimensione massima del campo di scansione. Per esempio, utilizzando un DO con un ingrandimento 2x e una zona target di 12 mm di diametro, gli scienziati possono immaginare un intero cervello di topo adulto. In questo studio, Yang et al. simultaneamente hanno ripreso la corteccia associativa frontale e il cervelletto del topo. In pratica, un obiettivo aereo 4x era adatto per ottenere una migliore risoluzione per osservare le strutture fini dei dendriti.

Imaging simultaneo del calcio nella V1, Regioni M1 e CA1 che utilizzano MATRIEX durante gli stati anestetizzati e svegli nei topi. Guarda il film completo su Credit:Light:Science &Applications, doi:10.1038/s41377-019-0219-x
Come prova di principio, il team di ricerca ha utilizzato MATRIEX per eseguire Ca . a due fotoni simultanei 2+ imaging di neuroni fluorescenti nella corteccia visiva primaria (regione V1), corteccia motoria primaria (regione M1) e regione ippocampale CA1 dei topi. Nella configurazione dei tre MO, gli scienziati hanno posizionato due MO adatti per la regione V1 e M1, direttamente sopra la corteccia e inserito un MO all'interno della regione CA1 dell'ippocampo dopo aver rimosso chirurgicamente un tessuto corticale. Il team ha quindi progettato le lenti per i piani oggetto corrispondenti a V1, M1 e CA1 per la coniugazione sullo stesso piano dell'immagine. Utilizzando un microscopio a due fotoni dotato di uno scanner risonante a 12 kHz, gli scienziati hanno scansionato l'immagine completa per osservare tre FOV e le loro singole cellule dopo aver ingrandito le tre diverse sezioni per risolvere i singoli neuroni. Quindi hanno notato la potenza del laser da distribuire tra più FOV.
Mentre Yang et al. avrebbe potuto ottenere questi risultati utilizzando l'imaging convenzionale a singolo FOV all'interno di una singola regione del cervello, la tecnica MATRIEX ha fornito loro dati superiori a quelli offerti con le tecniche di imaging a FOV singolo. Presi insieme, questi risultati hanno permesso una distribuzione e una trasformazione altamente disomogenee dei modelli di attività spontanea dallo stato anestetizzato allo stato di veglia nei topi, che copre un livello di circuito a livello di cervello alla risoluzione di una singola cella.
In questo modo, Menge Yang e collaboratori hanno sviluppato la tecnica MATRIEX basata sul principio dell'ingrandimento a due stadi e dell'accoppiamento ottico multiasse. Hanno condotto simultaneamente Ca . a due fotoni 2+ imaging nelle attività della popolazione neuronale a diverse profondità in diverse regioni (V1, M1 e CA1) in topi anestetizzati e svegli con risoluzione unicellulare. È importante sottolineare che qualsiasi microscopio convenzionale a due fotoni può essere trasformato in un microscopio MATRIEX, preservando tutte le funzionalità originali. La chiave per la trasformazione si basa sulla progettazione di un gruppo obiettivo composto. I ricercatori possono utilizzare diversi, MO accuratamente progettati per adattarsi a diverse regioni del cervello con una compatibilità al 100% tra la tecnica MATRIEX e la microscopia convenzionale. Il team di ricerca si aspetta che la tecnica MATRIEX faccia avanzare sostanzialmente la tridimensionalità, dinamica del circuito neurale dell'intero cervello alla risoluzione di una singola cellula.
© 2019 Scienza X Rete