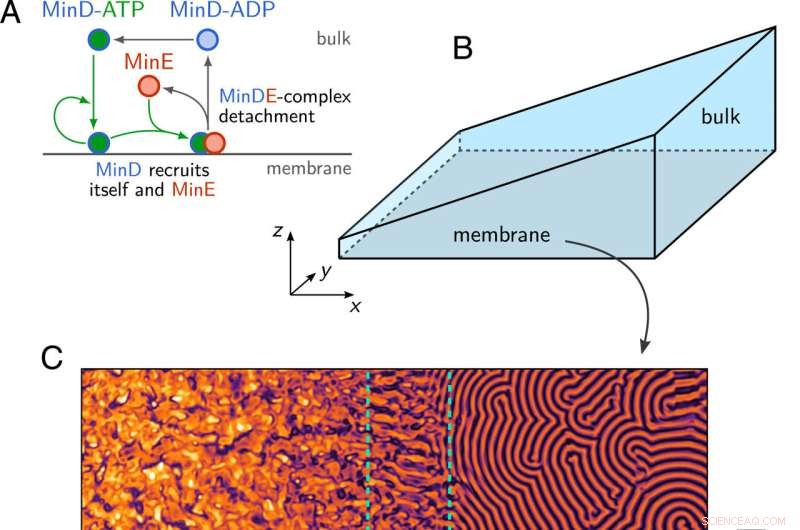
(A) Illustrazione schematica della rete di reazione proteica Min. (B) Geometria del cuneo con una superficie della membrana sul piano inferiore (z =0) e l'altezza di massa H(x) che aumenta linearmente lungo la direzione x. (C) Istantanea della densità di membrana di MinD, ottenuta simulando numericamente le Eq dinamiche Min. 1–3 nella geometria mostrata in B. Si osservano regioni con schemi caotici, onde stazionarie (SW, contorno verde tratteggiato) e onde viaggianti (TW) lungo la membrana ea diverse altezze di massa. Credito:Atti dell'Accademia Nazionale delle Scienze (2022). DOI:10.1073/pnas.2206888119
Per molti processi importanti per la vita come la divisione cellulare, la migrazione cellulare e lo sviluppo degli organi, la formazione spazialmente e temporalmente corretta di modelli biologici è essenziale. Per comprendere questi processi, il compito principale non consiste nello spiegare come i modelli si formano da una condizione iniziale omogenea, ma nello spiegare come i modelli semplici si trasformano in modelli sempre più complessi. Illuminare i meccanismi di questa complessa auto-organizzazione su varie scale spaziali e temporali è una sfida chiave per la scienza.
Le cosiddette tecniche di "grana grossa" consentono di semplificare tali sistemi multiscala, in modo tale da poter essere descritti con un modello ridotto a grandi scale di lunghezza e tempo. "Il prezzo da pagare per la grana grossa, tuttavia, è che informazioni importanti sui modelli su piccola scala, come il tipo di modello, vanno perse. Ma il fatto è che questi modelli svolgono un ruolo decisivo nei sistemi biologici. Per fare un esempio , controllano importanti processi cellulari", spiega Laeschkir Würthner, membro del team guidato dal fisico LMU Prof. Erwin Frey e autore principale di un nuovo studio pubblicato negli Proceedings of the National Academy of Sciences che supera questo problema.
In collaborazione con il gruppo di ricerca del Prof. Cees Dekker (TU Delft), il team di Frey ha sviluppato un nuovo approccio a grana grossa per i cosiddetti sistemi di reazione-diffusione a conservazione di massa, in cui l'analisi su larga scala delle densità totali di le particelle coinvolte consentono la previsione di modelli su piccola scala.
Gli scienziati hanno illustrato il potenziale del loro approccio con il sistema proteico Min, un modello paradigmatico per la formazione di pattern biologici. Il batterio E. coli utilizza varie proteine Min che circolano in una cellula per determinare in quale posizione avviene la divisione cellulare. Un fattore decisivo qui è che le proteine coinvolte si verificano a frequenze diverse a seconda della loro posizione nella cellula e dello stato chimico, vale a dire che hanno una varietà di densità diverse.
"Siamo ora riusciti a ridurre la complessità di questo sistema sviluppando una teoria che si basa esclusivamente sulla densità totale delle proteine, in modo tale da poter rispecchiare completamente la dinamica della formazione del modello", afferma Frey. "Si tratta di un'enorme riduzione. I calcoli numerici ora vengono eseguiti in pochi minuti anziché in mesi."
I ricercatori sono stati in grado di confermare sperimentalmente le previsioni teoriche del modello, secondo cui la distribuzione delle proteine dipende dalla geometria dell'ambiente. Lo hanno fatto ricostruendo il sistema proteico Min in una cella a flusso in vitro, con i risultati che mostrano gli stessi schemi proteici rivelati nella simulazione.
"Tale ricostruzione delle informazioni su piccola scala da dinamiche ridotte a livello macroscopico apre nuove strade per una migliore comprensione dei complessi sistemi multiscala, che si verificano in un'ampia gamma di sistemi fisici", afferma Frey. + Esplora ulteriormente