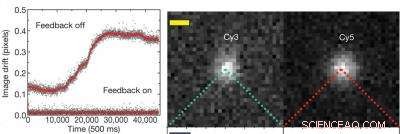
Il grafico a sinistra mostra che con il sistema di feedback attivo spento si ha una deriva di risoluzione di circa 0,3 pixel o 19 nanometri, ma con il sistema di feedback la risoluzione si mantiene migliore di 0,01 pixel, o circa 0,64 nanometri. L'immagine a destra mostra le singole molecole di colorante fluorescente di cianina (Cy) - Cy3 e Cy5 - utilizzate per etichettare 20 coppie di basi di DNA a doppio filamento. Credito:per gentile concessione del gruppo di ricerca Steven Chu
La saggezza convenzionale sostiene che la microscopia ottica non può essere utilizzata per "vedere" qualcosa di così piccolo come una singola molecola. Ma come è consuetudine, la scienza intelligente ha rovesciato ancora una volta la saggezza convenzionale. Segretario dell'Energia, Il premio Nobel ed ex direttore del Lawrence Berkeley National Laboratory (Berkeley Lab) Steven Chu ha guidato lo sviluppo di una tecnica che consente l'uso della microscopia ottica per visualizzare oggetti o la distanza tra loro con risoluzioni di appena 0,5 nanometri - la metà di un miliardesimo di metro, o un ordine di grandezza inferiore al migliore precedente.
"La capacità di ottenere una risoluzione sub-nanometrica in ambienti acquosi biologicamente rilevanti ha il potenziale per rivoluzionare la biologia, in particolare biologia strutturale, " dice il segretario Chu. "Una delle motivazioni per questo lavoro, Per esempio, era misurare le distanze tra le proteine che formano multi-dominio, strutture molto complesse, come l'assemblaggio proteico che forma il sistema della RNA polimerasi II umana, che avvia la trascrizione del DNA."
Il segretario Chu è il coautore di un articolo che ora appare sulla rivista Natura che descrive questa ricerca. Il documento è intitolato "Localizzazione subnanometrica di singole molecole, registrazione e misurazioni della distanza." Gli altri autori sono Alexandros Pertsinidis, un ricercatore post-dottorato e membro del gruppo di ricerca di Chu presso l'Università della California (UC) Berkeley, che ora è assistente professore allo Sloan-Kettering Institute, e Yunxiang Zhang, un membro del gruppo di ricerca di Chu alla Stanford University.
Secondo una legge della fisica nota come "limite di diffrazione, " l'immagine più piccola che un sistema ottico può risolvere è circa la metà della lunghezza d'onda della luce utilizzata per produrre quell'immagine. Per l'ottica convenzionale, questo corrisponde a circa 200 nanometri. A confronto, una molecola di DNA misura circa 2,5 nanometri di larghezza.
Mentre i sistemi di imaging non ottici, come microscopi elettronici, può risolvere bene gli oggetti nella scala subnanometrica, questi sistemi operano in condizioni non ideali per lo studio di campioni biologici. Rilevamento di singole etichette fluorescenti attaccate a molecole biologiche di interesse utilizzando dispositivi ad accoppiamento di carica (CCD) - matrici di chip di silicio che convertono la luce in ingresso in una carica elettrica, ha prodotto risoluzioni fino a cinque nanometri. Però, fino ad ora questa tecnologia non è stata in grado di visualizzare singole molecole o distanze tra una coppia di molecole molto inferiori a 20 nanometri.
Chu e i suoi coautori sono stati in grado di utilizzare la stessa tecnologia di fluorescenza CCD per risolvere le distanze con precisione e accuratezza subnanometriche correggendo un trucco della luce. Le cariche elettriche in un array CCD vengono create quando i fotoni colpiscono il silicio e staccano gli elettroni, con la forza della carica proporzionale all'intensità dei fotoni incidenti. Però, dipende precisamente da dove un fotone colpisce la superficie di un chip di silicio, può esserci una leggera differenza nel modo in cui il fotone viene assorbito e se genera una carica misurabile. Questa non uniformità nella risposta dell'array di silicio CCD ai fotoni in ingresso, che è probabilmente un artefatto del processo di fabbricazione del chip, provoca una sfocatura dei pixel che rende difficile la risoluzione di due punti che si trovano a pochi nanometri l'uno dall'altro.
"Abbiamo sviluppato un sistema di feedback attivo che ci consente di posizionare l'immagine di una singola molecola fluorescente ovunque sull'array CCD con precisione sub-pixel, che a sua volta ci consente di lavorare in una regione più piccola della tipica scala di lunghezza di tre pixel della non uniformità del CCD, "dice Pertsinidis, chi è l'autore principale dell'articolo su Nature. "Con questo sistema di feedback e l'uso di fasci ottici aggiuntivi per stabilizzare il sistema del microscopio, possiamo creare una regione calibrata sull'array di silicio dove l'errore dovuto alla non uniformità è ridotto a 0,5 nanometri. Posizionando le molecole che vogliamo misurare al centro di questa regione, possiamo ottenere una risoluzione subnanometrica utilizzando un microscopio ottico convenzionale che puoi trovare in qualsiasi laboratorio di biologia".
Chu afferma che la capacità di spostare il tavolino di un microscopio per piccole distanze e calcolare il centro geometrico (centroide) dell'immagine rende possibile non solo misurare la non uniformità della foto-risposta tra i pixel, ma anche per misurare la non uniformità all'interno di ogni singolo pixel.
"Conoscere questa disuniformità ci permette poi di apportare correzioni tra la posizione apparente e la posizione reale del baricentro dell'immagine, " dice Chu. "Poiché questa risposta non uniforme è integrata nell'array CCD e non cambia di giorno in giorno, il nostro sistema di feedback attivo ci consente di visualizzare ripetutamente la stessa posizione dell'array CCD."
Pertsinidis sta continuando a lavorare con Chu e altri nel gruppo sull'ulteriore sviluppo e applicazione di questa tecnica di super risoluzione. Oltre al sistema della RNA polimerasi II umana, lui e il gruppo lo stanno usando per determinare la struttura delle molecole di caderina epiteliale che sono responsabili dell'adesione cellula-cellula che tiene insieme i tessuti e altri materiali biologici. Pertsinidis, Zhang, e un altro postdoc nel gruppo di ricerca di Chu, Sang Ryul Park, utilizzano questa tecnica anche per creare misurazioni 3D dell'organizzazione molecolare all'interno delle cellule cerebrali.
"L'idea è di determinare la struttura e la dinamica del processo di fusione delle vescicole che rilascia le molecole di neurotrasmettitore utilizzate dai neuroni per comunicare tra loro, " Dice Pertsinidis. "In questo momento stiamo ottenendo misurazioni in situ con una risoluzione di circa 10 nanometri, ma pensiamo di poter spingere questa risoluzione entro due nanometri".
In collaborazione con Joe Gray, Direttore associato del Berkeley Lab per le scienze della vita e uno dei principali ricercatori sul cancro, anche i postdoc del gruppo di ricerca di Chu utilizzano la tecnica della super-risoluzione per studiare l'attacco delle molecole di segnalazione sulla proteina RAS, che è stato collegato a una serie di tumori, compresi quelli del seno, pancreas, polmone e colon. Questa ricerca potrebbe aiutare a spiegare perché le terapie contro il cancro che funzionano bene su alcuni pazienti sono inefficaci su altri.
Oltre alle sue applicazioni biologiche, Pertsinidis, Zhang e Chu nella loro Natura carta affermano che la loro tecnica di super-risoluzione dovrebbe anche rivelarsi preziosa per caratterizzare e progettare sistemi di imaging fotometrico di precisione in fisica atomica o astronomia, e consentire nuovi strumenti in litografia ottica e nanometrologia.