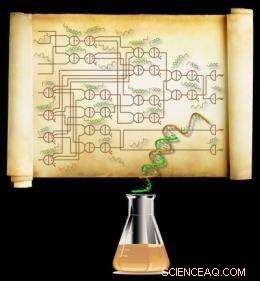
Uno schema elettrico che specifica un sistema di 74 molecole di DNA che costituiscono il più grande circuito sintetico di questo tipo mai realizzato. Il circuito calcola la radice quadrata di un numero fino a 15 e arrotonda per difetto all'intero più vicino (la radice quadrata discreta di un numero intero a quattro bit). Attestazione:Caltech/Lulu Qian
In molti modi, la vita è come un computer. Il genoma di un organismo è il software che dice al macchinario cellulare e molecolare, l'hardware, cosa fare. Ma invece di circuiti elettronici, la vita si basa su circuiti biochimici, reti complesse di reazioni e percorsi che consentono agli organismi di funzionare. Ora, i ricercatori del California Institute of Technology (Caltech) hanno costruito il circuito biochimico più complesso mai creato da zero, realizzati con dispositivi a base di DNA in una provetta analoghi ai transistor elettronici su un chip di computer.
L'ingegneria di questi circuiti consente ai ricercatori di esplorare i principi dell'elaborazione delle informazioni nei sistemi biologici, e per progettare percorsi biochimici con capacità decisionali. Tali circuiti darebbero ai biochimici un controllo senza precedenti nella progettazione di reazioni chimiche per applicazioni nell'ingegneria e nelle industrie biologiche e chimiche. Per esempio, in futuro un circuito biochimico sintetico potrebbe essere introdotto in un campione di sangue clinico, rilevare i livelli di una varietà di molecole nel campione, e integrare tali informazioni in una diagnosi della patologia.
"Stiamo cercando di prendere in prestito le idee che hanno avuto un enorme successo nel mondo elettronico, come rappresentazioni astratte di operazioni di calcolo, linguaggi di programmazione, e compilatori, e applicarli al mondo biomolecolare, "dice Lulu Qian, uno studioso postdottorato senior in bioingegneria al Caltech e autore principale di un articolo pubblicato nel numero del 3 giugno della rivista Scienza .
Insieme a Erik Winfree, Professore di informatica al Caltech, sistemi di calcolo e neurali, e bioingegneria, Qian ha utilizzato un nuovo tipo di componente basato sul DNA per costruire il più grande circuito biochimico artificiale mai realizzato. I precedenti circuiti biochimici realizzati in laboratorio erano limitati perché funzionavano in modo meno affidabile e prevedibile quando ridimensionati a dimensioni maggiori, spiega Qian. La probabile ragione alla base di questa limitazione è che tali circuiti necessitano di varie strutture molecolari per implementare funzioni diverse, rendendo i sistemi di grandi dimensioni più complicati e difficili da eseguire il debug. Il nuovo approccio dei ricercatori, però, coinvolge componenti semplici, standardizzato, affidabile, e scalabile, il che significa che è possibile realizzare circuiti ancora più grandi e complessi e continuare a funzionare in modo affidabile.
"Puoi immaginare che nell'industria informatica, vuoi creare computer sempre migliori, " Qian dice. "Questo è il nostro sforzo per fare lo stesso. Vogliamo realizzare circuiti biochimici sempre migliori in grado di svolgere compiti più sofisticati, spingendo i dispositivi molecolari ad agire sul loro ambiente."
Per costruire i loro circuiti, i ricercatori hanno utilizzato pezzi di DNA per creare le cosiddette porte logiche, dispositivi che producono segnali di uscita on-off in risposta a segnali di ingresso on-off. Le porte logiche sono gli elementi costitutivi dei circuiti logici digitali che consentono a un computer di eseguire le azioni giuste al momento giusto. In un computer convenzionale, le porte logiche sono realizzate con transistor elettronici, che sono cablati insieme per formare circuiti su un chip di silicio. circuiti biochimici, però, sono costituiti da molecole che galleggiano in una provetta di acqua salata. Invece di dipendere dagli elettroni che entrano ed escono dai transistor, Le porte logiche basate sul DNA ricevono e producono molecole come segnali. I segnali molecolari viaggiano da una porta specifica all'altra, collegando il circuito come se fossero fili.
Winfree e i suoi colleghi hanno costruito per la prima volta un circuito biochimico di questo tipo nel 2006. In questo lavoro, Le molecole del segnale del DNA collegavano tra loro diverse porte logiche del DNA, formando quello che viene chiamato un circuito multistrato. Ma questo circuito precedente consisteva solo di 12 diverse molecole di DNA, e il circuito rallentava di alcuni ordini di grandezza quando si espandeva da una singola porta logica a un circuito a cinque strati. Nel loro nuovo design, Qian e Winfree hanno progettato porte logiche più semplici e affidabili, consentendo loro di realizzare circuiti almeno cinque volte più grandi.
Le loro nuove porte logiche sono costituite da pezzi di breve, DNA a singolo filamento o DNA parzialmente a doppio filamento in cui i singoli filamenti sporgono come code dalla doppia elica del DNA. Le molecole di DNA a singolo filamento agiscono come segnali di input e output che interagiscono con quelle parzialmente a doppio filamento.
"Le molecole stanno semplicemente fluttuando nella soluzione, urtandosi di tanto in tanto, " spiega Winfree. "Di tanto in tanto, un filamento in arrivo con la giusta sequenza di DNA si comprimerà fino a un filamento e contemporaneamente ne aprirà un altro, rilasciandolo in soluzione e permettendogli di reagire con un altro filamento." Poiché i ricercatori possono codificare qualsiasi sequenza di DNA vogliano, hanno il pieno controllo su questo processo. "Hai questa interazione programmabile, " lui dice.
Qian e Winfree hanno fatto diversi circuiti con il loro approccio, ma il più grande, contenente 74 diverse molecole di DNA, può calcolare la radice quadrata di qualsiasi numero fino a 15 (tecnicamente parlando, qualsiasi numero binario a quattro bit) e arrotondare il risultato all'intero più vicino. I ricercatori monitorano quindi le concentrazioni delle molecole in uscita durante i calcoli per determinare la risposta. Il calcolo richiede circa 10 ore, quindi non sostituirà il tuo laptop in qualunque momento presto. Ma lo scopo di questi circuiti non è competere con l'elettronica; è dare agli scienziati il controllo logico sui processi biochimici.
I loro circuiti hanno diverse caratteristiche nuove, dice Qian. Perché le reazioni non sono mai perfette:le molecole non si legano sempre correttamente, per esempio, c'è un rumore intrinseco nel sistema. Ciò significa che i segnali molecolari non sono mai completamente accesi o spenti, come sarebbe il caso della logica binaria ideale. Ma le nuove porte logiche sono in grado di gestire questo rumore sopprimendo e amplificando i segnali, ad esempio amplificando un segnale che è all'80 percento, o inibendone uno che è al 10 percento, risultando in segnali che sono presenti o inesistenti al 100%.
Tutte le porte logiche hanno strutture identiche con sequenze diverse. Di conseguenza, possono essere standardizzati, in modo che gli stessi tipi di componenti possano essere cablati insieme per realizzare qualsiasi circuito tu voglia. Cosa c'è di più, Qian dice, non devi sapere nulla del macchinario molecolare dietro il circuito per crearne uno. Se vuoi un circuito che, dire, diagnostica automaticamente una malattia, devi semplicemente inviare una rappresentazione astratta delle funzioni logiche nel tuo progetto a un compilatore che i ricercatori forniscono online, che poi tradurrà il progetto nei componenti del DNA necessari per costruire il circuito. Nel futuro, un produttore esterno può quindi realizzare quelle parti e fornirti il circuito, pronto ad andare.
Anche i componenti del circuito sono sintonizzabili. Regolando le concentrazioni dei tipi di DNA, i ricercatori possono modificare le funzioni delle porte logiche. I circuiti sono versatili, con componenti plug-and-play che possono essere facilmente riconfigurati per ricablare il circuito. La semplicità delle porte logiche consente anche tecniche più efficienti che le sintetizzano in parallelo.
"Come la legge di Moore per l'elettronica al silicio, che dice che i computer stanno diventando esponenzialmente più piccoli e più potenti ogni anno, i sistemi molecolari sviluppati con la nanotecnologia del DNA raddoppiano di dimensioni all'incirca ogni tre anni, " dice Winfree. Qian aggiunge, "Il sogno è che i circuiti biochimici sintetici un giorno raggiungano complessità paragonabili alla vita stessa".