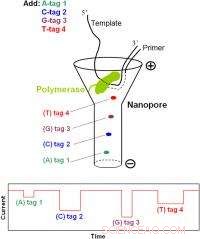
Schema del sequenziamento del DNA di una singola molecola da parte di un nanoporo con nucleotidi marcati con fosfato. Ciascuno dei quattro nucleotidi porterà un tag diverso. Durante SBS, questi tag, attaccato tramite il terminale-fosfato del nucleotide, verranno rilasciati nel nanoporo uno alla volta dove produrranno firme di blocco correnti uniche per la determinazione della sequenza. Una vasta gamma di tali nanopori porterà al sequenziamento del DNA ad alto rendimento.
(Phys.org)—Il sequenziamento del DNA è la forza trainante delle scoperte chiave in medicina e biologia. Ad esempio, la sequenza completa del genoma di un individuo fornisce importanti marcatori e linee guida per la diagnostica medica e l'assistenza sanitaria. Fino ad ora, il principale ostacolo è stato il costo e la velocità di ottenere sequenze di DNA altamente accurate. Sebbene negli ultimi 10 anni siano stati fatti numerosi progressi, la maggior parte degli attuali strumenti di sequenziamento ad alto rendimento dipendono da tecniche ottiche per il rilevamento dei quattro elementi costitutivi del DNA:A, C, G e T. Per migliorare ulteriormente la capacità di misurazione, è stato anche sviluppato il sequenziamento elettronico del DNA di un insieme di modelli di DNA.
Recentemente, è stato dimostrato che il DNA può essere infilato attraverso pori su nanoscala proteica sotto una corrente elettrica applicata per produrre segnali elettronici a livello di singola molecola. Però, perché i quattro nucleotidi sono molto simili nelle loro strutture chimiche, non possono essere facilmente distinti usando questa tecnica. Così, la ricerca e lo sviluppo di una piattaforma di sequenziamento elettronico del DNA a singola molecola è l'area di indagine più attiva e ha il potenziale per produrre un sequenziatore di DNA portatile in grado di decifrare il genoma per la medicina personalizzata e la ricerca biomedica di base.
Un team di ricercatori della Columbia University, diretto dal Dr. Jingyue Ju (il Professore di Ingegneria Samuel Ruben-Peter G. Viele, Professore di Ingegneria Chimica e Farmacologia, Direttore del Centro per la tecnologia del genoma e l'ingegneria biomolecolare), con i colleghi del National Institute of Standards and Technology (NIST) guidati dal Dr. John Kasianowicz (Fellow dell'American Physical Society), hanno sviluppato un nuovo approccio per potenzialmente sequenziare il DNA in nanopori elettronicamente a livello di singola molecola con risoluzione a base singola. Questo lavoro, intitolato "PEG-Labeled Nucleotides and Nanopore Detection for Single Molecule DNA Sequencing by Synthesis" è ora disponibile nella rivista online ad accesso aperto, Rapporti scientifici , dal gruppo Nature Publication.
La strategia di sequenziamento per sintesi basata su nanopori riportata (Nano-SBS) può distinguere con precisione quattro basi di DNA rilevando 4 tag di dimensioni diverse rilasciati da nucleotidi modificati 5'-fosfato a livello di singola molecola per la determinazione della sequenza. Il principio di base della strategia Nano-SBS è descritto come segue. Poiché ogni analogo nucleotidico è incorporato nel filamento di DNA in crescita durante la reazione della polimerasi, il suo tag viene rilasciato dalla formazione del legame fosfodiestere. I tag entreranno in un nanoporo nell'ordine del loro rilascio, producendo firme uniche di blocco della corrente ionica a causa delle loro strutture chimiche distinte, determinando così la sequenza del DNA elettronicamente a livello di singola molecola con risoluzione su singola base. Come prova di principio, il team di ricerca ha attaccato quattro tag polimerici di diversa lunghezza al fosfato terminale del 2'-deossiguanosina-5'-tetrafosfato (un elemento costitutivo del DNA modificato) e ha dimostrato un'efficace incorporazione degli analoghi nucleotidici durante la reazione della polimerasi, nonché una discriminazione migliore della linea di base tra i quattro tag a livello di singola molecola in base alle loro firme di blocco della corrente ionica nanopori. Questo approccio accoppiato con la polimerasi attaccata ai nanopori in un formato array dovrebbe produrre una piattaforma elettronica Nano-SBS a singola molecola.
Nei lavori precedenti, il Centro di tecnologia del genoma e ingegneria biomolecolare presso la Columbia University, guidato dal Professor Ju e dal Dr. Nicholas J. Turro (William P. Schweitzer Professor of Chemistry), sviluppato una piattaforma di sequenziamento del DNA a quattro colori per sintesi (SBS) utilizzando terminatori reversibili di nucleotidi fluorescenti scindibili (NRT), che è concesso in licenza a Intelligent Bio-Systems, Inc., una società QIAGEN. SBS con NRT fluorescenti scindibili è l'approccio dominante utilizzato nei sistemi di sequenziamento del DNA di nuova generazione. Il Dr. Kasianowicz e il suo gruppo al NIST hanno aperto la strada allo studio dei nanopori per l'analisi di singole molecole. In precedenza hanno riferito che polimeri di diversa lunghezza, glicoli polietilenici (PEG), potrebbero essere distinti per i loro effetti unici sulle letture correnti nei nanopori di una proteina α-emolisina a livello di singola molecola e successivamente hanno sviluppato una teoria per il metodo. I loro risultati forniscono la prova del concetto per la spettrometria di massa a singola molecola. La combinazione del concetto SBS con i distinti tag elettronici rilevabili dai nanopori per etichettare i blocchi costitutivi del DNA ha portato allo sviluppo dell'approccio elettronico Nano-SBS a singola molecola che ha descritto l'attuale Rapporti scientifici articolo.
Come sottolinea l'autore principale Dr. Shiv Kumar, "La novità del nostro approccio risiede nella progettazione e nell'uso di quattro nucleotidi diversamente marcati, che in seguito all'incorporazione da parte della DNA polimerasi, rilasciano quattro tag di diverse dimensioni che si distinguono l'uno dall'altro a livello di singola molecola quando attraversano il nanoporo. Questo approccio supera i vincoli imposti dalle piccole differenze tra i quattro nucleotidi, una sfida che la maggior parte dei metodi di sequenziamento dei nanopori ha affrontato per decenni." Inoltre, la tecnica è abbastanza flessibile; con tag PEG come prototipi, altri tag chimici possono essere scelti per fornire una separazione ottimale in diversi sistemi di nanopori.
Con l'ulteriore sviluppo di questo approccio Nano-SBS, come l'uso di grandi array di proteine o nanopori solidi, questo sistema ha il potenziale per sequenziare accuratamente un intero genoma umano rapidamente e a basso costo, consentendo così di essere utilizzato nelle diagnosi mediche di routine.