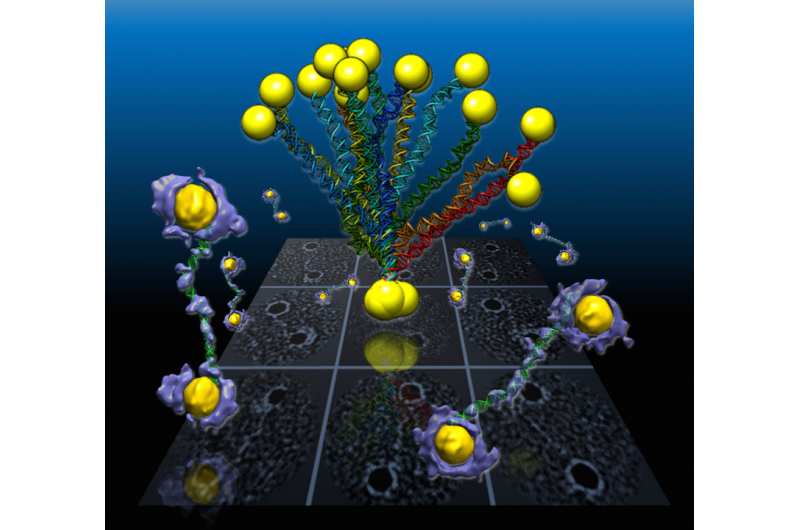
In uno studio condotto dal Berkeley Lab, segmenti flessibili di DNA a doppia elica collegati a nanoparticelle d'oro sono rivelati dalle mappe di densità 3-D (viola e gialla) ricostruite da campioni individuali utilizzando una tecnica sviluppata da Berkeley Lab chiamata tomografia elettronica a particelle individuali o IPET. Le proiezioni delle strutture sono mostrate nella griglia di sfondo. Credito:Berkeley Lab
Un team internazionale che lavora presso il Lawrence Berkeley National Laboratory (Berkeley Lab) del Dipartimento dell'Energia ha catturato le prime immagini 3D ad alta risoluzione da singoli segmenti di DNA a doppia elica attaccati alle due estremità alle nanoparticelle d'oro. Le immagini descrivono in dettaglio la struttura flessibile dei segmenti di DNA, che appaiono come corde per saltare su scala nanometrica.
Questa capacità di imaging unica, sperimentato dagli scienziati del Berkeley Lab, potrebbe aiutare nell'uso di segmenti di DNA come elementi costitutivi per dispositivi molecolari che funzionano come sistemi di somministrazione di farmaci su scala nanometrica, marcatori per la ricerca biologica, e componenti per memorie di computer e dispositivi elettronici. Potrebbe anche portare a immagini di importanti proteine rilevanti per la malattia che si sono dimostrate elusive per altre tecniche di imaging, e del processo di assemblaggio che forma il DNA da separato, singoli filamenti.
Le forme dei filamenti di DNA arrotolati, che erano racchiusi tra nanoparticelle d'oro a forma di poligono, sono stati ricostruiti in 3-D utilizzando una tecnica di microscopio elettronico all'avanguardia accoppiata con un processo di colorazione proteica e un software sofisticato che ha fornito dettagli strutturali alla scala di circa 2 nanometri, o due miliardesimi di metro.
"Non avevamo idea di come sarebbe stato il DNA a doppio filamento tra le particelle di nanooro, " ha detto Gang "Gary" Ren, uno scienziato del Berkeley Lab che ha guidato la ricerca. "Questa è la prima volta per visualizzare direttamente un singolo segmento di DNA a doppio filamento in 3-D, " ha detto. I risultati sono stati pubblicati nell'edizione del 30 marzo di Comunicazioni sulla natura .

Gang Ren (in piedi) e Lei Zhang hanno partecipato a uno studio presso la Molecular Foundry del Berkeley Lab che ha prodotto riproduzioni 3D di singoli campioni di segmenti di DNA a doppia elica attaccati a nanoparticelle d'oro. Credito:Roy Kaltschmidt/Berkeley Lab
Il metodo sviluppato da questo team, chiamata tomografia elettronica a particelle individuali (IPET), aveva precedentemente catturato la struttura 3-D di una singola proteina che svolge un ruolo chiave nel metabolismo del colesterolo umano. Catturando immagini 2D dello stesso oggetto da diverse angolazioni, la tecnica consente ai ricercatori di assemblare un'immagine 3D di quell'oggetto. Il team ha anche utilizzato la tecnica per scoprire la fluttuazione di un'altra ben nota proteina flessibile, immunoglobulina umana 1, che svolge un ruolo nel nostro sistema immunitario.
Per questo ultimo studio sulle nanostrutture del DNA, Ren ha utilizzato una tecnica di studio a fascio di elettroni chiamata microscopia crioelettronica (crio-EM) per esaminare campioni di DNA-nanooro congelati, e ha utilizzato l'IPET per ricostruire immagini 3D da campioni colorati con sali di metalli pesanti. Il team ha anche utilizzato strumenti di simulazione molecolare per testare le variazioni di forma naturali, chiamate "conformazioni, "nei campioni, e confrontato queste forme simulate con le osservazioni.
Ren ha spiegato che la dinamica naturalmente flessibile dei campioni, come un uomo che agita le braccia, non può essere completamente dettagliato con alcun metodo che utilizzi una media di molte osservazioni.
Un modo popolare per visualizzare i dettagli strutturali su scala nanometrica di campioni biologici delicati è trasformarli in cristalli e fulminarli con i raggi X, anche se questo non preserva la loro forma naturale e i campioni di DNA-nanooro in questo studio sono incredibilmente difficili da cristallizzare. Altre tecniche di ricerca comuni possono richiedere una raccolta di migliaia di oggetti quasi identici, visto al microscopio elettronico, per compilare un unico, struttura 3D media. Ma questa immagine 3D potrebbe non mostrare adeguatamente le fluttuazioni naturali della forma di un dato oggetto.
I campioni nell'ultimo esperimento sono stati formati da nanostrutture d'oro poligonali individuali, misura circa 5 nanometri di diametro, collegati a singoli filamenti di segmenti di DNA con 84 paia di basi. Le coppie di basi sono elementi costitutivi chimici di base che conferiscono al DNA la sua struttura. Ogni singolo segmento di DNA e nanoparticella d'oro si sono uniti naturalmente con un partner per formare il segmento di DNA a doppio filamento con una particella d'oro alle due estremità.
I campioni sono stati congelati al flash per preservare la loro struttura per lo studio con l'imaging crio-EM, e la distanza tra le due particelle d'oro nei singoli campioni variava da 20-30 nanometri in base alle diverse forme osservate nei segmenti di DNA. I ricercatori hanno utilizzato un microscopio crioelettronico presso la Molecular Foundry del Berkeley Lab per questo studio.
Hanno raccolto una serie di immagini inclinate degli oggetti macchiati, e ricostruito 14 mappe di densità elettronica che dettagliavano la struttura dei singoli campioni utilizzando la tecnica IPET. Hanno raccolto una dozzina di conformazioni per i campioni e hanno scoperto che le variazioni della forma del DNA erano coerenti con quelle misurate nei campioni crio-EM congelati. Le forme erano anche coerenti con i campioni studiati utilizzando altri metodi di imaging a base di elettroni e diffusione di raggi X, e con simulazioni al computer.
Mentre le ricostruzioni 3-D mostrano la struttura di base su scala nanometrica dei campioni, Ren ha detto che il prossimo passo sarà lavorare per migliorare la risoluzione alla scala sub-nanometrica.
"Anche in questo stato attuale cominciamo a vedere strutture 3D con una risoluzione da 1 a 2 nanometri, " ha detto. "Attraverso una migliore strumentazione e migliori algoritmi di calcolo, sarebbe promettente spingere la risoluzione a quella visualizzazione di una singola elica del DNA all'interno di una singola proteina".
La tecnica, Egli ha detto, ha già suscitato l'interesse di alcune importanti aziende farmaceutiche e ricercatori di nanotecnologie, e il suo team scientifico ha già in cantiere dozzine di progetti di ricerca correlati.
Negli studi futuri, i ricercatori potrebbero tentare di migliorare la risoluzione delle immagini per strutture complesse che incorporano più segmenti di DNA come una sorta di "origami di DNA, " ha detto Ren. I ricercatori sperano di costruire e caratterizzare meglio i dispositivi molecolari su scala nanometrica utilizzando segmenti di DNA che possono, Per esempio, immagazzinare e consegnare farmaci ad aree mirate del corpo.
"Il DNA è facile da programmare, sintetizzare e replicare, quindi può essere utilizzato come materiale speciale per autoassemblarsi rapidamente in nanostrutture e per guidare il funzionamento di dispositivi su scala molecolare, " ha detto. "Il nostro studio attuale è solo una prova del concetto per l'imaging di questo tipo di strutture di dispositivi molecolari".