
Estratto dal filmato time-lapse del rilevamento del miRNA in 1 ora utilizzando il test miRacles. Credito:progressi scientifici, doi:10.1126/sciadv.aau9443
I microRNA (miRNA) sono brevi RNA regolatori non codificanti che possono reprimere l'espressione genica post-trascrizionale e sono quindi sempre più utilizzati come biomarcatori di malattia. Rilevare i miRNA può essere arduo e costoso in quanto richiedono l'amplificazione, etichettatura e sonde radioattive. In un recente rapporto pubblicato su Progressi scientifici , Arun Richard Chandrasekaran e collaboratori presso l'Istituto RNA e Dipartimento di Scienze Biologiche, presso l'Università di Albany, Università statale di New York, riportato in un unico passaggio, test di rilevamento di microRNA non enzimatici utilizzando nanoswitch di DNA conformazionalmente non responsivi.
Gli scienziati hanno chiamato il test "miRacles, ' per abbreviare 'ciclo condizionale attivato da micro-RNA di interruttori ingegnerizzati'. Il saggio ha dimostrato la specificità del subattomole e del singolo nucleotide utilizzando una lettura dell'elettroforesi su gel di agarosio. Negli esperimenti, hanno rilevato microRNA cellulari da estrazioni di RNA ottenute dalla differenziazione dei muscoli su scala di nanogrammi. Gli scienziati hanno presentato una configurazione sperimentale conveniente per rilevare i miRNA in un lasso di tempo di un'ora per fornire un'alternativa convincente ai metodi esistenti di reazione a catena della polimerasi quantitativa (qPCR) e Northern blotting per quantificare i materiali genetici regolatori.
i miRNA possono regolare molti processi biologici durante il normale sviluppo fisiologico e la malattia influenzando la proliferazione cellulare, differenziazione e apoptosi in vivo. L'espressione dei miRNA può essere quantificata nei tessuti, cellule e fluidi corporei come biomarcatori stabili per eventi cellulari e diagnosi di malattie, sottolineando l'importanza della loro individuazione. Tuttavia, Il rilevamento dei miRNA è difficile a causa della scarsa abbondanza, piccole dimensioni e somiglianze in sequenza. Le biomolecole comprendono circa lo 0,01% del contenuto totale di RNA e le singole copie di mRNA per intervallo di cellule possono variare ampiamente. Inoltre, i miRNA in una famiglia possono differire di un singolo nucleotide, mentre specifici miRNA possono essere regolati durante la malattia e la normale funzione cellulare. Di conseguenza, le strategie di rilevazione per miRNA richiedono un'elevata specificità e la capacità di identificare correttamente poche molecole da un campione abbondante con molecole di RNA predominanti.
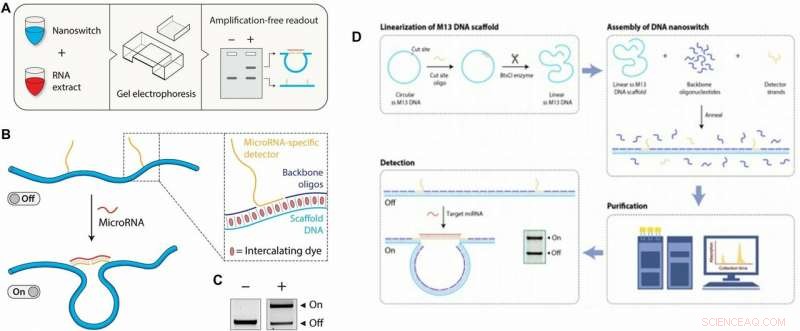
A sinistra:concetto e flusso di lavoro del test miRacles. (A) Flusso di lavoro del test miRacles:i nanoswitch di DNA personalizzati vengono miscelati con il campione di miRNA target, incubato, ed eseguire su un gel di agarosio per il rilevamento. (B) I nanointerruttori del DNA subiscono un cambiamento conformazionale da uno stato "off" lineare a uno stato "on" ad anello quando sono legati a un miRNA bersaglio. Riquadro:il nanoswitch è composto da un'impalcatura M13 a filamento singolo, oligonucleotidi della spina dorsale, ed estensioni a filamento singolo (rivelatori) complementari al miRNA bersaglio. I coloranti intercalanti intrinseci al processo di elettroforesi forniscono il segnale per visualizzare i nanointerruttori. (C) Le due conformazioni sono risolvibili in un gel di agarosio standard. A destra:esame approfondito del processo di costruzione del nanoswitch del DNA. (D) Linearizzazione:la preparazione di uno scaffold M13 lineare a singolo filamento procede ricottura di un oligonucleotide per consentire la scissione nel sito a doppio filamento da parte di un enzima di restrizione (BtsCI in questo caso). Assemblaggio:l'assemblaggio del DNA nanoswitch viene eseguito mescolando un eccesso 10x molare di oligonucleotidi della spina dorsale e filamenti del rivelatore (specifici per il bersaglio) all'M13 a filamento singolo e applicando una rampa di raffreddamento da 90˚C a 25˚C a 1˚C/min . Purificazione:per rimuovere gli oligonucleotidi in eccesso, viene utilizzato un metodo di cromatografia liquida. Rilevamento:per utilizzare i nanoswitch, vengono miscelati con una soluzione contenente bersagli di microRNA ed eseguiti su un gel per risolvere gli stati on e off come mostrato nell'inserto. Credito:progressi scientifici, doi:10.1126/sciadv.aau9443
I metodi tradizionali per il rilevamento dei miRNA includono il Northern blotting, reazione a catena della polimerasi a trascrizione inversa quantitativa (qRT-PCR), sequenziamento di nuova generazione e ibridazione basata su microarray per separare il segnale del miRNA dal rumore. Dei metodi indicati, Il Northern blotting può identificare direttamente i miRNA nativi, mentre altri si basano su metodi di etichettatura aggiuntivi o sull'amplificazione graduale, aggiungendo al costo, complessità e prestazioni di rilevamento. Per esempio, nanostrutture di DNA innovative possono essere utilizzate per il rilevamento di miRNA, dove vari gruppi di ricerca hanno precedentemente combinato nanostrutture con nanoparticelle, reazioni a catena di ibridazione e nanosheet di dicalcogenuro di metalli di transizione per consentire il processo.
Nel presente lavoro, Chandrasekaran et al. ha sviluppato un dispositivo basato sul DNA relativamente semplice per risolvere una complessa sfida biomedica. Nel saggio miRacles, gli scienziati hanno utilizzato un "reagente intelligente" composto da nanointerruttori di DNA progettati razionalmente per il rilevamento di miRNA nativi semplice ed economico senza utilizzare apparecchiature specializzate in laboratorio. I nanointerruttori del DNA sono stati originariamente progettati come strumenti per esperimenti di biofisica a singola molecola e successivamente riconosciuti per la loro capacità di rilevare e quantificare le interazioni biomolecolari mediante l'elettroforesi su gel. I precedenti sforzi di ricerca collaborativa dello stesso team di ricerca si sono concentrati sul rilevamento molecolare per quantificare i livelli di proteine e rilevare sequenze di DNA sintetiche come prova del concetto.
Il presente lavoro ha ampliato gli studi e i concetti preliminari per produrre il rilevamento e la quantificazione di miRNA multiplex pronti per l'utente. Gli scienziati hanno analizzato nanogrammi di estratti di RNA cellulare in un breve lasso di tempo utilizzando una configurazione sperimentale costruita utilizzando comuni forniture di laboratorio. Hanno progettato il nanoswitch del DNA come un duplex lineare che forma un anello in presenza della molecola di miRNA bersaglio. Per costruire il nanointerruttore, Chandrasekaran et al. ha utilizzato l'approccio dell'origami del DNA ibridando brevi oligonucleotidi complementari a uno scaffold di DNA a singolo filamento.
Hanno progettato due filamenti "rivelatori" distanti con sporgenze complementari a diversi segmenti del miRNA bersaglio. Quando il miRNA riconosciuto e legato al costrutto, l'interruttore è stato riconfigurato dallo stato "off" lineare allo stato "on" in loop. Hanno quantificato i due stati utilizzando l'elettroforesi su gel di agarosio standard per rilevare il segnale proveniente dal nanoswitch ad anello. Il segnale è stato amplificato solo da un singolo miRNA di interesse, i risultati sono stati confrontati favorevolmente con la tecnica del trasferimento di energia per risonanza di fluorescenza (FRET).

Validazione del saggio miRacles. (A) Specificità dei nanoswitch del DNA con rivelatori progettati per let-7b. La mancata corrispondenza di 1 nt tra i rilevatori e il miRNA target elimina il segnale. (B) Limite di rilevamento del dosaggio. NC, controllo negativo. AU, unità arbitrarie. (C) Andamento temporale del dosaggio per un target a bassa concentrazione. (D) Intervallo dinamico del dosaggio a diversi tempi di reazione. Credito:progressi scientifici, doi:10.1126/sciadv.aau9443.
Per convalidare il concetto, il team di ricercatori ha scelto un miRNA bersaglio let-7b a causa della sua famiglia altamente conservata di oltre una dozzina di miRNA correlati che variavano di uno o più nucleotidi. Questi miRNA erano adatti per il loro ruolo critico nelle funzioni biologiche e nella disregolazione in molteplici malattie umane. Per eliminare la diafonia e il rumore tra il nanoswitch e il target, che ha portato a una riduzione dell'intensità del segnale rispetto a una corrispondenza perfetta, gli scienziati hanno ridisegnato razionalmente i nanointerruttori. Per ottenere una specificità perfetta, hanno destabilizzato l'interazione sul lato contenente il mismatch. I risultati dello studio hanno mostrato un'elevata specificità del test così sviluppato, fornendo una risposta a una sfida chiave nella rilevazione dei miRNA che è culminata da un elevato rapporto segnale/rumore.
La bassa abbondanza di miRNA richiedeva anche un'elevata sensibilità di rilevamento, che gli scienziati hanno ottenuto ottimizzando il protocollo. Hanno quindi eseguito esperimenti simili per altre due varianti di miRNA (miR-15 a e miR-206), con conseguente subattomole ai livelli di rilevamento di singolo attomole. Ad esempio, per una bassa concentrazione di un campione target (6 pM), il segnale è aumentato per 4 ore, con pochi cambiamenti oltre quel lasso di tempo.
Rilevamento di miRNA dalla differenziazione delle cellule di mioblasti. (A) Schema che mostra le cellule di mioblasti, raccolto durante la coltivazione in GM e nei giorni di differenziazione 1 e 4, processati per produrre frazioni di RNA totali e piccole. Un marcatore precoce di differenziazione miogenico, miogo, e un marcatore di differenziazione miogenico tardivo, MHC, sono stati misurati mediante (B) Western blotting e (C) mediante immunocitochimica per confermare la differenziazione. Sia Myog che MHC erano up-regolati in DM1 e DM4. GAPDH (gliceraldeide-3-fosfato deidrogenasi) è servito come controllo in (B). (D) Chandrasekaran et al. rilevato miR-206 nei campioni differenziati con 50 ng di piccoli RNA e (E) con 500 ng di RNA totali. La quantificazione delle intensità del gel mostra una forte e progressiva up-regulation durante la differenziazione, simile in entrambi i campioni di RNA piccolo (F) e di RNA totale (G). Da campioni DM4, notiamo il rilevamento da un minimo di (H) 200 pg di piccoli RNA e (I) 500 pg di RNA totali. Credito:progressi scientifici, doi:10.1126/sciadv.aau9443. 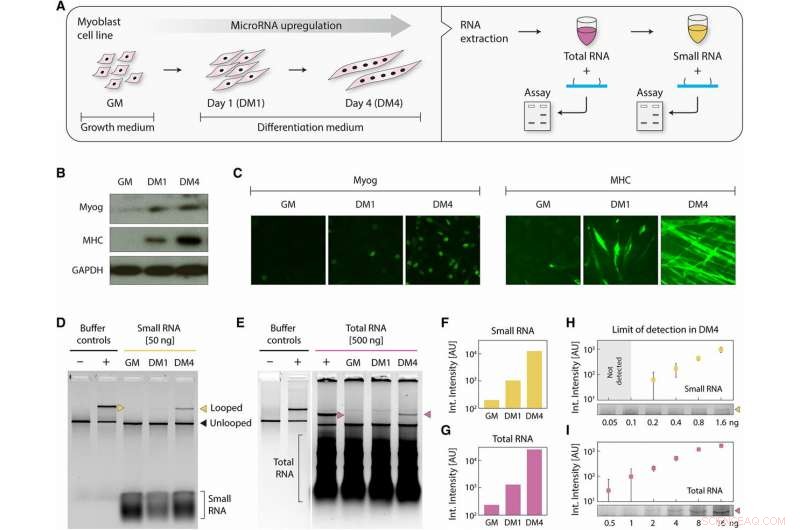
Poiché più mRNA possono anche alterare la loro espressione durante diversi stadi cellulari o di malattia, il fenomeno richiedeva capacità di rilevamento aggiuntive. Per ottenere ciò nel setup sperimentale, Chandrasekaran et al. ha utilizzato la programmabilità dei nanoswitch e ha sviluppato un sistema di multiplexing in grado di rilevare più miRNA dallo stesso campione. Hanno posizionato i fili del rivelatore nelle posizioni desiderate dell'impalcatura del DNA, con conseguente loop di dimensioni diverse quando legato al miRNA bersaglio. La dimensione dell'anello del nanoswitch ha quindi determinato la migrazione del gel, risultante in una banda unica sul gel per un rilevamento accurato. Per l'esperimento, gli scienziati hanno scelto quattro miRNA presenti nelle cellule muscolari; miR-206, miR-125b, miR-24 e miR-133-3p e un miRNA di controllo negativo; miR-39 specifico per la specie Caenorhabditis elegans.
Entro 50 ng da piccoli RNA, gli scienziati hanno rilevato i quattro miRNA a vari livelli di espressione, confermando l'assenza di rilevamento con il controllo negativo. La strategia di multiplexing ha permesso agli scienziati di confrontare direttamente i livelli di miRNA in un singolo campione, senza etichettatura o amplificazione. In totale, il lavoro ha fornito un ulteriore passo nella direzione di espandere il rendimento del test miRacles. La capacità può essere ampliata per ospitare anche più miRNA per switch.
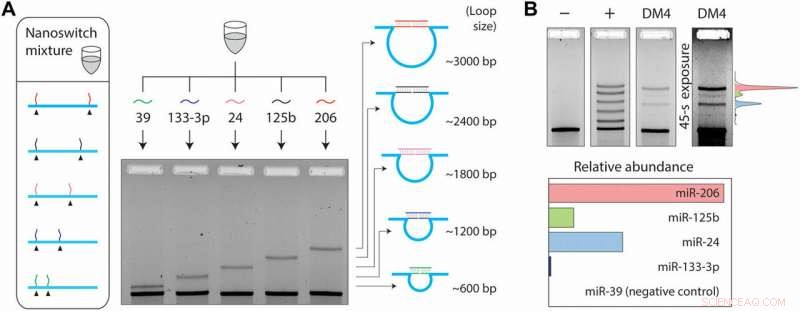
Configurazione multiplexing a cinque canali. (A) Il multiplexing consente il rilevamento di diversi miRNA con diverse dimensioni del ciclo. (B) Una miscela di nanoswitch multiplex mostra cinque bande con intensità simile in un controllo positivo costituito da tutti e cinque i miRNA target. In 50 ng di piccoli RNA DM4, quattro diversi miRNA vengono rilevati a vari livelli di espressione, con miR-39 (un miRNA specifico di C. elegans) non rilevato.
In questo modo, Chandrasekaran et al. sostanzialmente spostato in avanti dalla loro rilevazione preliminare proof-of-concept di sequenze di DNA sintetiche; Stabilire, caratterizzare e ottimizzare un saggio di rilevamento di miRNA pronto all'uso con estratti biologici. Il lavoro dimostrato è stato un primo esempio di utilizzo dei nanointerruttori del DNA per rilevare i miRNA da un vero campione biologico. Sebbene le prestazioni del dosaggio del miRNA siano state competitive rispetto ad altre tecniche comunemente usate, la selettività di 1 nucleotide vista nel presente lavoro era difficile da realizzare con i metodi esistenti. La sensibilità di miRacles ha anche superato il Northern blotting e i microarray. Il test potrebbe misurare i miRNA senza bisogno di amplificazione, con protocolli più semplici e senza l'errore aggiunto di un'ulteriore elaborazione del campione. Il protocollo ha semplicemente miscelato i nanoswitch con il liquido campione per l'elettroforesi su gel, per produrre risultati di alta qualità in laboratorio. Il lavoro di ricerca è potenzialmente trasferibile da campioni biologici a campioni clinici per diagnosticare e monitorare la malattia.
Ma ancora più importante, il presente lavoro si allinea con il concetto più ampio di scienza frugale; una visione promettente di una scienza conveniente che ha già prodotto soluzioni a basso costo per la centrifugazione del sangue e le tecniche di purificazione dell'acqua nell'ingegneria biomedica. Chandrasekaran et al. mirano a continuare a contribuire alla tendenza emergente nella scienza, interrompendo l'attuale rapporto costo/prestazioni per fornire un accesso su vasta scala a semplici metodi di rilevamento dei miRNA con reagenti intelligenti mix-and-read.
© 2019 Scienza X Rete