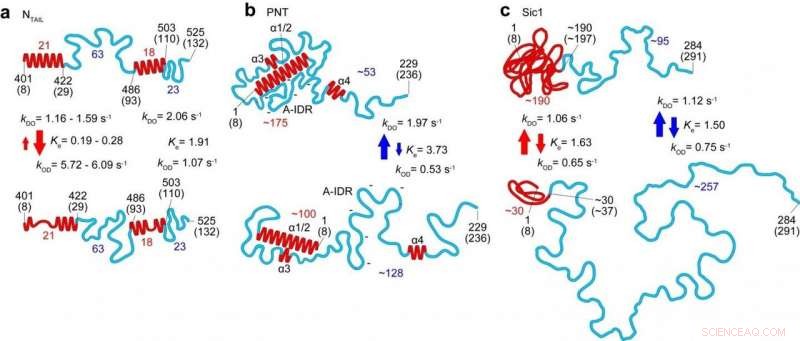
Caratteristiche strutturali e dinamiche di tre sfollati interni (N CODA , PNT e Sic1) rivelati dall'imaging HS-AFM. I pannelli superiore e inferiore corrispondono agli stati più ordinati e meno ordinati, rispettivamente. I numeri in rosso e blu rappresentano i numeri di amminoacidi contenuti nelle rispettive regioni piegate e regioni completamente disordinate, rispettivamente. Le frecce rosse e blu indicano la natura cinetica delle variazioni di altezza delle regioni piegate e la distanza da un capo all'altro di regioni completamente disordinate, rispettivamente. Credito:Università di Kanazawa
La nostra comprensione delle proteine biologiche non è sempre correlata a quanto siano comuni o importanti. La metà di tutte le proteine, molecole che svolgono un ruolo fondamentale nei processi cellulari, sono intrinsecamente disordinati, il che significa che molte delle tecniche standard per sondare le biomolecole non funzionano su di esse. Ora i ricercatori dell'Università di Kanazawa in Giappone hanno dimostrato che la loro tecnologia di microscopia a forza atomica ad alta velocità prodotta in casa può fornire informazioni non solo sulle strutture di queste proteine, ma anche sulla loro dinamica.
Comprendere come una proteina viene assemblata fornisce preziosi indizi sulle sue funzioni. Lo sviluppo della cristallografia proteica negli anni '30 e '50 ha mostrato per la prima volta diverse strutture proteiche, ma è diventato gradualmente evidente che una grande frazione di proteine manca di una singola struttura fissa che le rende intrattabili alla cristallografia a raggi X. Poiché sono troppo sottili per la microscopia elettronica, le uniche alternative praticabili per molte di queste proteine intrinsecamente disordinate (IDP) sono la risonanza magnetica nucleare e la diffusione dei raggi X a piccolo angolo. I dati raccolti da queste tecniche sono mediati su insiemi e quindi non forniscono alcuna indicazione chiara delle conformazioni delle singole proteine o della frequenza con cui si verificano. La microscopia a forza atomica, d'altra parte, è in grado di eseguire immagini biologiche con risoluzione su scala nanometrica ad alta velocità, quindi può catturare dinamiche e strutture proteiche.
In questo ultimo lavoro i ricercatori della Kanazawa University insieme a collaboratori in Giappone, Francia e Italia hanno applicato la tecnica allo studio di diversi IDP e hanno identificato i parametri che definiscono la forma, dimensione e lunghezza della catena delle regioni proteiche, così come una legge di potenza che correla la dimensione della proteina alla lunghezza della proteina, e una descrizione quantitativa dell'effetto della superficie della mica sulle dimensioni delle proteine. La dinamica delle conformazioni proteiche catturate grazie alle capacità ad alta velocità della tecnica ha rivelato globuli che appaiono e scompaiono, e trasformazioni tra conformazioni completamente non strutturate e leggermente piegate in segmenti lunghi fino a 160 amminoacidi.
Gli studi sulla nucleoproteina del virus del morbillo, in particolare, hanno aiutato a identificare non solo la forma e le dimensioni, ma anche le caratteristiche delle transizioni ordine-disordine nella regione responsabile del riconoscimento molecolare, che consente ai virus di identificare i fattori dell'ospite in modo che possano riprodursi. Potrebbero anche determinare strutture su scala più ampia della fosfoproteina del virus che non sono accessibili alla risonanza magnetica nucleare (che può solo dare un'indicazione delle distanze tra amminoacidi separati da meno di 2 nm). I ricercatori suggeriscono che la formazione di alcune forme compatte osservate potrebbe spiegare la resistenza alla proteolisi, la rottura delle proteine.
Nella loro relazione sul lavoro, i ricercatori sottolineano che, oltre a essere un potente strumento a sé stante, "Quando tutte le caratteristiche molecolari rivelate da HS-AFM sono combinate con la struttura locale piegata data da NMR, le informazioni combinate consentono una delineazione quantitativa dei caratteri strutturali e dinamici degli sfollati interni, in maniera più realistica rispetto alle immagini raffigurate singolarmente, come dimostrato per la PNT [fosfoproteina del virus del morbillo]."
Microscopia a forza atomica ad alta velocità
La microscopia a forza atomica è stata sviluppata negli anni '80 e ha portato la risoluzione su scala atomica ottenuta dalla microscopia a effetto tunnel (che ha vinto il Premio Nobel per la fisica nel 1986) a campioni non conduttivi. Funziona utilizzando un minuscolo cantilever con una punta su scala nanometrica all'estremità, che sente la superficie molto simile a una puntina di un disco in vinile o la tocca. Sia regolando l'altezza della punta o la frequenza di risonanza del tapping, le interazioni tra punta e superficie forniscono un segnale che può essere utilizzato per generare un'immagine.
While AFM images brought huge benefits to biological research, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.