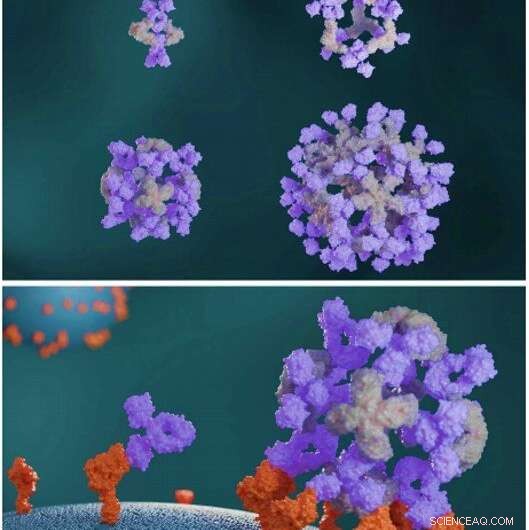
Le proteine progettate assemblano gli anticorpi in grandi architetture simmetriche. Le proteine progettate per raggruppare gli anticorpi (grigio chiaro) assemblano gli anticorpi (viola) in diverse architetture di nanogabbie (in alto). Le nanogabbie di anticorpi migliorano la segnalazione cellulare rispetto agli anticorpi liberi (in basso). Credito:Scienza, doi:10.1126/science.abd9994
In un nuovo rapporto ora pubblicato su Scienza , Robby Divine e un gruppo di ricerca interdisciplinare presso il dipartimento di biochimica, medicina rigenerativa, e vaccini e malattie infettive presso l'Università di Washington negli Stati Uniti e la School of Medicine, presso l'Università di Scienze Mediche di Teheran, Iran, ha condotto progetti computazionali di nanogabbie per assemblare anticorpi in architetture precise. Durante la costruzione, un componente strutturale ha formato un anticorpo o fusione del ligando Fc e il secondo design ha formato un omo-oligomero che si lega all'anticorpo per guidare l'assemblaggio della nanogabbia con diverse valenze e simmetria. Il team ipotizza come questo processo possa anche aumentare la neutralizzazione di uno pseudovirus; sindrome respiratoria acuta grave coronavirus-2 (SARS-COV-2) tramite anticorpi monoclonali α- SARS-COV-2 e proteine di fusione dell'enzima di conversione dell'angiotensina 2 (ACE-2) Fc.
Anticorpi nella ricerca medica
Gli anticorpi che si legano specificamente a bersagli di interesse svolgono un ruolo centrale nella ricerca biomedica e nella medicina. I ricercatori possono generare gruppi di anticorpi collegando geneticamente i frammenti di anticorpi per migliorare la segnalazione. Attualmente è difficile formare complessi di anticorpi con una varietà di architetture e valenze precise. In questo lavoro, Divino et al. proteine progettate in modo computazionale che hanno assemblato anticorpi in architetture precise con diverse valenze e simmetrie. Il team ha ipotizzato che tali progetti potrebbero guidare in modo robusto anticorpi arbitrari in nanogabbie omogenee e strutturalmente ben definite per effetti pronunciati sulla segnalazione cellulare. I ricercatori hanno progettato proteine per guidare l'assemblaggio di anticorpi arbitrari in assiemi simmetrici con strutture ben definite. Per questo, hanno rigidamente fuso insieme tre tipi di unità "building block" contenenti domini anticorpo Fc-binding, connettori a ripetizione elicoidale e moduli formanti oligomeri ciclici. Nella sua architettura, l'unità di legame Fc si è posizionata con il dimero dell'anticorpo C2, l'omo-oligomero ciclico ha formato il secondo asse di simmetria ciclica nella nanogabbia, e il connettore di ripetizione elicoidale collegava gli assi di simmetria dell'anticorpo e dell'omo-oligomero ciclico nell'orientamento corretto per formare le nanogabbie dell'anticorpo denominate AbC.
Progettazione della nanogabbia di anticorpi (AbC). (A) La geometria poliedrica è specificata. In senso orario da in alto a sinistra:icosaedrica, diedro, ottaedrico, e le geometrie tetraedriche sono mostrate. (B) Un modello di anticorpo Fc da hIgG1 è allineato a uno degli assi C2 (in questo caso, è mostrato un diedro D2). (C) I leganti Fc dell'anticorpo sono fusi a connettori ripetuti elicoidali che vengono quindi fusi alla subunità monomerica degli oligomeri ciclici elicoidali. Vengono campionate tutte le combinazioni di elementi costitutivi e giunzioni di elementi costitutivi (riquadro inferiore ombreggiato in grigio; i numeri tra parentesi si riferiscono al numero di elementi costitutivi disponibili). (D) Le fusioni tripartite vengono controllate per garantire il corretto allineamento degli assi di simmetria C2 Fc con quello dell'architettura poliedrica (nel caso della simmetria D2 mostrata qui, gli assi C2 devono intersecarsi con un angolo di 90°). (E) Le fusioni che superano i criteri geometrici vanno avanti con la riprogettazione della catena laterale, dove, Per esempio, gli amminoacidi sono ottimizzati per garantire che i residui del core-packing siano non polari e strettamente imballati e che i residui esposti al solvente siano polari. (F) Gli oligomeri che formano AbC progettati sono espressi battericamente, purificato, e assemblato con anticorpo Fc o IgG. Credito:Scienza, doi:10.1126/science.abd9994 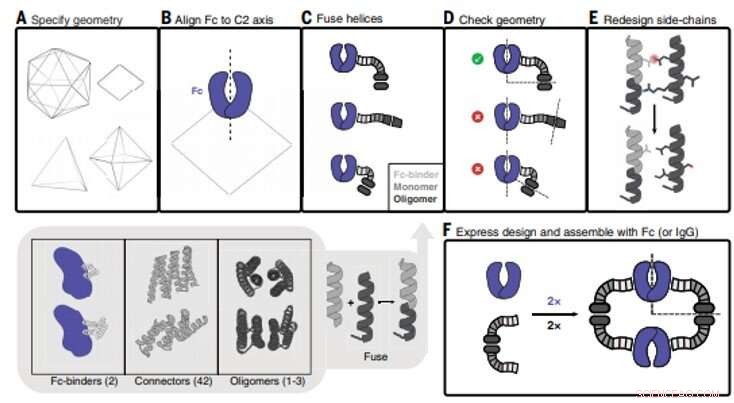
Per formare modelli di gabbia anticorpale (AbC), Divino et al. anticorpo rigidamente fuso molecole che legano il dominio costante a oligomeri ciclici utilizzando domini spaziatori elicoidali. Hanno facilitato il processo attraverso domini distanziatori elicoidali in modo che gli assi di simmetria dell'anticorpo dimerico e dell'oligomero ciclico potessero trovarsi in orientamenti che hanno generato diverse architetture diedriche o poliedriche. Gli scienziati hanno ottimizzato le regioni di giunzione tra i blocchi di costruzione collegati per piegarsi alle strutture progettate. L'approccio alla fusione si basava su ampi set di elementi costitutivi con molti possibili siti di fusione per elemento costitutivo come criteri geometrici rigorosi per formare l'architettura simmetrica desiderata. Il team ha utilizzato un metodo computazionale per la progettazione della gabbia anticorpale per generare diedri, tetraedrico, AbC ottaedrici e icosaedrici e hanno utilizzato una convenzione di denominazione per descrivere le architetture finali delle nanogabbie. Divino et al. successivamente hanno espresso geni sintetici codificanti i disegni in colture batteriche di Escherichia coli. Ad esempio, i progetti di successo includevano il decaedro D-2 (tre disegni), architetture T-32 (un disegno) e 152 icosaedrica (due modelli) contenenti due, sei, 12 o 30 anticorpi, rispettivamente. Hanno caratterizzato gli Fc AbC utilizzando la diffusione di raggi X a piccolo angolo e la microscopia elettronica. Le nanogabbie ricostruite erano in stretto accordo con i modelli di progettazione computazionale. Per valutare la stabilità delle nanogabbie, Divino et al. hanno utilizzato letture di diffusione della luce dinamica per ottenere una stabilità incoraggiante per consentire la successiva caratterizzazione del loro impatto biologico.
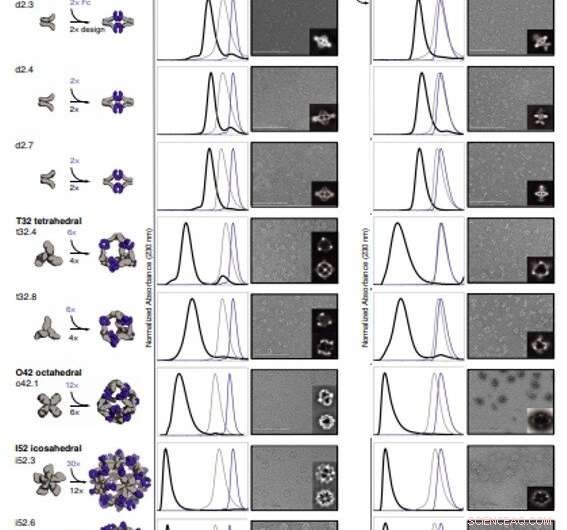
Caratterizzazione strutturale degli AbC. (A) Modelli di progettazione, con l'anticorpo Fc (viola) e oligomeri che formano AbC (grigio). (B) Sovrapposizione di tracce rappresentative SEC di assemblaggio formate mescolando disegno e Fc (nero) con quelle dei singoli componenti in grigio (disegno) o viola (Fc). (C) immagini EM con medie di classe 2D senza riferimento nel riquadro; tutti i dati provengono da NS-EM, ad eccezione dei modelli o42.1 e i52.3 (crio-EM). (D ed E) Micrografie rappresentative SEC (D) e NS-EM con medie di classe 2D senza riferimento (E) delle stesse gabbie di anticorpi progettate assemblate con IgG1 umane complete (con le 2 regioni Fab intatte). In tutti i casi EM mostrati in (C) e (E), le assemblee sono state prima purificate tramite SEC, e le frazioni corrispondenti al picco più a sinistra sono state raggruppate e utilizzate per l'imaging per rimuovere qualsiasi disegno in eccesso o componente di Ig. Barre della scala, 200 nm. Credito:Scienza, doi:10.1126/science.abd9994
Effetti sulla segnalazione cellulare
Gli AbC progettati hanno fornito una piattaforma generale per comprendere l'effetto della valenza e della geometria dell'impegno del recettore sull'attivazione della via di segnalazione. L'ampia gamma di anticorpi leganti il recettore e ligandi naturali formati con il metodo AbC sviluppato in questo lavoro ha permesso di sondare prontamente e sistematicamente l'effetto della geometria e della valenza dell'associazione della subunità del recettore sulla segnalazione cellulare per quasi tutte le vie. Per esplorare la praticità di questo approccio, Divino et al. anticorpi assemblati o fusioni di ligando Fc che mirano a una varietà di percorsi di segnalazione in gabbie di anticorpi (AbC) e ne hanno studiato l'impatto sulla segnalazione. Ad esempio, gli AbC, formato con un anticorpo diretto contro il recettore della morte indotta dall'apoptosi (morte cellulare) di linee cellulari tumorali che fino a quel momento erano rimaste inalterate dall'anticorpo solubile o dal ligando nativo. Inoltre, l'assemblaggio di fusioni Fc o anticorpi in AbCs, ha permesso una maggiore segnalazione della via dell'angiopoietina, Segnalazione CD-40 e proliferazione delle cellule T. La formazione di AbC ha inoltre consentito la neutralizzazione di uno pseudovirus in vitro, come la sindrome respiratoria acuta grave coronavirus 2.
Ricostruzioni 3D di AbCs formati con Fc. I modelli di progettazione computazionale (rappresentazione dei cartoni animati) di ciascun AbC sono inseriti nella densità 3D determinata sperimentalmente da EM. Ogni nanogabbia viene visualizzata lungo un asse di simmetria non occupato (a sinistra) e ruotata per guardare in basso uno degli assi di simmetria C2 occupati da Fc (a destra). Le ricostruzioni tridimensionali di o42.1 e i52.3 provengono dall'analisi cryo-EM, tutti gli altri provengono da NS-EM. Credito:Scienza, doi:10.1126/science.abd9994 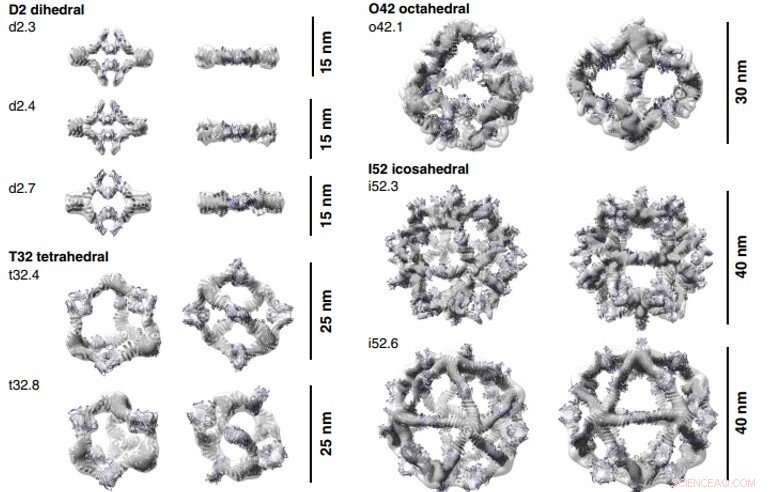
Il metodo descritto in questo lavoro, è andato oltre i precedenti sforzi di progettazione computazionale per creare nanomateriali proteici che integrano forma e funzione. Gli AbC hanno quindi utilizzato anticorpi come componenti sia strutturali che funzionali per ottenere un'ampia gamma di geometrie e orientamenti. Questa strategia è applicabile alla progettazione di vaccini con nanogabbie assemblate con glicoproteine virali utilizzando componenti che terminano in domini di legame della glicoproteina per massimizzare la vicinanza dei siti attivi. In questo modo, Robby Divine e colleghi hanno progettato più proteine che formano una gabbia di anticorpi per raggruppare accuratamente qualsiasi anticorpo che si lega alle proteine in nanogabbie attraverso valenza e geometria controllate. La squadra ne ha usati due, sei e 12 o 30 anticorpi all'interno degli AbC semplicemente mescolando l'anticorpo con la corrispondente proteina progettata senza ulteriori modifiche covalenti. Gli scienziati hanno aggiunto anticorpi che legano il recettore o neutralizzano i virus negli ABC per migliorare la loro attività biologica in vari sistemi cellulari. Il team si aspetta che questo risultato per l'assemblaggio rapido di anticorpi all'interno di nanogabbie ordinate senza modifiche covalenti abbia ampie applicazioni nella ricerca e nella medicina.

Gli AbC attivano le vie di segnalazione dell'apoptosi e dell'angiogenesi. (A e B) La caspasi-3/7 è attivata da AbCs formati con l'anticorpo a-DR5 (A), ma non l'anticorpo libero, nelle cellule tumorali renali RCC4 (B). (C e D) a-DR5 AbC (C), ma non controlli Fc AbC (D), ridurre la vitalità cellulare 4 giorni dopo il trattamento. (E) Gli AbC a-DR5 riducono la vitalità 6 giorni dopo il trattamento. (F e G) o42.1 a-DR5 AbCs migliorano la scissione di PARP, un marker di segnalazione apoptotica; (G) è una quantificazione di (F) relativa al controllo PBS. (H) The F-domain from angiopoietin-1 was fused to Fc (A1F-Fc) and assembled into octahedral (o42.1) and icosahedral (i52.3) AbCs. (I) Representative Western blots show that A1F-Fc AbCs, but not controls, increase pAKT and pERK1/2 signals. (J) Quantification of (I):pAKT quantification is normalized to o42.1 A1F-Fc signaling (no pAKT signal in the PBS control); pERK1/2 is normalized to PBS. (K) A1F-Fc AbCs increase vascular stability after 72 hours. (Left) Quantification of vascular stability compared with PBS. (Right) Representative images; scale bars, 100 mm. All error bars represent means ± SEM; means were compared using analysis of variance and Dunnett post-hoc tests (tables S8 and S9). *P ≤ 0.05; **P ≤ 0.01; ***P ≤ 0.001; ****P ≤ 0.0001 Credit:Science, doi:10.1126/science.abd9994
© 2021 Science X Network