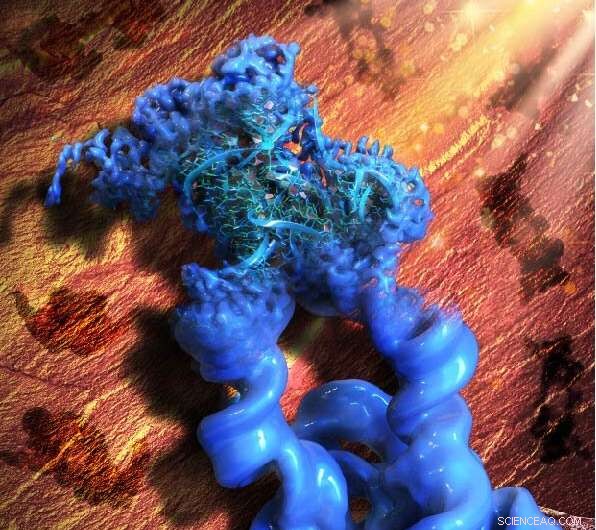
Questa illustrazione è ispirata alla pittura rupestre paleolitica nella grotta di Lascaux, a significare l'acronimo del nostro metodo, ROCK. In senso figurato, i modelli dell'arte rupestre sullo sfondo (marrone) sono le proiezioni 2D del costrutto dimerico ingegnerizzato dell'introne del gruppo Tetrahymena I, mentre l'oggetto principale nella parte anteriore (blu) è la mappa crio-EM 3D ricostruita del dimero, con un monomero a fuoco e raffinato all'alta risoluzione che ha permesso ai collaboratori di costruire un modello atomico dell'RNA. Credito:Wyss Institute presso l'Università di Harvard
Viviamo in un mondo fatto e gestito dall'RNA, il fratello altrettanto importante della molecola genetica del DNA. In effetti, i biologi evoluzionisti ipotizzano che l'RNA sia esistito e si sia auto-replicato anche prima della comparsa del DNA e delle proteine da esso codificate. Avanti veloce fino agli esseri umani moderni:la scienza ha rivelato che meno del 3% del genoma umano viene trascritto in molecole di RNA messaggero (mRNA) che a loro volta vengono tradotte in proteine. Al contrario, l'82% di esso viene trascritto in molecole di RNA con altre funzioni, molte delle quali rimangono ancora enigmatiche.
Per capire cosa fa una singola molecola di RNA, la sua struttura 3D deve essere decifrata a livello dei suoi atomi costituenti e dei suoi legami molecolari. I ricercatori hanno studiato di routine il DNA e le molecole proteiche trasformandole in cristalli regolarmente imballati che possono essere esaminati con un raggio di raggi X (cristallografia a raggi X) o onde radio (risonanza magnetica nucleare). Tuttavia, queste tecniche non possono essere applicate alle molecole di RNA con quasi la stessa efficacia perché la loro composizione molecolare e flessibilità strutturale impediscono loro di formare facilmente cristalli.
Ora, una collaborazione di ricerca guidata dal membro della Wyss Core Faculty Peng Yin, Ph.D. presso il Wyss Institute for Biologically Inspired Engineering presso l'Università di Harvard, e Maofu Liao, Ph.D. presso la Harvard Medical School (HMS), ha riportato un approccio fondamentalmente nuovo allo studio strutturale delle molecole di RNA. ROCK, come viene chiamato, utilizza una tecnica nanotecnologica dell'RNA che gli consente di assemblare più molecole di RNA identiche in una struttura altamente organizzata, che riduce significativamente la flessibilità delle singole molecole di RNA e ne moltiplica il peso molecolare. Applicato a noti RNA modello con diverse dimensioni e funzioni come parametri di riferimento, il team ha dimostrato che il loro metodo consente l'analisi strutturale delle subunità di RNA contenute con una tecnica nota come microscopia crioelettronica (crio-EM). Il loro progresso è riportato in Metodi della natura .
"ROCK sta infrangendo gli attuali limiti delle indagini strutturali sull'RNA e consente di sbloccare strutture 3D di molecole di RNA a cui è difficile o impossibile accedere con i metodi esistenti e con una risoluzione quasi atomica", ha affermato Yin, che insieme a Liao ha condotto lo studio . "Ci aspettiamo che questo progresso rinvigorisca molte aree della ricerca fondamentale e dello sviluppo di farmaci, compreso il fiorente campo della terapia dell'RNA". Yin è anche leader della Molecular Robotics Initiative del Wyss Institute e professore presso il Dipartimento di Biologia dei Sistemi presso HMS.
Ottenere il controllo sull'RNA
Il team di Yin al Wyss Institute ha sperimentato vari approcci che consentono alle molecole di DNA e RNA di autoassemblarsi in grandi strutture basate su principi e requisiti diversi, inclusi mattoni di DNA e origami di DNA. Hanno ipotizzato che tali strategie potrebbero essere utilizzate anche per assemblare molecole di RNA presenti in natura in complessi circolari altamente ordinati in cui la loro libertà di flettersi e muoversi è fortemente limitata dal collegamento specifico tra loro. Molti RNA si ripiegano in modi complessi ma prevedibili, con piccoli segmenti che si accoppiano tra loro. Il risultato è spesso un "nucleo" stabilizzato e "anse staminali" che si gonfiano verso la periferia.
"Nel nostro approccio installiamo 'kissing loop' che collegano diversi stem-loop periferici appartenenti a due copie di un RNA identico in un modo che consente la formazione di un anello stabilizzato complessivo, contenente più copie dell'RNA di interesse", ha affermato Di Liu, Ph.D., uno dei due primi autori e un Postdoctoral Fellow nel gruppo di Yin. "Abbiamo ipotizzato che questi anelli di ordine superiore potessero essere analizzati ad alta risoluzione da cryo-EM, che era stato applicato alle molecole di RNA con il primo successo."
Rappresentazione dell'RNA stabilizzato
In cryo-EM, molte singole particelle vengono congelate a temperature criogeniche per impedire ulteriori movimenti, quindi visualizzate con un microscopio elettronico e l'aiuto di algoritmi computazionali che confrontano i vari aspetti delle proiezioni 2D della superficie di una particella e ne ricostruiscono l'architettura 3D . Peng e Liu hanno collaborato con Liao e il suo ex studente laureato François Thélot, Ph.D., l'altro co-primo autore dello studio. Liao con il suo gruppo ha dato importanti contributi al campo crio-EM in rapida evoluzione e all'analisi sperimentale e computazionale di singole particelle formate da proteine specifiche.
"Cryo-EM presenta grandi vantaggi rispetto ai metodi tradizionali nel vedere dettagli ad alta risoluzione di molecole biologiche tra cui proteine, DNA e RNA, ma le piccole dimensioni e la tendenza al movimento della maggior parte degli RNA impediscono la determinazione delle strutture dell'RNA. Il nostro nuovo metodo di assemblaggio di multimeri di RNA risolve questi due problemi allo stesso tempo, aumentando le dimensioni dell'RNA e riducendone il movimento", ha affermato Liao, che è anche professore associato di biologia cellulare presso l'HMS. "Il nostro approccio ha aperto le porte alla determinazione rapida della struttura di molti RNA mediante cryo-EM". L'integrazione della nanotecnologia dell'RNA e degli approcci cryo-EM ha portato il team a chiamare il loro metodo "RNA oligomerization-enabled cryo-EM tramite l'installazione di kissing loop" (ROCK).
Per fornire una prova di principio per ROCK, il team si è concentrato su un grande RNA di introne di Tetrahymena, un organismo unicellulare, e un piccolo RNA di introne di Azoarcus, un batterio che fissa l'azoto, nonché il cosiddetto riboswitch FMN . Gli RNA di introni sono sequenze di RNA non codificanti sparse nelle sequenze di RNA appena trascritti e devono essere "unite" per generare l'RNA maturo. Il riboswitch FMN si trova negli RNA batterici coinvolti nella biosintesi dei metaboliti della flavina derivati dalla vitamina B2. Dopo aver legato uno di essi, il flavin mononucleotide (FMN), cambia la sua conformazione 3D e sopprime la sintesi del suo RNA madre.
"L'assemblaggio dell'introne del gruppo I di Tetrahymena in una struttura ad anello ha reso i campioni più omogenei e ha consentito l'uso di strumenti computazionali che sfruttano la simmetria della struttura assemblata. Sebbene il nostro set di dati sia di dimensioni relativamente modeste, i vantaggi innati di ROCK ci hanno permesso risolvere la struttura con una risoluzione senza precedenti", ha affermato Thélot. "Il nucleo dell'RNA è risolto a 2,85 Å [un Ångström è un dieci miliardi (USA) di metro e la metrica preferita usata dai biologi strutturali], rivelando caratteristiche dettagliate delle basi nucleotidiche e della spina dorsale dello zucchero. avrebbe potuto arrivarci senza ROCK, o almeno non senza risorse considerevolmente maggiori".
Cryo-EM è anche in grado di catturare molecole in diversi stati se, ad esempio, cambiano la loro conformazione 3D come parte della loro funzione. Applicando ROCK all'introne di Azoarcus RNA e al riboswitch FMN, il team è riuscito a identificare le diverse conformazioni attraverso le quali l'introne di Azoarcus passa durante il suo processo di autogiunzione e a rivelare la relativa rigidità conformazionale del sito di legame del ligando del riboswitch FMN .
"Questo studio di Peng Yin e dei suoi collaboratori mostra elegantemente come la nanotecnologia dell'RNA può funzionare come un acceleratore per far avanzare altre discipline. Essere in grado di visualizzare e comprendere le strutture di molte molecole di RNA presenti in natura potrebbe avere un enorme impatto sulla nostra comprensione di molte molecole biologiche e patologiche processi attraverso diversi tipi di cellule, tessuti e organismi e persino consentire nuovi approcci allo sviluppo di farmaci", ha affermato il direttore fondatore di Wyss Donald Ingber, M.D., Ph.D. + Esplora ulteriormente