I componenti molecolari dei computer potrebbero rappresentare una nuova rivoluzione informatica e aiutarci a creare computer più economici, più veloci, più piccoli e più potenti. Tuttavia i ricercatori faticano a trovare modi per assemblarli in modo più affidabile ed efficiente.
Per contribuire a raggiungere questo obiettivo, gli scienziati dell'Istituto di fisica dell'Accademia ceca delle scienze hanno studiato le possibilità dell'autoassemblaggio di macchine molecolari basandosi su soluzioni affinate dall'evoluzione naturale e utilizzando la sinergia con l'attuale produzione di chip.
Esiste un limite alla miniaturizzazione degli attuali chip per computer basati sul silicio. L’elettronica molecolare, che utilizza interruttori e memorie delle dimensioni di una singola molecola, potrebbe rivoluzionare le dimensioni, la velocità e le capacità dei computer, riducendo al contempo il loro crescente consumo energetico, ma la loro produzione di massa rappresenta una sfida. La nanofabbricazione e l’assemblaggio dei componenti su larga scala, con pochi difetti e accessibili rimangono sfuggenti. L'ispirazione presa dalla natura vivente potrebbe cambiare questo status quo.
Piccoli prototipi di circuiti molecolari composti da una coppia di molecole vengono attualmente prodotti mediante la microscopia a scansione di sonda, che li manipola una molecola alla volta mediante un cantilever macroscopico lento e pesante.
Prokop Hapala, che ha condotto lo studio pubblicato su ACS Nano , lo paragona alla costruzione di un delicato mosaico utilizzando un'enorme gru, una tessera alla volta. L’autoassemblaggio potrebbe risolvere questo problema, ma crea altre sfide. Ad esempio, come possiamo produrre una varietà di strutture quando solo una piccola quantità di informazioni strutturali può essere codificata nelle interazioni tra pochi gruppi funzionali?
I ricercatori dell’Istituto di fisica dell’Accademia ceca delle scienze si sono ispirati alla natura, dove i componenti funzionali e strutturali sono disaccoppiati in modelli polimerici come il DNA o l’RNA. Lì, gli zuccheri-fosfati rappresentano l'impalcatura e le basi azotate, legate da legami idrogeno, forniscono l'immagazzinamento delle informazioni.
Grazie a questi legami, questi polimeri informativi possono autoassemblarsi in forme complesse e guidare l’autoreplicazione o la sintesi di altre molecole più piccole. Questo approccio è già stato utilizzato negli "origami di DNA", che possono produrre molecole complesse con le forme e le funzioni desiderate. Ma come possiamo ampliare il processo e ottenere una maggiore varietà?
"Le coppie di basi del DNA conosciute, che ingenuamente si può pensare siano la scelta migliore, non possono essere utilizzate così come sono", spiega Paolo Nicolini, uno degli autori. "Funzionano benissimo nella cellula, ma ciò è dovuto all'ambiente e al resto del meccanismo cellulare. In condizioni compatibili con la nanofabbricazione, semplicemente non sono abbastanza selettivi."
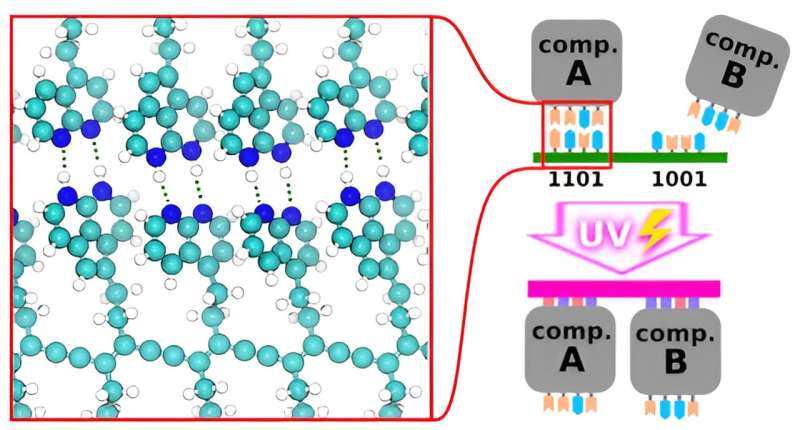
Mithun Manikandan, Paolo Nicolini e Prokop Hapala hanno deciso di combinare le possibilità offerte dall'origami del DNA e dalla fotolitografia per disporre strutture complesse di chip contemporanei. Ciò potrebbe aprire la strada alla produzione di massa di circuiti molecolari rivoluzionari integrati con la tecnologia contemporanea di produzione di chip, qualcosa che potrebbe consentire una transizione graduale dagli attuali macchinari informatici al livello successivo.
Per consentire ciò, i ricercatori hanno proposto di sostituire la struttura principale degli zuccheri e dei fosfati con diacetilene fotosensibile. Hanno utilizzato simulazioni dettagliate per individuare gruppi terminali complementari legati a idrogeno che avrebbero favorito l'autoassemblaggio su un reticolo nelle condizioni utilizzate nella produzione di chip.
I derivati del diacetilene sono stati utilizzati come struttura portante perché possono polimerizzare in modo efficiente in queste condizioni quando innescati mediante luce UV o iniezione di elettroni, e unità analoghe alle basi DNA/RNA (le "lettere" del codice genetico) sono state studiate in silico come gruppi terminali guidando l'assemblaggio dei componenti nelle forme previste.
L'obiettivo era trovare coppie complementari, in cui due unità si legano in modo affidabile l'una all'altra e non ad altre unità:questa caratteristica, ancora una volta analoga a come funziona il DNA, consentirebbe la creazione di schemi circuitali complessi deterministici. I ricercatori hanno scoperto che le unità contenenti gruppi terminali donatori di idrogeno puro erano particolarmente adatte. Sono state trovate sedici unità candidate promettenti, aprendo la strada alla ricerca sperimentale e ad eventuali applicazioni industriali.
I risultati hanno implicazioni interessanti per il calcolo del DNA e gli analoghi artificiali del DNA. La maggior parte degli alfabeti di quattro lettere riscontrati nello screening si trovavano in una regione molto ristretta di energie di legame di 15-25 kcal/mol e si basavano tutti su un piccolo sottoinsieme dei gruppi terminali testati.
Sebbene solo un piccolo sottoinsieme del possibile spazio delle lettere possa essere testato con elevata precisione, ciò suggerisce che l’alfabeto del DNA potrebbe non essere solo il risultato di un “incidente congelato nel tempo”, ma avrebbe potuto essere un’opzione stabile ed energeticamente favorevole. Nello spazio testato non sono stati trovati alfabeti di sei lettere, ma nuovi meccanismi di selettività e legami non covalenti diversi dai legami idrogeno (come i legami alogeni) potrebbero potenzialmente abilitarli. In modo simile si potrebbero testare le possibilità degli analoghi terapeutici e farmaceutici del DNA.
Questo lavoro migliorerà ulteriormente la disponibilità sintetica delle molecole e supererà le limitazioni sperimentali. Anche se la maggior parte di noi probabilmente legge questo articolo su macchine che si affidano a transistor a base di silicio, presto potremmo iniziare a passare senza problemi a macchine che utilizzano in parte la nanoelettronica molecolare. Questo lavoro rappresenta un altro passo verso tale futuro.
Ulteriori informazioni: Mithun Manikandan et al, Progettazione computazionale di modelli polimerici fotosensibili per guidare la nanofabbricazione molecolare, ACS Nano (2024). DOI:10.1021/acsnano.3c10575
Informazioni sul giornale: ACS Nano
Fornito dall'Accademia ceca delle scienze