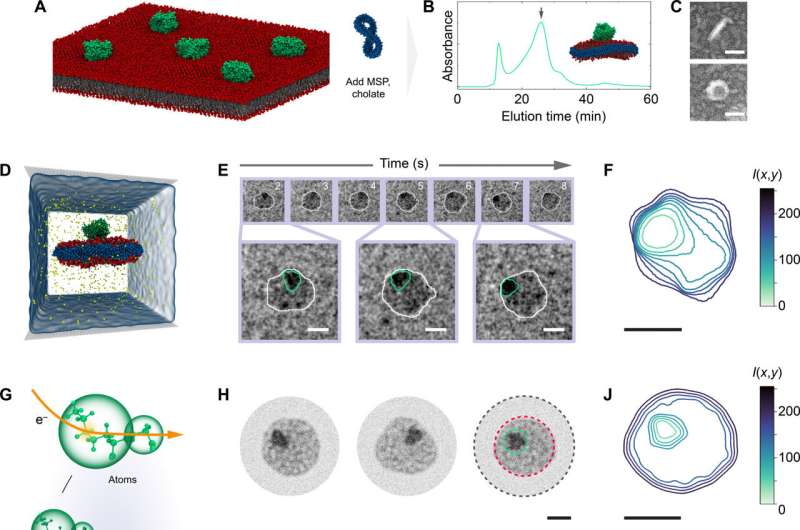
In una prima dimostrazione di "videografia elettronica", i ricercatori hanno catturato un'immagine microscopica in movimento della delicata danza tra proteine e lipidi presenti nelle membrane cellulari. La tecnica può essere utilizzata per studiare la dinamica di altre biomolecole, liberandosi dai vincoli che hanno limitato la microscopia alle immagini fisse di molecole fisse, affermano i ricercatori e i collaboratori di Urbana-Champaign dell'Università dell'Illinois presso il Georgia Institute of Technology.
"Stiamo andando oltre l'acquisizione di singole istantanee, che forniscono struttura ma non dinamica, per registrare continuamente le molecole nell'acqua, il loro stato nativo", ha affermato il leader dello studio Qian Chen, professore di scienza e ingegneria dei materiali dell'Illinois. "Possiamo davvero vedere come le proteine cambiano la loro configurazione e, in questo caso, come l'intera struttura autoassemblata proteina-lipide fluttua nel tempo."
I ricercatori hanno riportato la loro tecnica e i risultati sulla rivista Science Advances .
Le tecniche di microscopia elettronica riproducono immagini su scala molecolare o atomica, producendo immagini dettagliate su scala nanometrica. Tuttavia, spesso si basano su campioni che sono stati congelati o fissati sul posto, lasciando agli scienziati il compito di provare a dedurre come le molecole si muovono e interagiscono, come se tentassero di mappare la coreografia di una sequenza di danza da un singolo fotogramma di un film.
"Questa è la prima volta che osserviamo una proteina su scala individuale e non l'abbiamo congelata o etichettata", ha affermato il professore della Georgia Tech Aditi Das, autore corrispondente dello studio. "Di solito, dobbiamo cristallizzare o congelare una proteina, il che pone delle sfide nel catturare immagini ad alta risoluzione di proteine flessibili. In alternativa, alcune tecniche utilizzano un tag molecolare che tracciamo, invece di osservare la proteina stessa. In questo studio stiamo osservando la proteina così com'è, comportandosi in un ambiente liquido e osservando come i lipidi e le proteine interagiscono tra loro."
I ricercatori hanno ottenuto la videografia combinando un nuovo metodo di microscopia elettronica a trasmissione a base acquosa con una modellazione computazionale dettagliata a livello di atomo. La tecnica a base d’acqua prevede l’incapsulamento di goccioline su scala nanometrica nel grafene in modo che possano resistere al vuoto in cui opera il microscopio. Il confronto dei dati video risultanti con i modelli molecolari, che mostrano come le cose dovrebbero muoversi in base alle leggi della fisica, aiuta i ricercatori non solo a interpretare, ma anche a convalidare i loro dati sperimentali.
"Attualmente, questo è davvero l'unico modo sperimentale per filmare questo tipo di movimento nel tempo", ha detto John W. Smith, il primo autore dell'articolo, che ha completato il lavoro mentre era studente laureato all'Illinois. "La vita è allo stato liquido ed è in movimento. Stiamo cercando di arrivare ai dettagli più fini di questa connessione in modo sperimentale."
Per il nuovo studio, la prima dimostrazione pubblicata della tecnica della videografia elettronica, i ricercatori hanno esaminato i dischi su scala nanometrica delle membrane lipidiche e il modo in cui interagivano con le proteine normalmente presenti sulla superficie o incorporate nelle membrane cellulari.
"Le proteine di membrana sono all'interfaccia tra le cellule e tra l'interno e l'esterno della cellula, controllando ciò che entra ed esce", ha detto Smith. "Sono prevalentemente obiettivi della medicina; sono coinvolti in tutti i tipi di processi come il modo in cui i nostri muscoli si contraggono, il modo in cui funziona il nostro cervello, il riconoscimento immunitario; e tengono insieme cellule e tessuti. E tutta la complessità di come funziona una proteina di membrana deriva da non solo la sua struttura, ma anche il modo in cui percepisce i lipidi che lo circondano."
La videografia elettronica ha permesso ai ricercatori di vedere non solo come si muoveva l'intero assemblaggio lipide-proteico, ma anche la dinamica di ciascun componente. I ricercatori hanno scoperto che all'interno del nanodisco c'erano regioni distinte e che c'erano più fluttuazioni e più stabilità del previsto.
Mentre spesso si presume che l'influenza del movimento di una proteina di membrana sia limitata alle molecole lipidiche che la circondano direttamente, i ricercatori hanno osservato fluttuazioni più drammatiche su un intervallo più ampio, ha detto Smith. Le fluttuazioni assumevano la forma di un dito, come la melma schizzata su un muro. Tuttavia, anche dopo un movimento così drammatico, il nanodisco ritorna alla sua configurazione normale.
"Il fatto che abbiamo visto quei domini, e li abbiamo visti riprendersi da quei processi, suggerisce che le interazioni tra la proteina e la membrana in realtà hanno una gamma più ampia di quanto si pensi comunemente", ha detto Smith.
I ricercatori intendono utilizzare la loro tecnica di videografia elettronica per studiare altri tipi di proteine di membrana e altre classi di molecole e nanomateriali.
"Potremmo studiare i canali ionici che si aprono e si chiudono per regolare il flusso e le interazioni cellula-cellula utilizzando questa piattaforma", ha affermato Chen.
Qian Chen è anche affiliato al dipartimento di chimica, al Beckman Institute for Advanced Science and Technology, al Carle Illinois College of Medicine e al Materials Research Laboratory dell'Illinois.
Ulteriori informazioni: John W. Smith et al, Videografia elettronica di un tango lipidico-proteico, Science Advances (2024). DOI:10.1126/sciadv.adk0217
Informazioni sul giornale: La scienza avanza
Fornito dall'Università dell'Illinois a Urbana-Champaign