Gli enzimi che scindono le proteine svolgono un ruolo importante in molti processi fisiologici. Tali proteasi sono generalmente presenti in uno stato inattivo e si attivano solo in determinate condizioni. Alcuni sono collegati a malattie come infezioni o cancro, per cui è importante disporre di metodi in grado di rilevare selettivamente le proteasi attive.
In un articolo pubblicato sulla rivista Angewandte Chemie International Edition , gli scienziati hanno introdotto una nuova classe di sensori dell'attività della proteasi:nanoparticelle d'oro dotate di DNA peptidico.
Guidato da Devleena Samanta e Anna Capasso (Università del Texas ad Austin, USA), il team ha dimostrato che queste nanosonde possono rilevare più proteasi attive in parallelo (misurazione multiplex). Il metodo funziona a temperatura ambiente e non richiede una preparazione complicata del campione o strumenti elaborati.
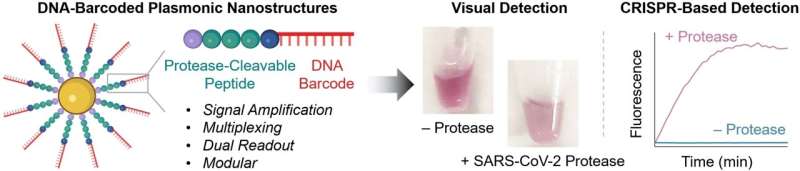
Al centro delle nuove sonde ci sono nanoparticelle d'oro dotate di catene costituite da un peptide e un frammento di DNA. La struttura peptidica è progettata per essere divisa dalla proteasi rilevata. Il DNA funge da codice a barre unico per identificare il peptide e amplifica anche il segnale. Se la proteasi desiderata è presente nella sua forma attiva nel campione, il peptide la scinde. Questo rilascia il codice a barre del DNA nella soluzione, dove può essere rilevato in base alla sua sequenza.
Per effettuare questa rilevazione, il team utilizza un test CRISPR/Cas12a:l’enzima Cas12a è legato a un RNA guida (gRNA) per formare un complesso inattivo. Il gRNA contiene un segmento che si lega specificamente al DNA del codice a barre. Questo attiva Cas12a, in modo che ora possa "tagliare" il DNA a filamento singolo (ssDNA).
Per il test, i ricercatori aggiungono molecole di ssDNA con un gruppo fluorescente (fluoroforo) a un'estremità e un quencher, che "spegne" la fluorescenza del fluoroforo (purché siano abbastanza vicini), all'altra. Se lo ssDNA viene tagliato, il fluoroforo e il quencher si allontanano ulteriormente. Ciò provoca una forte fluorescenza che indica che la proteasi da testare è presente (limite di rilevamento di circa 58 pM).
Se sul posto non sono disponibili strumenti e il test deve essere rapido, il rilevamento è possibile a occhio nudo:se la proteasi divide il peptide sulla sonda, la carica superficiale delle nanoparticelle d'oro cambia e queste si aggregano. Il colore di queste cosiddette "nanostrutture plasmoniche" dipende in modo significativo dal loro grado di aggregazione. È possibile rilevare concentrazioni di proteasi nanomolari in base al cambiamento di colore nella soluzione di prova.
Il rilevamento multiplex delle proteasi 3CL e della caspasi3 ha permesso al team di dimostrare l'elevata sensibilità e selettività del loro nuovo metodo. 3CL è un marcatore di infezione attiva da coronavirus e i pazienti COVID spesso hanno anche un’attività elevata del marcatore di apoptosi caspasi3. Il potenziale clinico di questo test è stato dimostrato anche dal rilevamento della catepsina B, una proteasi correlata al cancro del colon-retto, in tre diverse linee cellulari tumorali ottenute da pazienti.
Queste nanosonde producono segnali di fluorescenza 100 volte più elevati rispetto ai sensori di proteasi commerciali basati sulla fluorescenza. Inoltre, praticamente qualsiasi proteasi può essere rilevata se si conosce il peptide che scinde. Nel loro insieme, queste nanosonde possono potenzialmente consentire il rilevamento precoce delle malattie e migliorare la precisione e l'affidabilità dei test diagnostici attraverso il multiplexing.
Ulteriori informazioni: Subrata Pandit et al, Nanostrutture plasmoniche con codice a barre del DNA per il rilevamento della proteasi basato sull'attività, Angewandte Chemie International Edition (2023). DOI:10.1002/anie.202310964
Informazioni sul giornale: Edizione Internazionale Angewandte Chemie
Fornito da Wiley