Astratto grafico. Credito:Cella (2022). DOI:10.1016/j.cell.2022.08.003
Studi chiave nell'ultimo decennio hanno dimostrato che il microbioma intestinale, la raccolta di centinaia di specie batteriche che vivono nel sistema digestivo umano, influenza lo sviluppo neurale, la risposta alle immunoterapie contro il cancro e altri aspetti della salute. Ma queste comunità sono complesse e senza metodi sistematici per studiare i costituenti, le cellule e le molecole esatte legate a determinate malattie rimangono un mistero.
I ricercatori della Stanford University hanno costruito il microbioma sintetico più complesso e ben definito, creando una comunità di oltre 100 specie batteriche che è stata trapiantata con successo nei topi. La possibilità di aggiungere, rimuovere e modificare singole specie consentirà agli scienziati di comprendere meglio i legami tra il microbioma e la salute e, infine, sviluppare terapie microbiome di prima classe.
Molti studi chiave sul microbioma sono stati condotti utilizzando trapianti fecali, che introducono l'intero microbioma naturale da un organismo all'altro. Mentre gli scienziati silenziano regolarmente un gene o rimuovono una proteina da una cellula specifica o persino da un intero topo, non esiste un tale insieme di strumenti per rimuovere o modificare una specie tra le centinaia in un dato campione fecale.
"Tanto di ciò che sappiamo sulla biologia, non lo sapremmo se non fosse per la capacità di manipolare sistemi biologici complessi a tratti", ha affermato Michael Fischbach, Institute Scholar presso Sarafan ChEM-H e autore corrispondente dello studio, pubblicato nella Cella il 6 settembre.
Fischbach, professore associato di bioingegneria e microbiologia e immunologia, e altri hanno visto una soluzione:costruire un microbioma da zero coltivando individualmente e quindi mescolando i suoi batteri costituenti.
Costruire l'arca
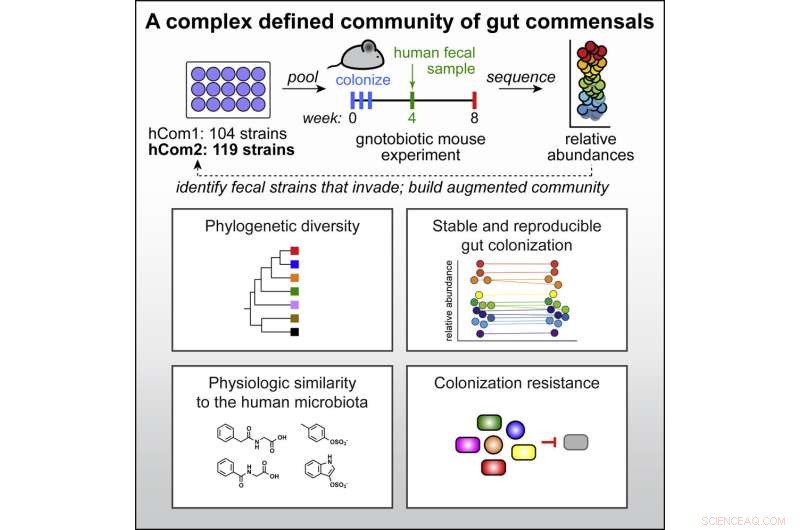
Ogni cellula del microbioma occupa una specifica nicchia funzionale, eseguendo reazioni che scompongono e accumulano molecole. Per costruire un microbioma, il team ha dovuto assicurarsi che la miscela finale non fosse solo stabile, mantenendo un equilibrio senza che nessuna singola specie prevalesse sul resto, ma anche funzionale, svolgendo tutte le azioni di un microbioma naturale e completo. Anche la selezione delle specie da includere nella loro comunità sintetica è stata difficile data la naturale variazione tra gli individui; due persone selezionate a caso condividono meno della metà dei loro geni microbici.
I ricercatori hanno deciso di costruire la loro colonia dai batteri più diffusi e si sono rivolti allo Human Microbiome Project (HMP), un'iniziativa del National Institutes of Health per sequenziare l'intero genoma microbico di oltre 300 adulti.
"Stavamo cercando l'Arca di Noè di specie batteriche nell'intestino umano, cercando di trovare quelle che erano quasi sempre presenti in qualsiasi individuo", ha detto Fischbach.
Hanno selezionato oltre 100 ceppi batterici che erano presenti in almeno il 20% degli individui HMP. L'aggiunta di alcune specie necessarie per alcuni studi successivi li ha portati a 104 specie, che sono cresciute in singoli ceppi e poi si sono mescolate in un'unica cultura combinata per creare quella che chiamano una comunità umana, o hCom1.
Sebbene soddisfatto che i ceppi potessero coesistere in laboratorio, il vero test era se la loro nuova colonia avrebbe messo radici nell'intestino. Hanno introdotto hCom1 nei topi che sono accuratamente progettati per non avere batteri presenti. hCom1 era notevolmente stabile, con il 98% delle specie costituenti che colonizzavano l'intestino di questi topi privi di germi e i livelli di abbondanza relativa di ciascuna specie rimanevano costanti per due mesi.
Invasione straniera
Per rendere la loro colonia più completa, i ricercatori volevano assicurarsi che tutte le funzioni vitali del microbioma fossero eseguite da una o più specie. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge
To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases. + Esplora ulteriormente