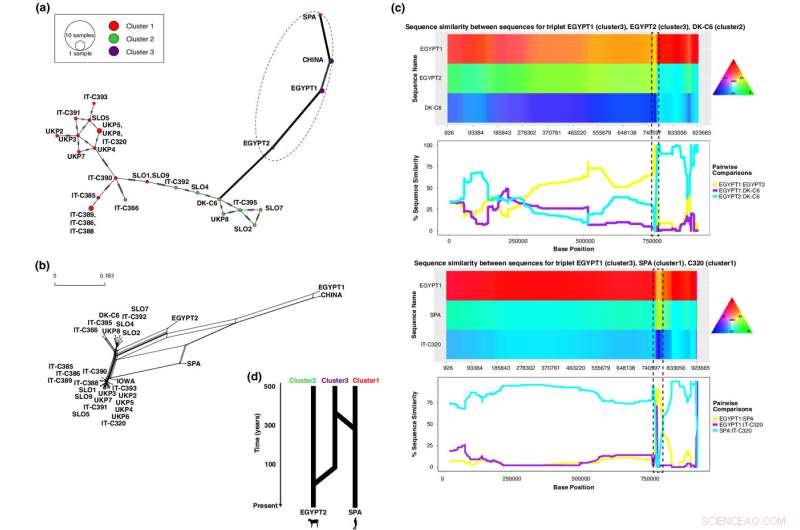
L'analisi di ricombinazione del cromosoma 1 identifica due sequenze ibride, Spa1 (un isolato umano dal cluster 1) e Egypt 2 (un isolato di vitello dal cluster 3). (a) Rete che mostra che gli aplotipi degli isolati ibridi Spa1 ed Egypt2 sono divergenti dal resto degli isolati e sono strettamente associati alle loro sequenze parentali minori (isolati del cluster 3). (b) Rete filogenetica che mostra anelli tra isolati ibridi, che rappresentano la potenziale ricombinazione. (c) Grafici di somiglianza di sequenza ottenuti con HybridCheck per diverse triplette di isolati coinvolti in un evento di ricombinazione (ibrido, genitore maggiore, genitore minore). La somiglianza di sequenza è mostrata lungo le posizioni cromosomiche (asse x) da una mappa dei colori in cui le regioni con lo stesso polimorfismo condividono i colori (in alto) e da grafici a linee che riportano la somiglianza percentuale sull'asse y (in basso). I grafici delle regioni ricombinanti che presentano un'elevata somiglianza tra gli isolati ibridi e le loro sequenze parentali minori nelle regioni ricombinanti sono racchiusi in riquadri tratteggiati. (d) Rappresentazione schematica degli eventi di ricombinazione negli isolati ibridi, che mostra che Egypt2 e Spa1 hanno entrambi ricevuto variazioni genetiche da un cluster 3, circa 49 (21–96; IC 95%) e 289 (204–395; IC 95%) anni fa, rispettivamente. Credito:Ecologia molecolare (2022). DOI:10.1111/mec.16556
È probabile che i parassiti che causano una grave diarrea diventino più virulenti a causa della velocità con cui si scambiano il DNA e si evolvono, secondo una nuova ricerca dell'Università dell'East Anglia.
I ricercatori hanno studiato i genomi di Cryptosporidium parvum, un parassita zoonotico che provoca grave diarrea sia negli esseri umani che negli animali.
Hanno scoperto che diversi lignaggi di questi parassiti stanno scambiando sempre più il loro DNA, il che ha aiutato il parassita a evolversi più velocemente, risultando potenzialmente in ceppi più virulenti e meglio adattati.
Il nuovo studio mostra che i lignaggi di Cryptosporidium parvum hanno scambiato più DNA negli ultimi 200 anni rispetto a tutto il tempo prima.
E lo attribuiscono alla globalizzazione e al nostro contatto sempre più stretto con gli animali che aumenta il tasso di eventi di ricaduta.
Uno dei ricercatori principali, il professor Cock Van Oosterhout, della School of Environmental Sciences dell'UEA, ha dichiarato:"Cryptosporidium è un genere importante di parassiti zoonotici ed è uno dei numerosi microrganismi che causano malattie diarroiche sia nell'uomo che in alcuni animali. In umano, è responsabile di circa 57.000 decessi ogni anno, l'80% dei quali tra bambini di età inferiore ai cinque anni.
"La stragrande maggioranza di questi si verifica nei paesi a basso reddito, ma i focolai si verificano anche nel Regno Unito e altrove in Europa. Non sono disponibili farmaci o vaccini efficaci, quindi è fondamentale comprendere la trasmissione e l'evoluzione di questo parassita.
"Sappiamo che esistono diversi lignaggi del parassita Cryptosporidium parvum, ma volevamo capire di più su come si evolvono e, in particolare, perché questa specie potrebbe diventare più virulenta di quanto non fosse in passato."
Il team, guidato dall'UEA in collaborazione con scienziati in Italia e Australia, ha utilizzato l'analisi della sequenza dell'intero genoma per saperne di più su questi scambi genetici.
Hanno confrontato 32 sequenze dell'intero genoma di ceppi umani e animali raccolti in Europa, Stati Uniti, Egitto e Cina.
Il Prof. Van Oosterhout ha dichiarato:"Abbiamo scoperto che diversi lignaggi di questi parassiti stanno scambiando sempre più il loro DNA. Negli ultimi 200 anni, circa il 22% del genoma di questi parassiti è stato scambiato.
"Questo è molto più del DNA in cui si sono scambiati tutto il tempo prima. I geni coinvolti nella virulenza sembrano essere particolarmente colpiti da tali scambi genetici. Il genoma dei parassiti che stanno infettando gli esseri umani possiede del DNA di parassiti che normalmente si trovano in mucche e agnelli.
"Riteniamo che questi scambi genetici aiutino il parassita a evolversi più velocemente e che ciò possa portare a parassiti umani più virulenti e meglio adattati. Questo è davvero importante perché, come ci ha mostrato il COVID-19, i parassiti umani possono evolversi rapidamente.
"Ipotizziamo che il tasso di evoluzione sia ulteriormente accelerato perché il parassita può evolversi in più specie ospiti. Ciò significa che nuovi adattamenti che si sono evoluti in parassiti che infettano una specie ospite possono ora essere utilizzati da parassiti che infettano un'altra specie di ospiti.
"La maggiore connettività nel nostro mondo globalizzato e lo stretto contatto tra esseri umani e animali domestici aumenta il tasso di eventi di spillover e spillback.
"Utilizzando l'analisi dell'intera sequenza del genoma, ora possiamo studiare questi scambi genetici e identificare quando e dove hanno luogo. Questo ci aiuterà a controllare meglio queste malattie infettive emergenti di parassiti zoonotici e agenti patogeni", ha aggiunto.
"Recenti scambi genetici e mescolanze modellano il genoma e la struttura della popolazione di un parassita zoonotico" è pubblicato sulla rivista Molecular Ecology .