La tecnologia di sequenziamento del genoma fornisce migliaia di nuovi genomi vegetali ogni anno. In agricoltura, i ricercatori uniscono queste informazioni genomiche con dati osservativi (che misurano vari tratti delle piante) per identificare le correlazioni tra varianti genetiche e tratti delle colture come il conteggio dei semi, la resistenza alle infezioni fungine, il colore o il sapore del frutto.
Tuttavia, la comprensione di come la variazione genetica influenza l’attività genetica a livello molecolare è piuttosto limitata. Questa lacuna nella conoscenza ostacola la selezione di "colture intelligenti" con una migliore qualità e un ridotto impatto ambientale negativo, ottenute mediante la combinazione di varianti genetiche specifiche con funzione nota.
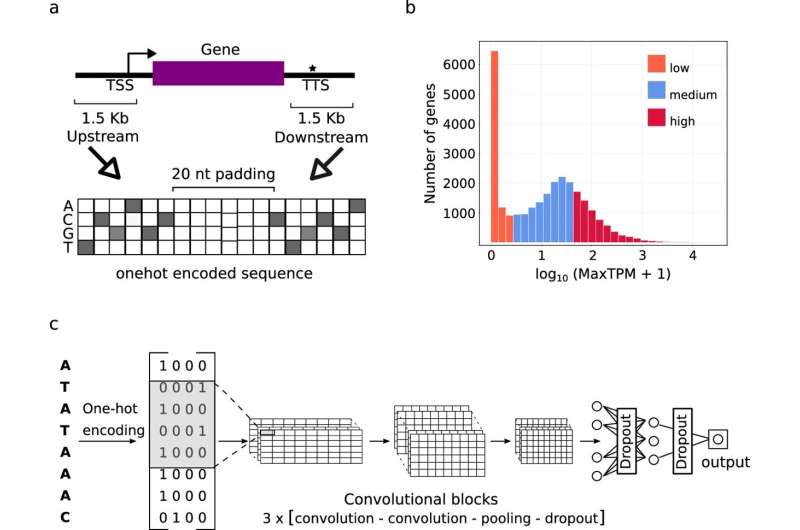
I ricercatori dell’Istituto IPK Leibniz e del Forschungszentrum Jülich (FZ) hanno fatto un passo avanti significativo per affrontare questa sfida. Guidato dal dottor Jedrzej Jakub Szymanski, il team di ricerca internazionale ha addestrato modelli interpretabili di deep learning, un sottoinsieme di algoritmi di intelligenza artificiale, su un vasto set di dati di informazioni genomiche di varie specie di piante.
"Questi modelli non solo sono stati in grado di prevedere con precisione l'attività genetica dalle sequenze, ma hanno anche individuato quali parti della sequenza contribuiscono a queste previsioni", spiega il capo del gruppo di ricerca dell'IPK "Network Analysis and Modeling". La tecnologia di intelligenza artificiale applicata dai ricercatori è simile a quella utilizzata nella visione artificiale, che prevede il riconoscimento delle caratteristiche facciali nelle immagini e la deduzione delle emozioni.
In contrasto con gli approcci precedenti basati sull'arricchimento statistico, qui i ricercatori hanno combinato l'identificazione delle caratteristiche della sequenza con la determinazione del numero di copie di mRNA nel quadro di un modello matematico che è stato addestrato tenendo conto delle informazioni biologiche sulla struttura del modello genetico e sull'omologia della sequenza, quindi gene evoluzione.
"Siamo rimasti davvero stupiti dall'efficacia. Nel giro di pochi giorni di formazione, abbiamo riscoperto molte sequenze regolatrici conosciute e abbiamo scoperto che circa il 50% delle caratteristiche identificate erano completamente nuove. Questi modelli si generalizzavano in modo eccellente tra le specie vegetali su cui non erano state addestrate, rendendole sono preziosi per analizzare i genomi appena sequenziati," afferma il dottor Szymanski.
"E abbiamo specificamente dimostrato la loro applicazione in diverse cultivar di pomodoro con dati di sequenziamento a lettura lunga. Abbiamo individuato specifiche variazioni della sequenza regolatoria che spiegavano le differenze osservate nell'attività genetica e, di conseguenza, le variazioni di forma, colore e robustezza. Si tratta di un notevole miglioramento rispetto a associazioni statistiche classicamente utilizzate di polimorfismi a singolo nucleotide."
Il team ha condiviso apertamente i propri modelli e ha fornito un'interfaccia web per il loro utilizzo. "È interessante notare che sono stati fatti molti sforzi per degradare le prestazioni del nostro modello. Per evitare risultati eccessivamente ottimistici dovuti alla ricerca di scorciatoie da parte dell'intelligenza artificiale, mi è stato richiesto di approfondire la biologia della regolazione genetica per eliminare qualsiasi potenziale errore, ridurre la perdita di dati e l'overfitting," afferma Fritz Forbang Peleke, il ricercatore capo sull'apprendimento automatico e primo autore dello studio, che è stato pubblicato sulla rivista Nature Communications .
Il dottor Simon Zumkeller, coautore e biologo evoluzionista della FZ Jülich, afferma:"Con le analisi presentate possiamo studiare e confrontare la regolazione genetica nelle piante e dedurne l'evoluzione. Anche per le applicazioni pratiche, il metodo fornisce una nuova base. Ci stiamo avvicinando all'identificazione di routine degli elementi regolatori dei geni nei genomi vegetali conosciuti e recentemente sequenziati, in vari tessuti e in diverse condizioni ambientali."
Ulteriori informazioni: Fritz Forbang Peleke et al, Apprendimento approfondito del codice di regolamentazione cis per l'espressione genetica in piante modello selezionate, Nature Communications (2024). DOI:10.1038/s41467-024-47744-0
Informazioni sul giornale: Comunicazioni sulla natura
Fornito dall'Istituto Leibniz di genetica vegetale e ricerca sulle piante coltivate