Credito:Università della Pennsylvania
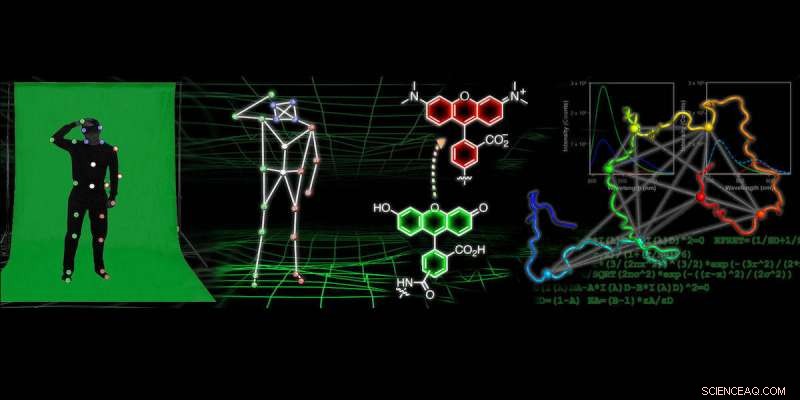
In molti film d'animazione moderni, il trucco per ottenere movimenti realistici per i singoli personaggi e oggetti risiede nella tecnologia di acquisizione del movimento. Questo processo spesso coinvolge qualcuno che indossa una tuta da ricerca coperta da piccoli, palline colorate mentre una telecamera cattura la posizione di quelle palline colorate, che viene poi utilizzato per rappresentare come si muove la persona.
I ricercatori dell'Università della Pennsylvania stanno sviluppando una tecnologia simile per ottenere "filmati" a risoluzione atomica che tracciano come le proteine si piegano e cambiano forma. Per generare questi filmati, gli scienziati etichettano la proteina con sonde in molte posizioni e osservano il movimento di tali etichette. I dati di fluorescenza sulle posizioni relative delle sonde possono quindi essere utilizzati per costruire modelli computazionali della struttura proteica in dettaglio atomico. Questa ricerca potrebbe portare a miglioramenti nei farmaci usati per trattare le malattie neurodegenerative, così come nuovi metodi di imaging che potrebbero portare alla loro individuazione precoce.
La ricerca è stata uno sforzo multidisciplinare guidato da E. James Petersson. Il documento è stato pubblicato in Giornale Biofisico .
"Una delle grandi domande fondamentali in biochimica è come le proteine si piegano in una certa forma, " ha detto Petersson, "e questo è dettato dalla sequenza degli amminoacidi nella proteina. L'informazione in tutte le interazioni delle catene laterali degli amminoacidi porta in qualche modo a piegarsi in una forma corretta".
In scenari sani, Petersson ha detto, quella forma corretta permette alla proteina di avere diverse funzioni, come trasportare ossigeno nel sangue o diventare "macchine molecolari" che alla fine portano a movimenti muscolari, come quelli necessari per camminare e correre.
Ma in alcuni stati patologici, in particolare nelle malattie neurodegenerative come l'Alzheimer e il Parkinson, le proteine si ripiegano male in una forma malsana, che può causare l'aggregazione di più copie delle proteine in "grovigli simili a spaghetti o lunghe fibrille". Queste fibrille, Petersson ha detto, sono tossici per i neuroni, che è alla base del morbo di Alzheimer e del morbo di Parkinson.
"Dato che tutto questo implica il ripiegamento delle proteine in una certa forma, allora quello che vorremmo fare è tenere traccia dei cambiamenti nella forma delle proteine, " ha detto. "Ci sono un certo numero di tecniche diverse che possono essere utilizzate per farlo, ma ci piace la fluorescenza perché è possibile acquisire dati di fluorescenza abbastanza velocemente da poter effettivamente osservare il ripiegamento delle proteine in tempo reale. In definitiva, vorremmo provare a osservare le proteine che si ripiegano nelle cellule".
Per ottenere informazioni sulla forma delle proteine utilizzando sonde fluorescenti, i ricercatori utilizzano una tecnica chiamata trasferimento di energia per risonanza di fluorescenza, che richiede loro di misurare molte distanze tra diversi punti sulla proteina e quindi utilizzare tali informazioni per comprenderne la forma, simile alla tecnologia di acquisizione del movimento.
In questo documento, i ricercatori hanno effettuato circa 30 misurazioni di diverse distanze all'interno della proteina alfa-sinucleina in diversi stati in cui sta cambiando forma. Hanno quindi usato quella raccolta di misurazioni della distanza in combinazione con complessi modelli computazionali per ottenere strutture di risoluzione atomica della forma della proteina.
Ferrie ha ricevuto una borsa di studio dalla Parkinson's Disease Foundation per trascorrere l'estate nel laboratorio di David Baker all'Università di Washington, dove Rosetta, uno dei programmi più comunemente usati per modellare le proteine, è stato sviluppato.
"Rosetta è progettata per modellare proteine stabili ben ripiegate, "Petersson ha detto, "proteine non disordinate che possono cambiare forma, quindi Jack ha dovuto riscrivere personalmente il codice per poter modellare queste proteine indisciplinate".
Ferrie e Yoon hanno effettuato una serie di misurazioni sperimentali che potrebbero essere utilizzate per dirigere il ripiegamento proteico in modo che fosse coerente con le misurazioni sperimentali, che gli ha permesso di modellare la forma delle proteine.
Poiché si trattava di un nuovo approccio, i ricercatori hanno voluto dimostrare che le strutture che escono dai modelli computazionali erano coerenti con la realtà. Per fare ciò, hanno condotto tre tipi di esperimenti per abbinare i dati reali con i modelli che escono da questi esperimenti di fluorescenza.
Hanno collaborato con il gruppo di Rhoades per convalidare le strutture modellate utilizzando misurazioni di fluorescenza di singole molecole effettuate da Pan. Lavorando nel gruppo di Fakhraii, Lin, ha utilizzato un diverso tipo di tecnica chiamata microscopia a forza atomica per visualizzare la proteina e convalidare i modelli. Chris Dobson, professore di chimica all'Università di Cambridge, dati condivisi di risonanza magnetica nucleare sull'alfa-sinucleina che hanno fornito un'ulteriore conferma dell'accuratezza dei modelli al computer.
Il documento rappresenta una delle più grandi librerie di proteine etichettate con fluorofori sintetici finora riportate. Secondo Petersson, i ricercatori avevano bisogno di effettuare molte misurazioni della distanza su diverse regioni della proteina per avere dati sufficienti per generare modelli computazionali. Ferrie e Haney hanno dovuto trovare un approccio semplificato per collegare diversi set di sonde che avrebbero funzionato su diversi intervalli di distanza.
I ricercatori stanno ora lavorando per applicare questa tecnica per modellare la struttura proteica nelle forme aggregate che sono tossiche per i neuroni e per modellare la sua risposta ai farmaci che ne farebbero cambiare forma, impedendo questa aggregazione.
"La capacità di osservare una proteina mentre cambia forma, " ha detto Petersson, "e ottenere effettivamente strutture da questo è un obiettivo scientifico di base davvero importante a cui abbiamo lavorato per 10 anni. Ci sono state alcune scoperte davvero impressionanti nell'ottenere strutture di proteine nelle malattie neurodegenerative, ma la tecnica della fluorescenza ha il potenziale per farlo nelle cellule viventi, che nessun'altra tecnica ha la capacità di fare."
Secondo Petersson, proteine che assumono più forme, impilando copie diverse l'una sull'altra e aggregando, come nelle malattie neurodegenerative, sono un importante problema di biochimica di base che altre tecniche di biologia strutturale non possono realmente affrontare. A better understanding of how that works and what those shapes are has the potential to make an impact on diseases such as Parkinson's and to give researchers an opportunity to figure out how drug or diagnostic models interact with the protein.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."