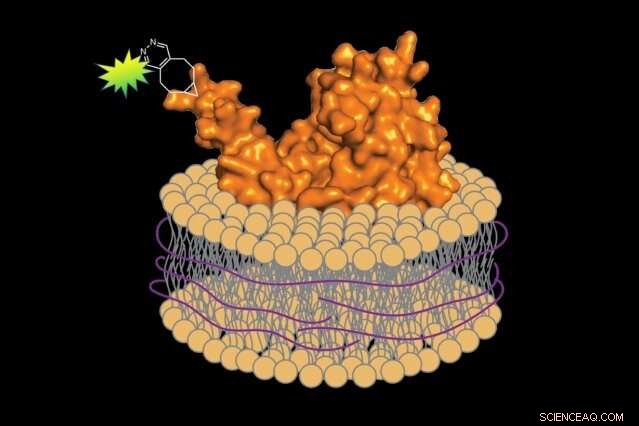
Utilizzando un metodo senza detersivo, i biologi possono preparare proteine fluorescenti insieme a un piccolo segmento della membrana cellulare associata, preservare l'ambiente nativo della proteina. Credito:Jean-Marie Swiecicki
Tutte le cellule hanno una membrana lipidica che circonda i loro componenti interni, formando una barriera protettiva per controllare ciò che entra e ciò che rimane fuori. Le proteine incorporate in queste membrane sono essenziali per la vita; aiutano a facilitare il trasporto dei nutrienti, conversione e stoccaggio dell'energia, e comunicazione cellulare. Sono importanti anche nelle malattie umane, e rappresentano circa il 60% dei bersagli farmacologici approvati. Per studiare queste proteine di membrana al di fuori della complessità della cellula, i ricercatori devono usare un detergente per rimuovere la membrana ed estrarli. Però, determinare il miglior detersivo per ciascuna proteina può comportare numerosi tentativi ed errori. E, rimuovere una proteina dal suo ambiente naturale rischia di destabilizzare la struttura piegata e di interrompere la funzione.
In uno studio pubblicato il 9 dicembre in Biologia chimica cellulare , scienziati del MIT hanno ideato un modo rapido e generalizzabile per estrarre, purificare, ed etichettare le proteine della membrana per l'imaging senza alcun detergente, portando con sé una porzione della membrana circostante per proteggere la proteina e simulare il suo ambiente naturale. Il loro approccio combina tecniche chimiche e biochimiche consolidate in un modo nuovo, isolando efficacemente la proteina in modo che possa essere etichettata con fluorescenza ed esaminata al microscopio.
"Scherzo sempre sul fatto che non è molto realistico studiare le proteine nel sapone, ", afferma l'autore senior Barbara Imperiali, professore di biologia e chimica. "Abbiamo creato un flusso di lavoro che consente di visualizzare le proteine di membrana mantenendo le loro identità e interazioni native. Si spera che ora meno persone eviteranno di studiare le proteine di membrana, data la loro importanza in molti processi fisiologici."
In qualità di membro del laboratorio Imperiali, l'ex postdoc e autore principale Jean-Marie Swiecicki ha studiato le proteine di membrana del patogeno alimentare Campylobacter jejuni. In questo studio, Swiecicki si è concentrato su PglC e PglA, due proteine di membrana che svolgono un ruolo nel consentire ai batteri di infettare le cellule umane. I suoi esperimenti richiedevano l'etichettatura di PglC e PglA con tag fluorescenti per tracciarli. Però, non era soddisfatto dei metodi esistenti per farlo.
In alcuni casi, i tag fluorescenti che devono essere incorporati nella proteina per visualizzarla sono troppo grandi per essere posizionati in posizioni definite. In altri casi, questi tag non brillano abbastanza, o interferire con la struttura e la funzione della proteina.
Per evitare tali problemi, Swiecicki decise di utilizzare un metodo noto come "mutagenesi innaturale di amminoacidi". Gli amminoacidi sono le unità che compongono la proteina, e la mutagenesi amminoacidica innaturale comporta l'aggiunta di un nuovo amminoacido contenente un gruppo chimico ingegnerizzato all'interno della sequenza proteica. Questo gruppo chimico può quindi essere etichettato con un'etichetta luminosa.
Swiecicki ha inserito il codice genetico per le proteine di membrana di C. jejuni in un batterio diverso, Escherichia coli. All'interno di E. coli, potrebbe incorporare l'aminoacido innaturale, che potrebbe essere chimicamente modificato per aggiungere l'etichetta fluorescente.
Quando è arrivato il momento di rimuovere le proteine dalla membrana, sostituì il detersivo con una sostanza diversa:un polimero di acido stirene-maleico (SMA). A differenza del detersivo, La SMA avvolge la proteina estratta e un piccolo segmento della membrana associata in un guscio protettivo, preservando il suo ambiente nativo. Imperiali spiega, "È come una sciarpa che ti protegge il collo dal freddo."
Swiecicki ha quindi potuto monitorare le proteine luminose al microscopio per verificare che la sua tecnica fosse sufficientemente selettiva da isolare le singole proteine di membrana. L'intero processo, lui dice, bastano pochi giorni, ed è generalmente molto più veloce e affidabile rispetto ai metodi di estrazione a base di detergenti, che possono richiedere mesi e richiedere l'esperienza di biochimici altamente qualificati per l'ottimizzazione.
"Non direi che è una bacchetta magica che funzionerà per ogni singola proteina, " dice. "Ma è uno strumento altamente efficiente che potrebbe rendere più facile lo studio di molti diversi tipi di proteine di membrana". lui dice, può anche aiutare a facilitare gli screening dei farmaci ad alto rendimento.
"Come qualcuno che lavora su complessi proteici di membrana, Posso attestare il grande bisogno di metodi migliori per studiarli, "dice Suzanne Walker, un professore di microbiologia alla Harvard Medical School che non è stato coinvolto nello studio. Spera di estendere l'approccio delineato nel documento ai complessi proteici che studia nel suo laboratorio. "Ho apprezzato l'ampio dettaglio incluso nel testo su come applicare con successo la strategia, "aggiunge.
I prossimi passi saranno testare la tecnica su proteine di mammifero, e isolare più proteine contemporaneamente nel guscio SMA per osservare le loro interazioni. E, Certo, ogni nuova tecnica merita un nome. "Stiamo ancora lavorando a un acronimo accattivante, " dice Imperiali. " Qualche idea?"
Questa storia è stata ripubblicata per gentile concessione di MIT News (web.mit.edu/newsoffice/), un popolare sito che copre notizie sulla ricerca del MIT, innovazione e didattica.