
Un'illustrazione mostra una proteina di maggiore istocompatibilità (grigia) che comprende un peptide estratto da un virus SARS-CoV (rosa). Il complesso aiuta a innescare l'attivazione delle cellule T che fanno parte del sistema immunitario. I ricercatori della Rice University hanno scoperto un residuo di legame non ancorato nel peptide che potrebbe contribuire sia al legame che all'attivazione delle cellule T necessarie per sconfiggere il virus. Credito:Kavraki Lab/Rice University
La "posizione 4" non sembrava importante fino a quando i ricercatori non hanno esaminato a lungo un particolare peptide.
Quella parte del peptide estratto da un virus SARS-CoV si è rivelata avere un'influenza inaspettata ma significativa su come si lega stabilmente con un recettore centrale per la capacità del sistema immunitario di attaccare le cellule malate.
In uno studio pubblicato da Atti dell'Accademia Nazionale delle Scienze , i ricercatori della Brown School of Engineering della Rice University e dell'MD Anderson Cancer Center dell'Università del Texas hanno rivelato modelli a risoluzione atomica che dettagliano non solo il legame ma anche, per la prima volta, i meccanismi di separazione che sono alla base di un componente chiave del sistema immunitario.
Dicono che una migliore comprensione dell'intero meccanismo potrebbe portare a progressi nell'immunoterapia che aumentano la capacità del corpo di combattere le malattie.
Lydia Kavraki, scienziata informatica del riso, l'alunno Jayvee Abella e il ricercatore postdottorato Dinler Antunes, condotto lo studio.
"Trovare buoni bersagli per innescare una risposta immunitaria protettiva è molto impegnativo, soprattutto nella ricerca sul cancro, Antunes ha detto. "Il fatto che questo particolare peptide è stato previsto per non legarsi agli HLA (antigeni leucocitari umani) da metodi basati sulla sequenza evidenzia un punto cieco nella nostra attuale capacità di previsione.
"Incorporando l'analisi strutturale, possiamo rilevare il contributo di queste interazioni secondarie al legame e alla stabilità dei peptidi, speriamo che ci consenta di trovare obiettivi migliori per lo sviluppo di vaccini antivirali e l'immunoterapia del cancro basata sulle cellule T, " Egli ha detto.
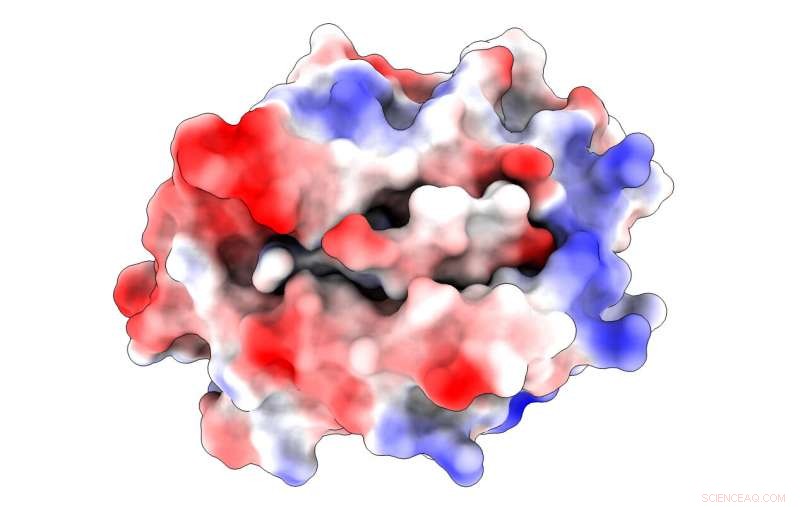
Un'illustrazione mostra le cariche elettrostatiche (il blu è positivo, rosso negativo) in una proteina maggiore di istocompatibilità legata a un peptide estratto da un virus SARS-CoV. Il complesso aiuta a innescare l'infezione delle cellule T che fanno parte del sistema immunitario. I ricercatori della Rice University hanno scoperto un residuo di legame non ancorato nel peptide che potrebbe contribuire sia al legame che all'attivazione delle cellule T necessarie per sconfiggere il virus. Credito:Kavraki Lab/Rice University
I ricercatori hanno usato le loro simulazioni per illuminare i dettagli di come il peptide SARS intracellulare, QFKDNVILL, si lega a una proteina recettore MHC nota come HLA-A*24:02, principalmente alle ancore dominanti su entrambe le estremità del peptide (nelle posizioni 2 e 9) e le presenta per l'ispezione alle cellule T del sistema immunitario.
Il legame stabile di un peptide e MHC è un prerequisito per l'attivazione delle cellule T, che cercano peptidi che normalmente non si trovano nelle cellule sane. Se il peptide e la proteina non si legano, la cellula T non è sollecitata ad attaccare.
"Questo era noto da studi precedenti sugli stati legati e non legati di molti di questi complessi, " Ha detto Kavraki. "Ciò che non hanno catturato sono stati gli stati intermedi e le transizioni che portano da uno stato all'altro, soprattutto lo svincolamento.
"Penso che questa sia l'unica analisi che mostra la separazione dei peptidi dall'MHC con risoluzione atomica, Kavraki ha detto. "Altri peptidi hanno caratteristiche simili e pensiamo che avrebbero comportamenti simili".
Tutte queste interazioni sono state rivelate in grande dettaglio attraverso modelli di stato di Markov che analizzano come i sistemi cambiano nel tempo. In questo caso, i modelli hanno rivelato l'importanza dei siti secondari che supportano le ancore primarie del peptide. Ecco dove spiccava la posizione 4.
"Ci sono i principali, ancore canoniche che la gente conosce, ma ci sono queste interazioni secondarie che contribuiscono al legame e alla stabilità, " disse Antunes. "Questi sono più difficili da catturare, ma in questo studio sembra che la posizione 4 giochi un ruolo molto importante. Quando lo muti, influenza il comportamento del peptide mentre si separa dalla molecola".
I ricercatori hanno modellato le mutazioni dell'MHC per vedere come avrebbero influenzato il legame e hanno scoperto di sostenere l'importanza della posizione 4 per la stabilità del complesso.
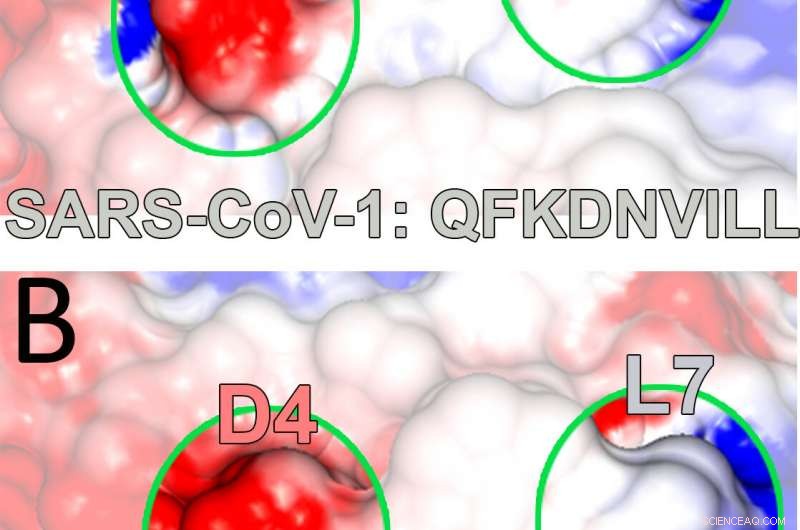
Le mappe del potenziale elettrostatico create dai modelli della Rice University mostrano una proteina di istocompatibilità importante legata a un peptide estratto dai virus SARS-CoV. Tali complessi dinamici possono innescare l'attivazione delle cellule T che fanno parte del sistema immunitario. Il team di Rice ha scoperto l'influenza stabilizzante di un residuo di legame alternativo (in posizione 4) comune sia al peptide che hanno studiato, in alto, e uno associato a SARS-CoV-2, in fondo, responsabile della malattia da COVID-19. Credito:Kavraki Lab/Rice University
"Il nostro approccio computazionale è stato in grado di fare previsioni sull'effetto delle mutazioni che vengono poi verificate sperimentalmente, " ha detto la coautrice Cecilia Clementi, un ex professore della Rice che recentemente è diventato Einstein Professor di Fisica presso la Libera Università di Berlino.
I ricercatori hanno sviluppato un processo in due fasi per semplificare la complessità computazionale dell'analisi su scala atomica di grandi molecole. La prima fase ha utilizzato una tecnica chiamata campionamento ad ombrello per accelerare l'esplorazione iniziale delle molecole. Il secondo, fase esplorativa ha utilizzato il campionamento adattivo, in cui le simulazioni sono guidate per accelerare la costruzione del modello di Markov.
"La sfida è che questi MHC sono sistemi piuttosto grandi che i chimici computazionali possono simulare, " disse Abella, le cui ricerche sull'argomento hanno formato gran parte della sua tesi di dottorato. "Abbiamo dovuto fare alcune approssimazioni e sfruttare i progressi in queste classi di metodi per andare avanti.
"Non siamo i primi a studiare lo svincolamento, ma ciò che caratterizza il nostro lavoro rispetto agli altri è che manteniamo la piena risoluzione atomica nelle nostre simulazioni, " ha detto. "Altre opere utilizzano una tecnica nota come una catena di Markov Monte Carlo, considerando che usiamo la dinamica molecolare, che ci consente di incorporare il tempo nei nostri calcoli per catturare la cinetica".
I loro metodi possono essere applicati ad altri complessi peptide-MHC con modelli 3-D esistenti. "Questo era, in un certo senso, uno studio di fattibilità per mostrare che possiamo usare la dinamica molecolare e costruire un modello di stato di Markov di un sistema di queste dimensioni, " disse Abella.
I ricercatori hanno anche notato la rilevanza dello studio per l'attuale lotta contro COVID-19, come il peptide SARS che hanno visto, QFKDNVILL, è molto simile al peptide NFKDQVILL in SARS-CoV-2, con le stesse tasche di rilegatura nelle posizioni 2, 4 e 9.
"Questi risultati suggeriscono che entrambi i peptidi possono legarsi a HLA-A*24:02 e fornire bersagli per le risposte antivirali delle cellule T, di grande interesse alla luce dell'attuale pandemia, " ha detto il co-autore Gregory Lizée, un professore nel dipartimento di oncologia medica del melanoma presso MD Anderson. "Ma questi risultati fanno luce anche su molti altri potenziali bersagli immunitari, compresi quelli di altri virus e persino i tumori umani".
Kavraki ha notato che il lavoro sperimentale della collaboratrice a lungo termine Lizée e Kyle Jackson, un assistente di ricerca laureato presso il laboratorio di Lizée che ha prodotto le proteine mutanti, erano fondamentali per convalidare le loro simulazioni. Il laboratorio di Kavraki ha vinto una sovvenzione per la ricerca sulla risposta rapida della National Science Foundation (NSF) per aiutare a identificare frammenti di proteine virali SARS-CoV-2 come possibili bersagli per lo sviluppo di vaccini.