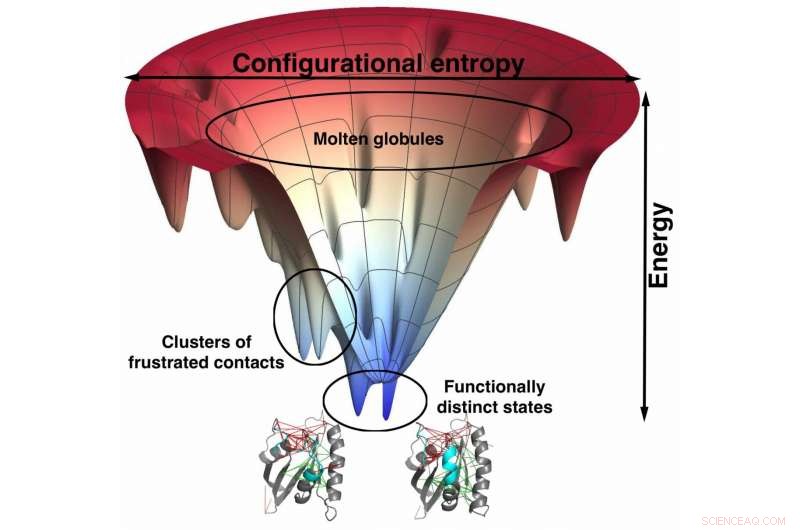
I modelli su scala atomica degli scienziati della Rice University basati su quelli utilizzati per prevedere come le proteine si ripiegano mostrano una forte correlazione tra i siti di legame minimamente frustrati e la specificità del farmaco. L'imbuto, una rappresentazione visiva del panorama energetico della proteina mentre si piega, aiuta a individuare quei siti frustrati. Tali modelli potrebbero portare a farmaci meglio progettati con minori effetti collaterali. Credito:Mingchen Chen/Rice University
Sapere con precisione dove sono frustrate le proteine potrebbe fare molto per produrre farmaci migliori.
Questo è uno dei risultati di un nuovo studio degli scienziati della Rice University alla ricerca dei meccanismi che stabilizzano o destabilizzano sezioni chiave delle biomolecole.
Modelli in scala atomica del teorico della Rice Peter Wolynes, l'autore principale e alunno Mingchen Chen e i loro colleghi del Center for Theoretical Biological Physics mostrano che non solo alcune specifiche sequenze frustrate nelle proteine sono necessarie per consentire loro di funzionare, la loro localizzazione offre anche indizi per ottenere una migliore specificità per i farmaci.
Questa conoscenza potrebbe anche aiutare a progettare farmaci con meno effetti collaterali, ha detto Wolynes.
Lo studio ad accesso aperto del team appare in Comunicazioni sulla natura .
I modelli su scala atomica si concentrano sulle interazioni all'interno di possibili siti di legame piuttosto che sulla stragrande maggioranza delle interazioni nelle proteine che guidano il loro ripiegamento. I modelli di risoluzione più fine consentono l'incorporazione di cofattori come ligandi chimicamente attivi, comprese le molecole dei farmaci. I ricercatori affermano che questa capacità fornisce nuove informazioni sul motivo per cui i ligandi vengono catturati al meglio solo da proteine specifiche e non da altre.
"leganti innaturali, "aka droghe, tendono a legarsi meglio con quelle tasche frustrate nelle proteine che diventano minimamente frustrate una volta che i farmaci si legano, ha detto Wolynes. Avere un modo per trovare e quindi apprendere i dettagli di questi siti minimamente frustrati aiuterebbe le aziende farmaceutiche a eliminare un sacco di tentativi ed errori.
"Il modo standard di progettare farmaci è provare 10, 000 siti di legame su una proteina per trovare quelli che si adattano, " ha detto Wolynes. "Stiamo dicendo che non è necessario campionare tutti i possibili siti di legame, solo un numero ragionevolmente giusto per capire le statistiche di cosa potrebbe funzionare negli ambienti locali.
"È la differenza tra fare un sondaggio e avere effettivamente un'elezione, " ha detto. "Il sondaggio è più economico, ma dovrai comunque controllare le cose."
I ricercatori di Rice sono noti per la loro teoria del panorama energetico su come le proteine si ripiegano. Di solito impiega modelli a grana grossa in cui gli amminoacidi sono rappresentati da pochi siti.
Quella strategia richiede meno potenza di calcolo rispetto al tentativo di determinare le posizioni nel tempo di ogni atomo in ogni residuo, eppure si è dimostrato estremamente accurato nel prevedere come le proteine si ripiegano in base alle loro sequenze. Ma per questo studio, i ricercatori hanno modellato proteine e complessi proteina-ligando a livello atomico per vedere se potevano scoprire come la frustrazione dia ad alcune parti di una proteina la flessibilità necessaria per legarsi ad altre molecole.
"Una delle cose grandiose della modellazione a risoluzione di tutti gli atomi è che ci permette di valutare se le molecole dei farmaci si adattano bene o meno ai siti di legame, " Wolynes ha detto. "Questo metodo è in grado di mostrare rapidamente se un sito di legame per un determinato farmaco sarà minimamente frustrato o rimarrà una regione frustrata. Se dopo che la molecola si lega il sito rimane frustrato, la proteina potrebbe riorganizzarsi o il farmaco potrebbe cambiare il suo orientamento in modo tale da provocare effetti collaterali".
Modellare i siti frustrati, e talvolta modificarli per vedere cosa accadrebbe, consente ai ricercatori di vedere come la specificità del farmaco è correlata alle tasche di legame. Analisi della frustrazione, scrissero, fornisce "un percorso per lo screening di composti più specifici per la scoperta di farmaci".
"Questo concetto di frustrazione era presente all'inizio del nostro lavoro sul ripiegamento delle proteine, " ha detto Wolynes. "Quando l'abbiamo applicato a vere molecole proteiche, abbiamo trovato alcuni esempi in cui il meccanismo di piegatura ha violato ciò che avremmo previsto da un imbuto perfetto. Poi abbiamo scoperto che queste deviazioni dall'immagine dell'imbuto si sono verificate dove si trovava la proteina, infatti, alquanto frustrato.
"Era come l'eccezione che conferma la regola, " ha detto. "Qualcosa che è sempre vero potrebbe essere banale. Ma se non è vero l'1% delle volte, è un problema da risolvere, e siamo stati in grado di farlo con AWSEM, il nostro software di previsione della struttura."
È possibile estendere il software per analizzare la frustrazione a livello atomico, come descritto dal gruppo in un altro recente documento. Ma il costo computazionale del tracciamento di ogni atomo in una proteina è così alto che i ricercatori avevano bisogno di un modo per campionare i movimenti di regioni specifiche in cui la frustrazione potrebbe confondere il percorso di piegatura.
"Mingchen si rese conto che esisteva un algoritmo efficiente per campionare gli ambienti locali nei siti di legame ma mantenere la risoluzione atomistica, " disse Wolynes, che ha notato lui e Chen, ora nell'industria privata, stanno usando i modelli per studiare possibili terapie, compresi i farmaci correlati al COVID-19.