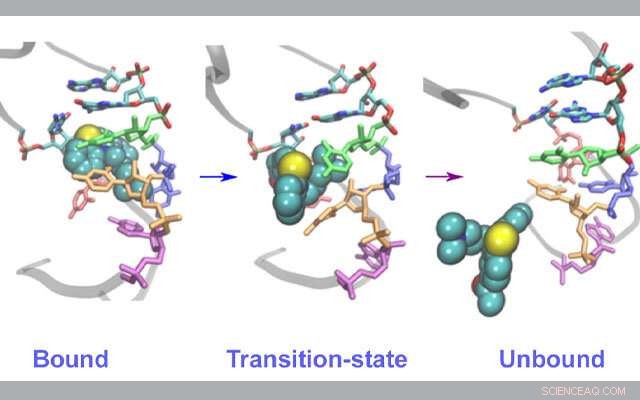
Cambiamenti strutturali nell'RNA durante il legame/svincolamento del farmaco. Credito:Lev Levinov, Università del New Hampshire
I ricercatori dell'Università del New Hampshire (UNH) hanno recentemente utilizzato Comet presso il San Diego Supercomputer Center presso l'UC San Diego e Stampede2 presso il Texas Advanced Computing Center per identificare nuovi percorsi di legame/disconnessione degli inibitori in un virus a base di RNA. I risultati potrebbero essere utili per capire come reagiscono questi inibitori e potenzialmente aiutare a sviluppare una nuova generazione di farmaci per colpire virus con alti tassi di mortalità, come l'HIV-1, Zika, Ebola, e SARS-CoV2, il virus che causa il COVID-19.
"Quando abbiamo iniziato questa ricerca, non ci saremmo mai aspettati di trovarci nel bel mezzo di una pandemia causata da un virus a RNA, " disse Harish Vashisth, professore associato di ingegneria chimica presso UNH. "Quando emergono questi tipi di virus, si spera che i nostri risultati offrano una migliore comprensione di come gli RNA virali interagiscono con gli inibitori e vengano utilizzati per progettare trattamenti migliori".
Simile a come gli umani codificano il loro genoma usando il DNA, molti virus hanno un corredo genetico di molecole di RNA. Questi genomi basati su RNA contengono potenziali siti in cui gli inibitori possono attaccare e disattivare il virus. Parte della sfida nello sviluppo di farmaci è rappresentata dalle variazioni o mutazioni nel genoma virale che possono impedire l'adesione degli inibitori.
Nel loro studio, recentemente pubblicato in Journal of Physical Chemistry Letters , Vashisth e il suo team hanno creato simulazioni di dinamica molecolare utilizzando i supercomputer Comet e Stampede2 per esaminare specificamente un frammento di RNA del virus HIV-1 e la sua interazione con l'acetilpromazina, una piccola molecola nota per interferire con il processo di replicazione del virus.
Gli scienziati si sono concentrati sugli elementi strutturali del genoma dell'RNA dell'HIV-1 perché sono considerati un buon modello per studiare gli stessi processi in un'ampia gamma di virus a RNA. Queste simulazioni hanno permesso loro di scoprire i percorsi dell'inibitore che si separa dall'RNA virale in diversi eventi rari, spesso difficili da osservare sperimentalmente, che hanno mostrato inaspettatamente un movimento coordinato in molte parti della tasca di legame che sono i mattoni dell'RNA.
Grazie alle allocazioni della National Science Foundation (NSF) Extreme Science and Engineering Environment (XSEDE) su Comet e Stampede2, i ricercatori sono stati in grado di eseguire centinaia di simulazioni contemporaneamente per osservare quelli che vengono chiamati rari eventi di base-flip coinvolti nel processo di legame/svincolamento dell'inibitore che ha fornito i nuovi dettagli del meccanismo sottostante di questo processo.
"La nostra speranza è che questo aggiunga nuove possibilità a un campo tradizionalmente incentrato sulle strutture biomolecolari statiche e porti a nuovi farmaci, " Ha detto Vashisth.