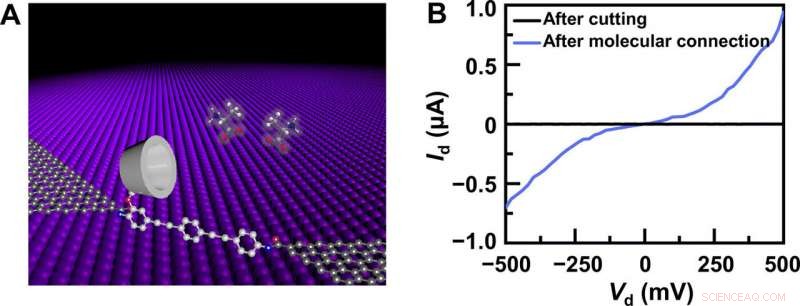
Struttura del dispositivo e caratterizzazione elettrica. (A) Rappresentazione schematica di un GMG-SMJ basato su PM-β-CD. La macchina molecolare dotata di un PM-β-CD è stata connessa in modo covalente con punti di contatto di grafene attraverso legami ammidici. (B) Curve I-V di GMG-SMJ dopo taglio al plasma di ossigeno e dopo ulteriore connessione molecolare. Credito: Progressi scientifici , doi:10.1126/sciadv.abe4365
La chimica analitica mira a discriminare in modo efficiente tra due amminoacidi. In un nuovo rapporto in Progressi scientifici , Zihao Liu e un gruppo di ricerca in Chimica, Fisica, e Scienza dei Materiali in Cina e Giappone, ha utilizzato un metodo elettrico a singola molecola basato su nanocircuiti molecolari per formare giunzioni stabili a singola molecola grafene-molecola-grafene. Il team ha sviluppato queste giunzioni molecolari legando in modo covalente una macchina molecolare. Quindi hanno usato il pH per variare il tipo e la carica degli amminoacidi per trovare fluttuazioni di corrente multimodali distinte originate da diverse interazioni ospite ospite relative a calcoli teorici. I dati di conduttanza hanno prodotto tempi di livellamento caratteristici e velocità di spostamento per ciascun amminoacido per consentire misurazioni in situ accurate e in tempo reale. Gli scienziati hanno testato quattro amminoacidi e i loro enantiomeri (molecole chirali che sono immagini speculari l'una dell'altra) per mostrare la capacità di distinguerli in pochi microsecondi per fornire un metodo semplice e preciso per l'identificazione degli amminoacidi e della proteina a molecola singola sequenziamento.
Elementi costitutivi delle proteine
Gli amminoacidi formano i mattoni delle proteine, molecole con funzioni fisiologiche speciali ed elementi strutturali chiave nei prodotti farmaceutici con applicazioni nelle scienze della vita. I ricercatori possono riconoscere gli enantiomeri degli amminoacidi per fornire importanti informazioni relative al riconoscimento chirale e alle funzioni fisiologiche nei sistemi biologici. Rilevamento della struttura, la purezza enantiomerica e il comportamento dinamico degli amminoacidi possono favorire la produzione di nuove tecniche per il sequenziamento delle proteine e le indagini farmaceutiche. Il rilevamento e l'identificazione di amminoacidi con diverse strutture e chiralità sono di fondamentale importanza in proteomica, farmaceutici e nanobiotecnologie. Però, le macchine molecolari esistenti funzionano solo confrontando i parametri termodinamici dell'insieme, che è insufficiente. È quindi importante sviluppare una tecnica molecolare generale per rivoluzionare i metodi esistenti e riconoscere le molecole bersaglio con elevata precisione. In questo lavoro, Liu et al. ha dimostrato una tecnica a singola molecola per identificare direttamente diversi amminoacidi proteinogenici e i loro enantiomeri tramite misurazioni dinamiche accurate delle interazioni ospite-ospite basate su una tecnica a singola molecola nota come giunzioni a singola molecola grafene-molecola-grafene abbreviate come GMG-SMJ a covalentemente incorporare singoli sistemi molecolari che si comportano come canale conduttivo in un nanocircuito elettrico.
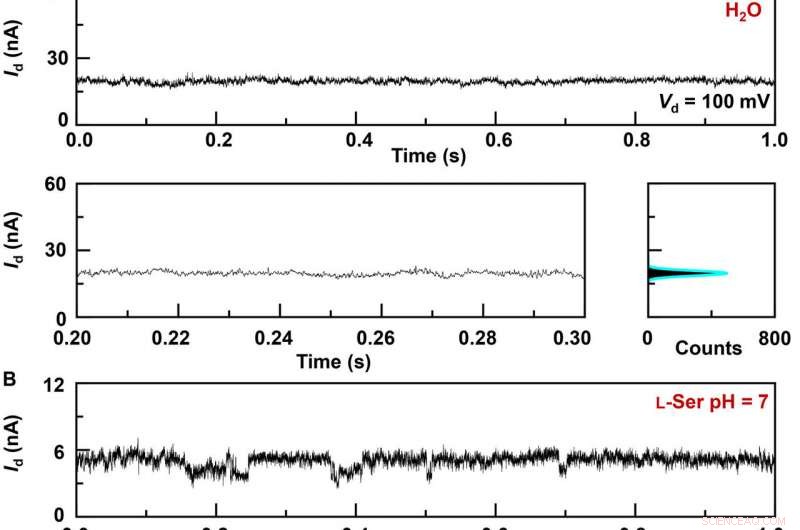
Registrazioni correnti in tempo reale delle dinamiche di interazione ospite-ospite in GMG-SMJ. Curve I-t di diversi GMG-SMJ (in alto), le parti ingrandite (in basso a sinistra), e i loro istogrammi corrispondenti (in basso a destra):(A) Un dispositivo funzionante in acqua pura, (B) lo stesso dispositivo in una soluzione acquosa di L-Ser da 100 nM, e (C) un altro dispositivo in una soluzione acquosa di L-Ala 100 nM. I riquadri negli istogrammi sono le parti ingrandite dei piccoli picchi. pH =7, T =298 K, e la tensione di polarizzazione =0,1 V. Credito:Science Advances, doi:10.1126/sciadv.abe4365
Il nuovo approccio ha fornito una solida piattaforma per formare un'elettronica a molecola singola per creare dispositivi optoelettronici molecolari. Per sviluppare i costrutti, Liu et al. ha inserito in modo covalente una macchina molecolare tra una coppia di punti di contatto in grafene nano-gapped per il rilevamento di amminoacidi e il riconoscimento della chiralità. I risultati apriranno nuove strade per sviluppare la nanotecnologia del sequenziamento accurato delle proteine a singola molecola per applicazioni pratiche.
Sviluppo del dispositivo, caratterizzazione elettrica e misurazioni in tempo reale
Il team di ricerca ha sintetizzato il grafene a strato singolo utilizzando la deposizione chimica da vapore su fogli di rame e lo ha trasferito su biossido di silicio/wafer di silicio e ha modellato gli elettrodi metallici utilizzando la fotolitografia. Per sviluppare gli elettrodi di contatto del punto di grafene nano-gapped, hanno usato un metodo litografico tratteggiato. Hanno quindi costruito GMG-SMJ (giunzioni a singola molecola grafene-molecola-grafene) collegando in modo covalente una singola macchina molecolare con elettrodi di grafene tramite legami ammidici. Il team ha misurato le curve corrente-tensione dei dispositivi in una varietà di fasi per identificare la formazione di GMG-SMJ. Il trasporto di carica attraverso la giunzione è il risultato di connessioni di singole molecole. Hanno quindi condotto caratterizzazioni elettriche dipendenti dal tempo per monitorare la conduttanza delle singole giunzioni molecolari (SMJ) permetilate-β-ciclodestrine (PM-β-CD) in tempo reale. Hanno prima misurato i costrutti in acqua e poi in diverse soluzioni di diversi amminoacidi. Le traiettorie rappresentative dell'ora corrente hanno rivelato distribuzioni di picchi multipli per diversi amminoacidi in soluzione come L-serina e L-alanina. I molteplici stati distinti di ciascun amminoacido hanno avuto origine solo dal processo di associazione/dissociazione tra il centro funzionale PM-β-CD e gli amminoacidi circostanti.
Analisi computazionali di L-Ala@PM-β-CD SMJ. (A) Diagramma schematico degli orbitali molecolari di frontiera calcolati della macchina molecolare. (B a D) Tipiche configurazioni molecolari durante l'associazione con amminoacidi con carica diversa:(B) catione, (C) zwitterione, e (D) anione. (E) Spettri di trasmissione corrispondenti a una tensione di polarizzazione zero. Il rosso, giallo, e le curve verdi mostrano la trasmissione quando l'anione, zwitterion, e le forme cationiche erano nella cavità, rispettivamente. La curva blu mostra la cavità vuota. Gli spettri completi sono forniti in fig. S4. (F a H) Rappresentazione schematica degli amminoacidi nel catione, zwitterion, e forme anioniche. Credito:progressi scientifici, doi:10.1126/sciadv.abe4365 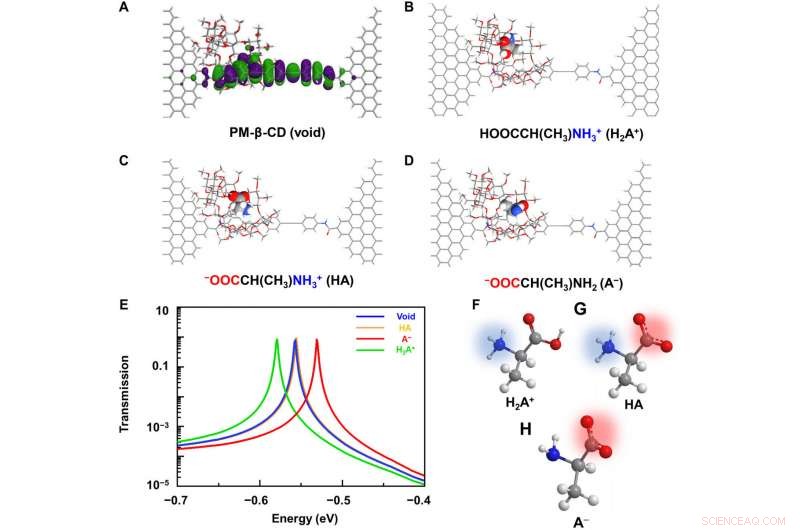
Per comprendere meglio la correlazione tra le interazioni ospite-ospite nelle giunzioni a singola molecola PM-β-CD basate su L-alanina, Liu et al. calcolato gli spettri di trasmissione dell'host PM-β-CD con ospiti L-alanina con carica diversa. Per realizzare questo, they used a nonequilibrium Green's function technique based on density functional theory as implied in the Atomistix Toolkit package. They noted the conductance contribution from the perturbed highest occupied molecular orbital (p-HOMO) to be dominant at low bias voltages as reflected with transmission spectra. The transmission spectra of the configurations were significantly different near the Fermi level of electrodes to afford different conductance stages. The researchers then analyzed the transitions between each level during amino acid recognition. They used L-alanine as an example to observe reversible transitions from the dissociation to form cations, zwitterions, and anion forms. The dissociation level dominated the device conductance, and the team collected the information using a four-state model, which described the transitions to understand the capacity to detect different structures of amino acids when interacting with PM-β-CD. The amino acids had at least two association processes for the carboxylic group and the other amino group. The greater the number of states recorded, the more accurate the recognition of results were.
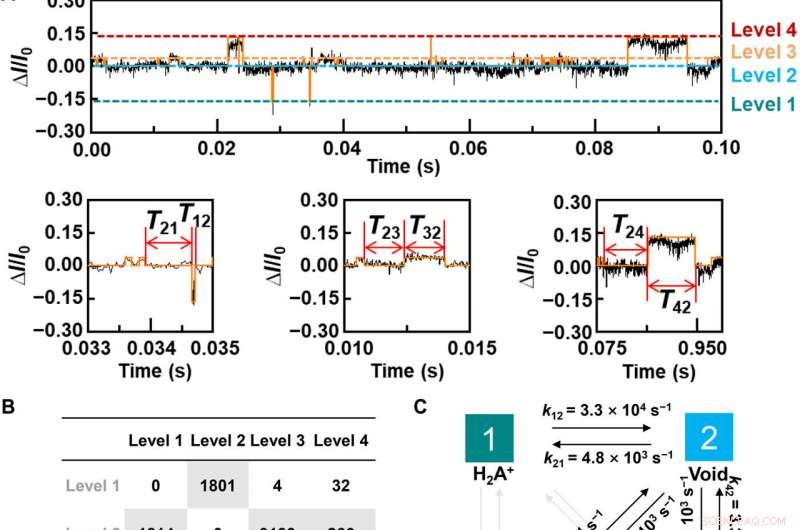
Dynamic analysis for L-Alanine detection. (A) Plot of ΔI/I0 as a function of time during the host-guest interaction. The red curve is the idealized fit by using a QuB software. pH =7, T =298 K, and the bias voltage =0.1 V. (B) Transition statistics between each state. The horizontal columns mean the initial state. (C) Kinetic model for the L-Ala recognition process. (D to I), Plots of time intervals of (D) level 1 to level 2, (E) level 2 to level 1, (F) level 2 to level 3, (G) level 3 to level 2, (H) level 2 to level 4, and (I) level 4 to level 2 at 298 K. Credit:Science Advances, doi:10.1126/sciadv.abe4365
Enantiomer recognition and outlook
Due to the similarity between enantiomers, it was more challenging to differentiate the structures compared to species identification. The associated energy differences were small and close to computational errors. To overcome these complexities, the team established a distinct "fingerprint database" for each amino acid to compare the conductance and kinetic data of different enantiomers. The team then achieved the current change and relaxation time data and compared them with the fingerprint database. Due to this universality, the method can also recognize chiral functional drug molecules to establish a broad approach for biomolecular detection at the single molecule-level.
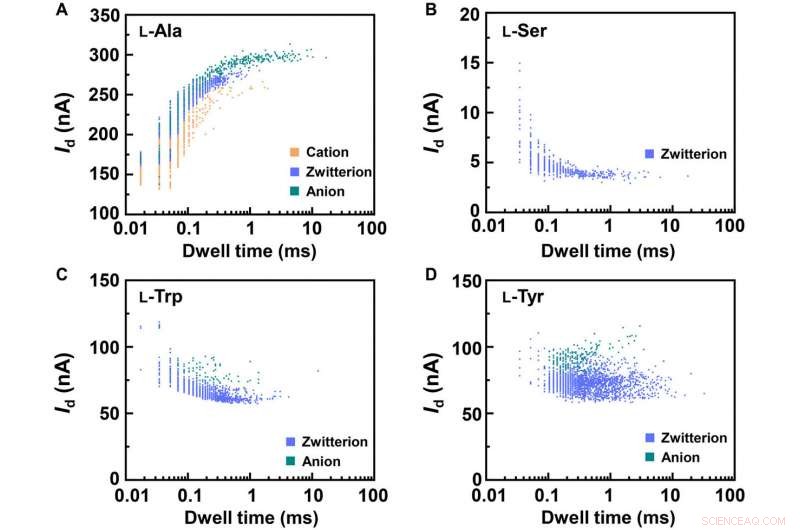
Statistical analyses of the translocation events for different amino acids. Translocation events (current as a function of the dwell time) were analyzed for (A) L-Ala, (B) L-Ser, (C) L-Trp, and (D) L-Tyr, rispettivamente. The two-dimensional (2D) contour plots are composed of all the events in 10-s recording at pH =7, showing the capability of detecting the different structures of amino acids when interacting with PM-β-CD. Credito:progressi scientifici, doi:10.1126/sciadv.abe4365
In questo modo, Zihao Liu and colleagues presented a practical single-molecule approach for real-time electrical recognition of amino acids with different structures and chirality within microseconds. The host-guest dynamic processes could be observed for diverse amino acids at the level of the anion, zwitterion and cation shuttling. Using the permethylated-β-cyclodextrin (PM-β-CD) complex, the team recognized differently charged states of amino acids based on current fluctuation range and thermodynamic/kinetic parameters. The technique can revolutionize existing methods to accurately sequence single-molecule gene/protein sequencing toward universal applications. The method can also offer a universal tool to recognize many important molecules in environmental or biological systems to understand the basis of life at the molecular level.
© 2021 Science X Network