Credito:Università di Kanazawa
I ricercatori dell'Università di Kanazawa riferiscono nel Journal of Physical Chemistry Letters come simulare immagini 3D di microscopia a forza atomica di sistemi fuori equilibrio che coinvolgono biomolecole. L'approccio fa uso di una celebre equazione della termodinamica applicabile a situazioni di non equilibrio.
La microscopia a forza atomica tridimensionale (3D-AFM) è una tecnica utilizzata per sondare la distribuzione di molecole di solvente alle interfacce solido-liquido. Inizialmente applicato per studiare situazioni in cui il solvente è l'acqua, il metodo viene ora utilizzato anche per altre molecole. Uno sviluppo recente consiste nell'utilizzare 3D-AFM per risolvere l'organizzazione di biopolimeri come cromosomi o proteine all'interno delle cellule. A causa della complessità di tali sistemi, tuttavia, sono necessarie simulazioni del processo di imaging 3D-AFM per assisterne l'interpretazione. I metodi di simulazione sviluppati finora hanno presupposto che il sistema sondato sia in equilibrio durante il ciclo di scansione AFM. Ciò limita la loro validità alle situazioni in cui le molecole di solvente si muovono molto più velocemente della sonda di scansione. Ora, Takeshi Fukuma dell'Università di Kanazawa e colleghi hanno sviluppato un approccio di simulazione 3D-AFM che funziona per sistemi non in equilibrio, applicabile a situazioni in cui il movimento molecolare avviene su scale temporali paragonabili o superiori a quelle del ciclo di indagine AFM.
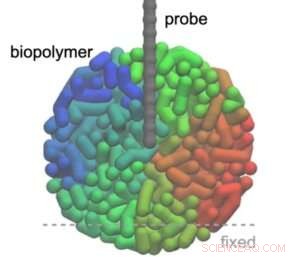
Il principio di base dell'AFM è quello di realizzare una punta molto piccola, attaccata a un cantilever, per scansionare la superficie di un campione. La risposta della punta alle differenze di altezza nella superficie scansionata fornisce informazioni strutturali del campione. In 3D-AFM, la punta viene fatta penetrare nel campione e la forza subita dalla punta è il risultato delle interazioni con le molecole vicine (parti di). Per una data posizione orizzontale (xy) della punta, la dipendenza della forza F dalla posizione verticale (z) della punta mentre penetra nel campione viene catturata in una curva forza-distanza (F contro z). Combinando tutte le curve forza-distanza ottenute durante la scansione xy si ottiene l'immagine 3D-AFM.
Fukuma e colleghi hanno considerato la situazione in cui una punta AFM sonda un biopolimero globulare e hanno modellato sia la punta che la molecola come perline collegate da molle (2.000 perline per la molecola, 50 perline per la punta). Hanno calcolato le curve forza-distanza utilizzando la cosiddetta uguaglianza di Jarzynski, un'equazione che mette in relazione la differenza di energia libera tra due stati di un sistema al lavoro (proporzionale alla forza) necessario per passare da uno stato all'altro. È importante sottolineare che l'uguaglianza vale per le situazioni di non equilibrio.
I ricercatori sono stati in grado di dimostrare che le simulazioni riproducevano la struttura interna del biopolimero, con alcune caratteristiche delle fibre chiaramente osservabili. Hanno anche esaminato il modo in cui la velocità di scansione influisce sui risultati della simulazione e hanno scoperto che esiste un intervallo di velocità ottimale per la scansione verticale (z). Infine, Fukuma e colleghi hanno simulato immagini 3D-AFM di fibre del citoscheletro per le quali esistono immagini 3D-AFM ottenute sperimentalmente e hanno scoperto che le simulazioni concordano bene con l'esperimento. Gli scienziati hanno quindi concluso che il loro metodo "è applicabile a varie fibre in cellule come il DNA e così via modificando parametri come la rigidità, fornendo un'importante base teorica per tali misurazioni sperimentali". + Esplora ulteriormente